+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3678 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the yeast R2TP complex | |||||||||
 マップデータ マップデータ | Structure of the yeast R2TP complex | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報TTT Hsp90 cochaperone complex / R2TP complex / Swr1 complex / Ino80 complex / box C/D snoRNP assembly / 3'-5' DNA helicase activity / NuA4 histone acetyltransferase complex / DNA helicase activity / rRNA processing / 5'-3' DNA helicase activity ...TTT Hsp90 cochaperone complex / R2TP complex / Swr1 complex / Ino80 complex / box C/D snoRNP assembly / 3'-5' DNA helicase activity / NuA4 histone acetyltransferase complex / DNA helicase activity / rRNA processing / 5'-3' DNA helicase activity / DNA helicase / forked DNA-dependent helicase activity / single-stranded 3'-5' DNA helicase activity / four-way junction helicase activity / double-stranded DNA helicase activity / protein stabilization / chromatin remodeling / DNA repair / regulation of DNA-templated transcription / regulation of transcription by RNA polymerase II / chromatin / ATP hydrolysis activity / ATP binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 8.37 Å | |||||||||
 データ登録者 データ登録者 | Rivera-Calzada A / Pal M / Munoz-Hernandez H / Luque-Ortega JR / Gil-Carton D / Degliesposti G / Skehel JM / Prodromou C / Pearl LH / Llorca O | |||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2017 ジャーナル: Structure / 年: 2017タイトル: The Structure of the R2TP Complex Defines a Platform for Recruiting Diverse Client Proteins to the HSP90 Molecular Chaperone System. 著者: Angel Rivera-Calzada / Mohinder Pal / Hugo Muñoz-Hernández / Juan R Luque-Ortega / David Gil-Carton / Gianluca Degliesposti / J Mark Skehel / Chrisostomos Prodromou / Laurence H Pearl / Oscar Llorca /   要旨: The R2TP complex, comprising the Rvb1p-Rvb2p AAA-ATPases, Tah1p, and Pih1p in yeast, is a specialized Hsp90 co-chaperone required for the assembly and maturation of multi-subunit complexes. These ...The R2TP complex, comprising the Rvb1p-Rvb2p AAA-ATPases, Tah1p, and Pih1p in yeast, is a specialized Hsp90 co-chaperone required for the assembly and maturation of multi-subunit complexes. These include the small nucleolar ribonucleoproteins, RNA polymerase II, and complexes containing phosphatidylinositol-3-kinase-like kinases. The structure and stoichiometry of yeast R2TP and how it couples to Hsp90 are currently unknown. Here, we determine the 3D organization of yeast R2TP using sedimentation velocity analysis and cryo-electron microscopy. The 359-kDa complex comprises one Rvb1p/Rvb2p hetero-hexamer with domains II (DIIs) forming an open basket that accommodates a single copy of Tah1p-Pih1p. Tah1p-Pih1p binding to multiple DII domains regulates Rvb1p/Rvb2p ATPase activity. Using domain dissection and cross-linking mass spectrometry, we identified a unique region of Pih1p that is essential for interaction with Rvb1p/Rvb2p. These data provide a structural basis for understanding how R2TP couples an Hsp90 dimer to a diverse set of client proteins and complexes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3678.map.gz emd_3678.map.gz | 3.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3678-v30.xml emd-3678-v30.xml emd-3678.xml emd-3678.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3678.png emd_3678.png | 305.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3678 http://ftp.pdbj.org/pub/emdb/structures/EMD-3678 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3678 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3678 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3678.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3678.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of the yeast R2TP complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.34 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
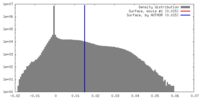
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Yeast R2TP complex
| 全体 | 名称: Yeast R2TP complex |
|---|---|
| 要素 |
|
-超分子 #1: Yeast R2TP complex
| 超分子 | 名称: Yeast R2TP complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 359 KDa |
-分子 #1: Rvb1p
| 分子 | 名称: Rvb1p / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MMVAISEVKE NPGVNSSNSG AVTRTAAHTH IKGLGLDESG VAKRVEGGFV GQIEAREACG V IVDLIKAK KMSGRAILLA GGPSTGKTAL ALAISQELGP KVPFCPLVGS ELYSVEVKKT ET LMENFRR AIGLRIKETK EVYEGEVTEL ...文字列: MGSSHHHHHH SSGLVPRGSH MMVAISEVKE NPGVNSSNSG AVTRTAAHTH IKGLGLDESG VAKRVEGGFV GQIEAREACG V IVDLIKAK KMSGRAILLA GGPSTGKTAL ALAISQELGP KVPFCPLVGS ELYSVEVKKT ET LMENFRR AIGLRIKETK EVYEGEVTEL TPEDAENPLG GYGKTISHVI VGLKSAKGTK TLR LDPTIY ESIQREKVSI GDVIYIEANT GAVKRVGRSD AYATEFDLET EEYVPLPKGE VHKK KEIVQ DVTLHDLDVA NARPQGGQDV ISMMGQLLKP KKTEITEKLR QEVNKVVAKY IDQGV AELI PGVLFIDEVN MLDIEIFTYL NKALESNIAP VVVLASNRGM TTVRGTEDVI SPHGVP PDL IDRLLIVRTL PYDKDEIRTI IERRATVERL QVESSALDLL ATMGTETSLR YALQLLA PC GILAQTSNRK EIVVNDVNEA KLLFLDAKRS TKILETSANY L |
-分子 #2: Rvb2p
| 分子 | 名称: Rvb2p / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO / EC番号: DNA helicase |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEQKLISEED LLRSEEQKLI SEEDLLRSEE QKLISEEDLL RSE SRGSHH HHHHLEVLFQ GPAS MSIQT SDPNETSDLK SLSLIAAHSH ITGLGLDENL QPRPTSEGMV GQLQARRAAG VILKM VQNG TIAGRAVLVA GPPSTGKTAL AMGVSQSLGK DVPFTAIAGS ...文字列: MEQKLISEED LLRSEEQKLI SEEDLLRSEE QKLISEEDLL RSE SRGSHH HHHHLEVLFQ GPAS MSIQT SDPNETSDLK SLSLIAAHSH ITGLGLDENL QPRPTSEGMV GQLQARRAAG VILKM VQNG TIAGRAVLVA GPPSTGKTAL AMGVSQSLGK DVPFTAIAGS EIFSLELSKT EALTQA FRK SIGIKIKEET ELIEGEVVEI QIDRSITGGH KQGKLTIKTT DMETIYELGN KMIDGLT KE KVLAGDVISI DKASGKITKL GRSFARSRDY DAMGADTRFV QCPEGELQKR KTVVHTVS L HEIDVINSRT QGFLALFTGD TGEIRSEVRD QINTKVAEWK EEGKAEIVPG VLFIDEVHM LDIECFSFIN RALEDEFAPI VMMATNRGVS KTRGTNYKSP HGLPLDLLDR SIIITTKSYN EQEIKTILS IRAQEEEVEL SSDALDLLTK TGVETSLRYS SNLISVAQQI AMKRKNNTVE V EDVKRAYL LFLDSARSVK YVQENESQYI DDQGNVQISI AKSADPDAMD TTE |
-分子 #3: Pih1p
| 分子 | 名称: Pih1p / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSHHHHHHMA DFLLRPIKQR HRNEDKYVSV DAADGSVSKI EPIADFVIKT KLLSANGPEK LQDGRKVFIN VCHSPLVPKP EVDFNARIVF PLIIQNEWEI PIITSCYRMD HDKKGQECYV WDCCINSDCS RWICDDIQLR EILVEWCLES CEIRDSVVLC RDRIAFPKMK ...文字列: MSHHHHHHMA DFLLRPIKQR HRNEDKYVSV DAADGSVSKI EPIADFVIKT KLLSANGPEK LQDGRKVFIN VCHSPLVPKP EVDFNARIVF PLIIQNEWEI PIITSCYRMD HDKKGQECYV WDCCINSDCS RWICDDIQLR EILVEWCLES CEIRDSVVLC RDRIAFPKMK KKGAELPALE VLNDELHQDY KAKMHKIIEE EAGDPMSILR GRNDDGDDNN DPDDGTLPPL FPIENKISGA KIEEIDKNEI AHRNLKQAPA PAPAPHEQQE DVPEYEVKMK RFKGAAYKLR ILIENKAPNS KPDRFSPSYN FAENILYING KLSIPLPRDI VVNAADIKIF HIRKERTLYI YI |
-分子 #4: Tah1p
| 分子 | 名称: Tah1p / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH WSHPQFEKGG GSGGGSGGSS AWSHPQFEKL EVLFQGPHMM SQFEKQKEQG NSLFKQGLY REAVHCYDQL ITAQPQNPVG YSNKAMALIK LGEYTQAIQM CQQGLRYTST AEHVAIRSKL QYRLELAQGA VGSVQIPVVE VDELPEGYDR S |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.45 mg/mL | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.8 構成要素:
| ||||||||||||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE | ||||||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - 画像ごとのフレーム数: 2-19 / 平均露光時間: 12.0 sec. / 平均電子線量: 48.0 e/Å2 詳細: Data was collected at two different tilts: 0 and 35 degrees. |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 39000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)