+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3633 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Localised reconstruction of alpha v beta 6 bound to Foot and Mouth Disease Virus O PanAsia - Pose A prime | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Foot and Mouth Disease Virus / FMDV / virus / OpanAsia / virus-receptor / complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Langerhans cell differentiation / integrin alphav-beta8 complex / integrin alphav-beta6 complex / transforming growth factor beta production / negative regulation of entry of bacterium into host cell / integrin alphav-beta5 complex / opsonin binding / enamel mineralization / symbiont-mediated perturbation of host chromatin organization / integrin alphav-beta1 complex ...Langerhans cell differentiation / integrin alphav-beta8 complex / integrin alphav-beta6 complex / transforming growth factor beta production / negative regulation of entry of bacterium into host cell / integrin alphav-beta5 complex / opsonin binding / enamel mineralization / symbiont-mediated perturbation of host chromatin organization / integrin alphav-beta1 complex / bronchiole development / Cross-presentation of particulate exogenous antigens (phagosomes) / extracellular matrix protein binding / Laminin interactions / integrin alphav-beta3 complex / negative regulation of lipoprotein metabolic process / alphav-beta3 integrin-PKCalpha complex / entry into host cell by a symbiont-containing vacuole / phospholipid homeostasis / alphav-beta3 integrin-HMGB1 complex / negative regulation of lipid transport / hard palate development / regulation of phagocytosis / Elastic fibre formation / alphav-beta3 integrin-IGF-1-IGF1R complex / surfactant homeostasis / transforming growth factor beta binding / positive regulation of small GTPase mediated signal transduction / filopodium membrane / extracellular matrix binding / negative regulation of low-density lipoprotein particle clearance / apolipoprotein A-I-mediated signaling pathway / apoptotic cell clearance / wound healing, spreading of epidermal cells / integrin complex / heterotypic cell-cell adhesion / Molecules associated with elastic fibres / negative chemotaxis / Mechanical load activates signaling by PIEZO1 and integrins in osteocytes / lung alveolus development / Syndecan interactions / cell adhesion mediated by integrin / skin development / positive regulation of osteoblast proliferation / microvillus membrane / cell-substrate adhesion / endodermal cell differentiation / PECAM1 interactions / TGF-beta receptor signaling activates SMADs / positive regulation of intracellular signal transduction / fibronectin binding / lamellipodium membrane / negative regulation of macrophage derived foam cell differentiation / negative regulation of lipid storage / ECM proteoglycans / Integrin cell surface interactions / vasculogenesis / voltage-gated calcium channel activity / specific granule membrane / coreceptor activity / phagocytic vesicle / ERK1 and ERK2 cascade / extrinsic apoptotic signaling pathway in absence of ligand / substrate adhesion-dependent cell spreading / positive regulation of cell adhesion / transforming growth factor beta receptor signaling pathway / protein kinase C binding / ribonucleoside triphosphate phosphatase activity / molecular function activator activity / Turbulent (oscillatory, disturbed) flow shear stress activates signaling by PIEZO1 and integrins in endothelial cells / cell-matrix adhesion / Signal transduction by L1 / integrin-mediated signaling pathway / negative regulation of extrinsic apoptotic signaling pathway / cellular response to ionizing radiation / wound healing / T=pseudo3 icosahedral viral capsid / cell-cell adhesion / bone development / calcium ion transmembrane transport / host cell cytoplasmic vesicle membrane / VEGFA-VEGFR2 Pathway / response to virus / ruffle membrane / integrin binding / cell morphogenesis / viral capsid / cell migration / host cell / regulation of translation / channel activity / positive regulation of cytosolic calcium ion concentration / virus receptor activity / protease binding / angiogenesis / monoatomic ion transmembrane transport / clathrin-dependent endocytosis of virus by host cell / host cell cytoplasm / RNA helicase activity / receptor complex 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Foot-and-mouth disease virus (口蹄疫ウイルス) / Foot-and-mouth disease virus (口蹄疫ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
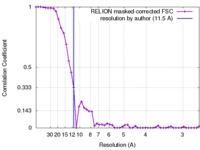
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 11.5 Å | ||||||||||||
 データ登録者 データ登録者 | Kotecha A / Stuart D | ||||||||||||
| 資金援助 |  英国, 3件 英国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Rules of engagement between αvβ6 integrin and foot-and-mouth disease virus. 著者: Abhay Kotecha / Quan Wang / Xianchi Dong / Serban L Ilca / Marina Ondiviela / Rao Zihe / Julian Seago / Bryan Charleston / Elizabeth E Fry / Nicola G A Abrescia / Timothy A Springer / Juha T ...著者: Abhay Kotecha / Quan Wang / Xianchi Dong / Serban L Ilca / Marina Ondiviela / Rao Zihe / Julian Seago / Bryan Charleston / Elizabeth E Fry / Nicola G A Abrescia / Timothy A Springer / Juha T Huiskonen / David I Stuart /     要旨: Foot-and-mouth disease virus (FMDV) mediates cell entry by attachment to an integrin receptor, generally αvβ6, via a conserved arginine-glycine-aspartic acid (RGD) motif in the exposed, antigenic, ...Foot-and-mouth disease virus (FMDV) mediates cell entry by attachment to an integrin receptor, generally αvβ6, via a conserved arginine-glycine-aspartic acid (RGD) motif in the exposed, antigenic, GH loop of capsid protein VP1. Infection can also occur in tissue culture adapted virus in the absence of integrin via acquired basic mutations interacting with heparin sulphate (HS); this virus is attenuated in natural infections. HS interaction has been visualized at a conserved site in two serotypes suggesting a propensity for sulfated-sugar binding. Here we determined the interaction between αvβ6 and two tissue culture adapted FMDV strains by cryo-electron microscopy. In the preferred mode of engagement, the fully open form of the integrin, hitherto unseen at high resolution, attaches to an extended GH loop via interactions with the RGD motif plus downstream hydrophobic residues. In addition, an N-linked sugar of the integrin attaches to the previously identified HS binding site, suggesting a functional role. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3633.map.gz emd_3633.map.gz | 7.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3633-v30.xml emd-3633-v30.xml emd-3633.xml emd-3633.xml | 24.6 KB 24.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3633_fsc.xml emd_3633_fsc.xml | 4.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3633.png emd_3633.png | 77.1 KB | ||
| Filedesc metadata |  emd-3633.cif.gz emd-3633.cif.gz | 7.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3633 http://ftp.pdbj.org/pub/emdb/structures/EMD-3633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3633 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3633 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3633_validation.pdf.gz emd_3633_validation.pdf.gz | 462.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3633_full_validation.pdf.gz emd_3633_full_validation.pdf.gz | 462.4 KB | 表示 | |
| XML形式データ |  emd_3633_validation.xml.gz emd_3633_validation.xml.gz | 7.9 KB | 表示 | |
| CIF形式データ |  emd_3633_validation.cif.gz emd_3633_validation.cif.gz | 9.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3633 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3633 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3633 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  5nerMC  3630C  3631C  3632C  3634C  3635C  5ne4C  5nedC  5nejC  5nemC  5netC  5neuC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3633.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3633.map.gz / 形式: CCP4 / 大きさ: 8 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Foot and mouth virus and Integrin
+超分子 #1: Foot and mouth virus and Integrin
+超分子 #2: Foot-and-mouth disease virus
+超分子 #3: Integrin
+分子 #1: O PanAsia VP1
+分子 #2: O PanAsia VP2
+分子 #3: O PanAsia VP3
+分子 #4: O PanAsia VP4
+分子 #5: Integrin alpha-V
+分子 #6: Integrin beta-6
+分子 #12: alpha-D-mannopyranose
+分子 #13: 2-acetamido-2-deoxy-beta-D-glucopyranose
+分子 #14: CALCIUM ION
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 構成要素 - 濃度: 50.0 mM / 構成要素 - 名称: HEPES |
| グリッド | モデル: C-flat-2/1 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 支持フィルム - Film thickness: 5 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 294 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 70.0 K / 最高: 70.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3838 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 2-20 / 撮影したグリッド数: 1 / 実像数: 360 / 平均露光時間: 5.0 sec. / 平均電子線量: 18.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 倍率(補正後): 37037 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 160000 |
| 試料ステージ | 試料ホルダーモデル: GATAN 910 MULTI-SPECIMEN SINGLE TILT CRYO TRANSFER HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 120 当てはまり具合の基準: Cross-correlation coefficient |
|---|---|
| 得られたモデル |  PDB-5ner: |
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















 Cricetinae gen. sp. (ネズミ)
Cricetinae gen. sp. (ネズミ)