[日本語] English
 万見
万見- EMDB-36085: Cryo-EM structure of nucleosome containing Arabidopsis thaliana H2A.W -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of nucleosome containing Arabidopsis thaliana H2A.W | ||||||||||||||||||||||||||||||||||||||||||
 マップデータ マップデータ | Cryo-EM map of nucleosome containing Arabidopsis thaliana H2A.W | ||||||||||||||||||||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||||||||||||||||||||
 キーワード キーワード | Chromatin / Epigenetics / Histon variant / chromatin remodeler / NUCLEAR PROTEIN | ||||||||||||||||||||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報heterochromatin organization / chloroplast thylakoid / chromocenter / thylakoid / response to water deprivation / plant-type vacuole / plasmodesma / chloroplast stroma / plastid / pericentric heterochromatin ...heterochromatin organization / chloroplast thylakoid / chromocenter / thylakoid / response to water deprivation / plant-type vacuole / plasmodesma / chloroplast stroma / plastid / pericentric heterochromatin / heterochromatin / chloroplast / structural constituent of chromatin / peroxisome / nucleosome / protein heterodimerization activity / chromatin binding / nucleolus / DNA binding / extracellular region / nucleus / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||||||||||||||||||||||||||||||||||||||
| 生物種 |  | ||||||||||||||||||||||||||||||||||||||||||
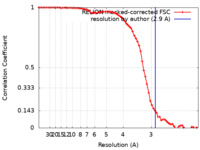
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.9 Å | ||||||||||||||||||||||||||||||||||||||||||
 データ登録者 データ登録者 | Osakabe A / Takizawa Y / Horikoshi N / Hatazawa S / Berger F / Kurumizaka H / Kakutani T | ||||||||||||||||||||||||||||||||||||||||||
| 資金援助 |  日本, 日本,  オーストリア, 13件 オーストリア, 13件
| ||||||||||||||||||||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Molecular and structural basis of the chromatin remodeling activity by Arabidopsis DDM1. 著者: Akihisa Osakabe / Yoshimasa Takizawa / Naoki Horikoshi / Suguru Hatazawa / Lumi Negishi / Shoko Sato / Frédéric Berger / Tetsuji Kakutani / Hitoshi Kurumizaka /   要旨: The histone H2A variant H2A.W occupies transposons and thus prevents access to them in Arabidopsis thaliana. H2A.W is deposited by the chromatin remodeler DDM1, which also promotes the accessibility ...The histone H2A variant H2A.W occupies transposons and thus prevents access to them in Arabidopsis thaliana. H2A.W is deposited by the chromatin remodeler DDM1, which also promotes the accessibility of chromatin writers to heterochromatin by an unknown mechanism. To shed light on this question, we solve the cryo-EM structures of nucleosomes containing H2A and H2A.W, and the DDM1-H2A.W nucleosome complex. These structures show that the DNA end flexibility of the H2A nucleosome is higher than that of the H2A.W nucleosome. In the DDM1-H2A.W nucleosome complex, DDM1 binds to the N-terminal tail of H4 and the nucleosomal DNA and increases the DNA end flexibility of H2A.W nucleosomes. Based on these biochemical and structural results, we propose that DDM1 counters the low accessibility caused by nucleosomes containing H2A.W to enable the maintenance of repressive epigenetic marks on transposons and prevent their activity. | ||||||||||||||||||||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36085.map.gz emd_36085.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36085-v30.xml emd-36085-v30.xml emd-36085.xml emd-36085.xml | 30.5 KB 30.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36085_fsc.xml emd_36085_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36085.png emd_36085.png | 130.4 KB | ||
| Filedesc metadata |  emd-36085.cif.gz emd-36085.cif.gz | 7.2 KB | ||
| その他 |  emd_36085_additional_1.map.gz emd_36085_additional_1.map.gz emd_36085_half_map_1.map.gz emd_36085_half_map_1.map.gz emd_36085_half_map_2.map.gz emd_36085_half_map_2.map.gz | 26.7 MB 23.4 MB 23.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36085 http://ftp.pdbj.org/pub/emdb/structures/EMD-36085 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36085 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36085 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_36085_validation.pdf.gz emd_36085_validation.pdf.gz | 772.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_36085_full_validation.pdf.gz emd_36085_full_validation.pdf.gz | 771.9 KB | 表示 | |
| XML形式データ |  emd_36085_validation.xml.gz emd_36085_validation.xml.gz | 12.7 KB | 表示 | |
| CIF形式データ |  emd_36085_validation.cif.gz emd_36085_validation.cif.gz | 17.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36085 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36085 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36085 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-36085 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36085.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36085.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of nucleosome containing Arabidopsis thaliana H2A.W | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Cryo-EM map of nucleosome containing Arabidopsis thaliana H2A.W...
| ファイル | emd_36085_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM map of nucleosome containing Arabidopsis thaliana H2A.W post-processed by DeepEMhancer | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_36085_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_36085_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nucleosome containing Arabidopsis thaliana H2A.W
| 全体 | 名称: Nucleosome containing Arabidopsis thaliana H2A.W |
|---|---|
| 要素 |
|
-超分子 #1: Nucleosome containing Arabidopsis thaliana H2A.W
| 超分子 | 名称: Nucleosome containing Arabidopsis thaliana H2A.W / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 200 KDa |
-超分子 #2: Nucleosome
| 超分子 | 名称: Nucleosome / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
-超分子 #3: DNA
| 超分子 | 名称: DNA / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #5-#6 |
|---|
-分子 #1: Histone H3.1
| 分子 | 名称: Histone H3.1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 15.583246 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMARTKQT ARKSTGGKAP RKQLATKAAR KSAPATGGVK KPHRFRPGTV ALREIRKYQK STELLIRKLP FQRLVREIAQ DFKTDLRFQ SSAVAALQEA AEAYLVGLFE DTNLCAIHAK RVTIMPKDIQ LARRIRGERA UniProtKB: Histone H3.1 |
-分子 #2: Histone H4
| 分子 | 名称: Histone H4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 11.718744 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMSGRGKG GKGLGKGGAK RHRKVLRDNI QGITKPAIRR LARRGGVKRI SGLIYEETRG VLKIFLENVI RDAVTYTEHA RRKTVTAMD VVYALKRQGR TLYGFGG UniProtKB: Histone H4 |
-分子 #3: HTA6
| 分子 | 名称: HTA6 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.280221 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMESTGKV KKAFGGRKPP GAPKTKSVSK SMKAGLQFPV GRITRFLKKG RYAQRLGGGA PVYMAAVLEY LAAEVLELAG NAARDNKKS RIIPRHLLLA IRNDEELGKL LSGVTIAHGG VLPNINSVLL PKKSATKPAE EKATKSPVKS PKKA UniProtKB: Probable histone H2A.7 |
-分子 #4: HTB9
| 分子 | 名称: HTB9 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 16.756738 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMAPRAEK KPAEKKPAAE KPVEEKSKAE KAPAEKKPKA GKKLPKEAGA GGDKKKKMKK KSVETYKIYI FKVLKQVHPD IGISSKAMG IMNSFINDIF EKLASESSKL ARYNKKPTIT SREIQTAVRL VLPGELAKHA VSEGTKAVTK FTSS UniProtKB: Histone H2B.6 |
-分子 #5: DNA (169-MER)
| 分子 | 名称: DNA (169-MER) / タイプ: dna / ID: 5 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 51.922059 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DG)(DA)(DC)(DC)(DC)(DT) (DA)(DT)(DC)(DG)(DC)(DG)(DA)(DG)(DC)(DC) (DA)(DG)(DG)(DC)(DC)(DT)(DG)(DA)(DG) (DA)(DA)(DT)(DC)(DC)(DG)(DG)(DT)(DG)(DC) (DC) (DG)(DA)(DG)(DG)(DC) ...文字列: (DA)(DT)(DC)(DG)(DG)(DA)(DC)(DC)(DC)(DT) (DA)(DT)(DC)(DG)(DC)(DG)(DA)(DG)(DC)(DC) (DA)(DG)(DG)(DC)(DC)(DT)(DG)(DA)(DG) (DA)(DA)(DT)(DC)(DC)(DG)(DG)(DT)(DG)(DC) (DC) (DG)(DA)(DG)(DG)(DC)(DC)(DG)(DC) (DT)(DC)(DA)(DA)(DT)(DT)(DG)(DG)(DT)(DC) (DG)(DT) (DA)(DG)(DA)(DC)(DA)(DG)(DC) (DT)(DC)(DT)(DA)(DG)(DC)(DA)(DC)(DC)(DG) (DC)(DT)(DT) (DA)(DA)(DA)(DC)(DG)(DC) (DA)(DC)(DG)(DT)(DA)(DC)(DG)(DC)(DG)(DC) (DT)(DG)(DT)(DC) (DC)(DC)(DC)(DC)(DG) (DC)(DG)(DT)(DT)(DT)(DT)(DA)(DA)(DC)(DC) (DG)(DC)(DC)(DA)(DA) (DG)(DG)(DG)(DG) (DA)(DT)(DT)(DA)(DC)(DT)(DC)(DC)(DC)(DT) (DA)(DG)(DT)(DC)(DT)(DC) (DC)(DA)(DG) (DG)(DC)(DA)(DC)(DG)(DT)(DG)(DT)(DC)(DA) (DG)(DA)(DT)(DA)(DT)(DA)(DT) (DA)(DC) (DA)(DT)(DC)(DC)(DG)(DA)(DT) |
-分子 #6: DNA (169-MER)
| 分子 | 名称: DNA (169-MER) / タイプ: dna / ID: 6 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 52.424352 KDa |
| 配列 | 文字列: (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT) ...文字列: (DA)(DT)(DC)(DG)(DG)(DA)(DT)(DG)(DT)(DA) (DT)(DA)(DT)(DA)(DT)(DC)(DT)(DG)(DA)(DC) (DA)(DC)(DG)(DT)(DG)(DC)(DC)(DT)(DG) (DG)(DA)(DG)(DA)(DC)(DT)(DA)(DG)(DG)(DG) (DA) (DG)(DT)(DA)(DA)(DT)(DC)(DC)(DC) (DC)(DT)(DT)(DG)(DG)(DC)(DG)(DG)(DT)(DT) (DA)(DA) (DA)(DA)(DC)(DG)(DC)(DG)(DG) (DG)(DG)(DG)(DA)(DC)(DA)(DG)(DC)(DG)(DC) (DG)(DT)(DA) (DC)(DG)(DT)(DG)(DC)(DG) (DT)(DT)(DT)(DA)(DA)(DG)(DC)(DG)(DG)(DT) (DG)(DC)(DT)(DA) (DG)(DA)(DG)(DC)(DT) (DG)(DT)(DC)(DT)(DA)(DC)(DG)(DA)(DC)(DC) (DA)(DA)(DT)(DT)(DG) (DA)(DG)(DC)(DG) (DG)(DC)(DC)(DT)(DC)(DG)(DG)(DC)(DA)(DC) (DC)(DG)(DG)(DA)(DT)(DT) (DC)(DT)(DC) (DA)(DG)(DG)(DC)(DC)(DT)(DG)(DG)(DC)(DT) (DC)(DG)(DC)(DG)(DA)(DT)(DA) (DG)(DG) (DG)(DT)(DC)(DC)(DG)(DA)(DT) |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 59.1 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 25.0 µm / 最小 デフォーカス(公称値): 10.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)