+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3571 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | dsRNA bacteriophage phi6 nucleocapsid | |||||||||
 マップデータ マップデータ | dsRNA bacteriophage phi6 nucleocapsid | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | icosahedral virus capsid shell / virus | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報T=2 icosahedral viral capsid / T=13 icosahedral viral capsid / viral procapsid / viral genome packaging / viral inner capsid / viral outer capsid / ribonucleoside triphosphate phosphatase activity / nucleoside-triphosphate phosphatase / viral capsid / viral nucleocapsid ...T=2 icosahedral viral capsid / T=13 icosahedral viral capsid / viral procapsid / viral genome packaging / viral inner capsid / viral outer capsid / ribonucleoside triphosphate phosphatase activity / nucleoside-triphosphate phosphatase / viral capsid / viral nucleocapsid / RNA binding / ATP binding / identical protein binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) | |||||||||
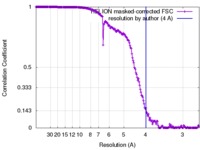
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.0 Å | |||||||||
 データ登録者 データ登録者 | Sun Z / El Omari K | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2017 ジャーナル: Nat Commun / 年: 2017タイトル: Double-stranded RNA virus outer shell assembly by bona fide domain-swapping. 著者: Zhaoyang Sun / Kamel El Omari / Xiaoyu Sun / Serban L Ilca / Abhay Kotecha / David I Stuart / Minna M Poranen / Juha T Huiskonen /   要旨: Correct outer protein shell assembly is a prerequisite for virion infectivity in many multi-shelled dsRNA viruses. In the prototypic dsRNA bacteriophage φ6, the assembly reaction is promoted by ...Correct outer protein shell assembly is a prerequisite for virion infectivity in many multi-shelled dsRNA viruses. In the prototypic dsRNA bacteriophage φ6, the assembly reaction is promoted by calcium ions but its biomechanics remain poorly understood. Here, we describe the near-atomic resolution structure of the φ6 double-shelled particle. The outer T=13 shell protein P8 consists of two alpha-helical domains joined by a linker, which allows the trimer to adopt either a closed or an open conformation. The trimers in an open conformation swap domains with each other. Our observations allow us to propose a mechanistic model for calcium concentration regulated outer shell assembly. Furthermore, the structure provides a prime exemplar of bona fide domain-swapping. This leads us to extend the theory of domain-swapping from the level of monomeric subunits and multimers to closed spherical shells, and to hypothesize a mechanism by which closed protein shells may arise in evolution. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3571.map.gz emd_3571.map.gz | 464.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3571-v30.xml emd-3571-v30.xml emd-3571.xml emd-3571.xml | 18.2 KB 18.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3571_fsc.xml emd_3571_fsc.xml | 17.8 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3571.png emd_3571.png | 216.1 KB | ||
| Filedesc metadata |  emd-3571.cif.gz emd-3571.cif.gz | 7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3571 http://ftp.pdbj.org/pub/emdb/structures/EMD-3571 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3571 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3571 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3571_validation.pdf.gz emd_3571_validation.pdf.gz | 299.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3571_full_validation.pdf.gz emd_3571_full_validation.pdf.gz | 298.3 KB | 表示 | |
| XML形式データ |  emd_3571_validation.xml.gz emd_3571_validation.xml.gz | 15.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3571 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3571 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3571 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3571 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3571.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3571.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | dsRNA bacteriophage phi6 nucleocapsid | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.35 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Pseudomonas phage phi6
| 全体 | 名称:  Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Pseudomonas phage phi6
| 超分子 | 名称: Pseudomonas phage phi6 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: The viral envelope was removed by Triton X-114 extraction NCBI-ID: 10879 / 生物種: Pseudomonas phage phi6 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Pseudomonas syringae (バクテリア) / 株: pv.phaseolicola HB10Y Pseudomonas syringae (バクテリア) / 株: pv.phaseolicola HB10Y |
| ウイルス殻 | Shell ID: 1 / 名称: Outer shell / 直径: 565.0 Å / T番号(三角分割数): 13 |
| ウイルス殻 | Shell ID: 2 / 名称: Inner shell / 直径: 500.0 Å / T番号(三角分割数): 1 |
-分子 #1: Major inner protein P1
| 分子 | 名称: Major inner protein P1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
| 分子量 | 理論値: 85.080711 KDa |
| 配列 | 文字列: MFNLKVKDLN GSARGLTQAF AIGELKNQLS VGALQLPLQF TRTFSASMTS ELLWEVGKGN IDPVMYARLF FQYAQAGGAL SVDELVNQF TEYHQSTACN PEIWRKLTAY ITGSSNRAIK ADAVGKVPPT AILEQLRTLA PSEHELFHHI TTDFVCHVLS P LGFILPDA ...文字列: MFNLKVKDLN GSARGLTQAF AIGELKNQLS VGALQLPLQF TRTFSASMTS ELLWEVGKGN IDPVMYARLF FQYAQAGGAL SVDELVNQF TEYHQSTACN PEIWRKLTAY ITGSSNRAIK ADAVGKVPPT AILEQLRTLA PSEHELFHHI TTDFVCHVLS P LGFILPDA AYVYRVGRTA TYPNFYALVD CVRASDLRRM LTALSSVDSK MLQATFKAKG ALAPALISQH LANAATTAFE RS RGNFDAN AVVSSVLTIL GRLWSPSTPK ELDPSARLRN TNGIDQLRSN LALFIAYQDM VKQRGRAEVI FSDEELSSTI IPW FIEAMS EVSPFKLRPI NETTSYIGQT SAIDHMGQPS HVVVYEDWQF AKEITAFTPV KLANNSNQRF LDVEPGISDR MSAT LAPIG NTFAVSAFVK NRTAVYEAVS QRGTVNSNGA EMTLGFPSVV ERDYALDRDP MVAIAALRTG IVDESLEARA SNDLK RSMF NYYAAVMHYA VAHNPEVVVS EHQGVAAEQG SLYLVWNVRT ELRIPVGYNA IEGGSIRTPE PLEAIAYNKP IQPSEV LQA KVLDLANHTT SIHIWPWHEA STEFAYEDAY SVTIRNKRYT AEVKEFELLG LGQRRERVRI LKPTVAHAII QMWYSWF VE DDRTLAAARR TSRDDAEKLA IDGRRMQNAV TLLRKIEMIG TTGIGASAVH LAQSRIVDQM AGRGLIDDSS DLHVGINR H RIRIWAGLAV LQMMGLLSRS EAEALTKVLG DSNALGMVVA TTDIDPSL UniProtKB: Major inner protein P1 |
-分子 #2: Packaging enzyme P4
| 分子 | 名称: Packaging enzyme P4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO / EC番号: nucleoside-triphosphate phosphatase |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
| 分子量 | 理論値: 35.198426 KDa |
| 配列 | 文字列: MPIVVTQAHI DRVGIAADLL DASPVSLQVL GRPTAINTVV IKTYIAAVME LASKQGGSLA GVDIRPSVLL KDTAIFTKPK AKSADVESD VDVLDTGIYS VPGLARKPVT HRWPSEGIYS GVTALMGATG SGKSITLNEK LRPDVLIRWG EVAEAYDELD T AVHISTLD ...文字列: MPIVVTQAHI DRVGIAADLL DASPVSLQVL GRPTAINTVV IKTYIAAVME LASKQGGSLA GVDIRPSVLL KDTAIFTKPK AKSADVESD VDVLDTGIYS VPGLARKPVT HRWPSEGIYS GVTALMGATG SGKSITLNEK LRPDVLIRWG EVAEAYDELD T AVHISTLD EMLIVCIGLG ALGFNVAVDS VRPLLFRLKG AASAGGIVAV FYSLLTDISN LFTQYDCSVV MVVNPMVDAE KI EYVFGQV MASTVGAILC ADGNVSRTMF RTNKGRIFNG AAPLAADTHM PSMDRPTSMK ALDHTSIASV APLERGSVDT DDR NSAPRR GANFSL UniProtKB: Packaging enzyme P4 |
-分子 #3: Major outer capsid protein
| 分子 | 名称: Major outer capsid protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 10 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas phage phi6 (ファージ) Pseudomonas phage phi6 (ファージ) |
| 分子量 | 理論値: 16.018418 KDa |
| 配列 | 文字列: MLLPVVARAA VPAIESAIAA TPGLVSRIAA AIGSKVSPSA ILAAVKSNPV VAGLTLAQIG STGYDAYQQL LENHPEVAEM LKDLSFKAD EIQPDFIGNL GQYREELELV EDAARFVGGM SNLIRLRQAL ELDIKYYGLK MQLNDMGYRS UniProtKB: Major outer capsid protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 |
| グリッド | モデル: C-flat / 材質: COPPER / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 15 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 80.0 K / 最高: 120.0 K |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum LS エネルギーフィルター - エネルギー下限: 0 eV エネルギーフィルター - エネルギー上限: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 3710 pixel / デジタル化 - サイズ - 縦: 3710 pixel / デジタル化 - 画像ごとのフレーム数: 1-22 / 実像数: 900 / 平均露光時間: 0.2 sec. / 平均電子線量: 0.73 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 3.0 µm / 倍率(補正後): 37037 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 0.3 µm / 倍率(公称値): 160000 |
| 試料ステージ | 試料ホルダーモデル: OTHER / ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A / Chain - Residue range: 1-760 / Chain - Source name: PDB / Chain - Initial model type: experimental model |
|---|---|
| 詳細 | The structure of P1 was fitted in the map using COOT as a rigid body in two different positions corresponding to subunits P1A and P1B. P1A and P1B main-chains and side-chains were adjusted using manual and real space fitting in COOT. Structure was refined in Phenix.real_space_refine applying secondary structure, rotamer, and Ramachandran plot restraints. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-5muu: |
-原子モデル構築 2
| 詳細 | The structure of P8 (L3 to Y147) was built manually in COOT and side-chains were adjusted using manual and real space fitting in COOT. Structure was refined in Phenix.real_space_refine applying secondary structure, rotamer, and Ramachandran plot restraints. |
|---|---|
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-5muu: |
-原子モデル構築 3
| 詳細 | The structure of P4 C-terminus (R292 to L332) was built manually in COOT and side-chains were adjusted using manual and real space fitting in COOT. Structure was refined in Phenix.real_space_refine applying secondary structure, rotamer, and Ramachandran plot restraints. |
|---|---|
| 精密化 | プロトコル: AB INITIO MODEL |
| 得られたモデル |  PDB-5muu: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)