+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the DMCHA-bound mTAAR9-Gs complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | DMCHA / mTAAR9 / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Amine ligand-binding receptors / trace-amine receptor activity / G alpha (s) signalling events / Olfactory Signaling Pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / Activation of the phototransduction cascade / G beta:gamma signalling through PLC beta / Presynaptic function of Kainate receptors / Thromboxane signalling through TP receptor / G protein-coupled acetylcholine receptor signaling pathway ...Amine ligand-binding receptors / trace-amine receptor activity / G alpha (s) signalling events / Olfactory Signaling Pathway / adenylate cyclase-activating G protein-coupled receptor signaling pathway / Activation of the phototransduction cascade / G beta:gamma signalling through PLC beta / Presynaptic function of Kainate receptors / Thromboxane signalling through TP receptor / G protein-coupled acetylcholine receptor signaling pathway / Activation of G protein gated Potassium channels / Inhibition of voltage gated Ca2+ channels via Gbeta/gamma subunits / G-protein activation / G beta:gamma signalling through CDC42 / Prostacyclin signalling through prostacyclin receptor / Glucagon signaling in metabolic regulation / G beta:gamma signalling through BTK / Synthesis, secretion, and inactivation of Glucagon-like Peptide-1 (GLP-1) / ADP signalling through P2Y purinoceptor 12 / photoreceptor disc membrane / Glucagon-type ligand receptors / Sensory perception of sweet, bitter, and umami (glutamate) taste / Adrenaline,noradrenaline inhibits insulin secretion / Vasopressin regulates renal water homeostasis via Aquaporins / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / G alpha (z) signalling events / ADP signalling through P2Y purinoceptor 1 / cellular response to catecholamine stimulus / ADORA2B mediated anti-inflammatory cytokines production / G beta:gamma signalling through PI3Kgamma / adenylate cyclase-activating dopamine receptor signaling pathway / Cooperation of PDCL (PhLP1) and TRiC/CCT in G-protein beta folding / GPER1 signaling / G-protein beta-subunit binding / cellular response to prostaglandin E stimulus / heterotrimeric G-protein complex / G alpha (12/13) signalling events / Inactivation, recovery and regulation of the phototransduction cascade / extracellular vesicle / sensory perception of taste / Thrombin signalling through proteinase activated receptors (PARs) / signaling receptor complex adaptor activity / retina development in camera-type eye / GTPase binding / Ca2+ pathway / fibroblast proliferation / High laminar flow shear stress activates signaling by PIEZO1 and PECAM1:CDH5:KDR in endothelial cells / G alpha (i) signalling events / G alpha (s) signalling events / phospholipase C-activating G protein-coupled receptor signaling pathway / G alpha (q) signalling events / Ras protein signal transduction / Extra-nuclear estrogen signaling / cell population proliferation / G protein-coupled receptor signaling pathway / lysosomal membrane / GTPase activity / synapse / protein-containing complex binding / signal transduction / extracellular exosome / membrane / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.46 Å | |||||||||
 データ登録者 データ登録者 | Sun JP / Li Q / Yang F / Xu YF / Guo LL / Lian S / Zhang MH / Rong NK | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2023 ジャーナル: Nature / 年: 2023タイトル: Structural basis of amine odorant perception by a mammal olfactory receptor. 著者: Lulu Guo / Jie Cheng / Shuo Lian / Qun Liu / Yan Lu / Yuan Zheng / Kongkai Zhu / Minghui Zhang / Yalei Kong / Chao Zhang / Naikang Rong / Yuming Zhuang / Guoxing Fang / Jingjing Jiang / ...著者: Lulu Guo / Jie Cheng / Shuo Lian / Qun Liu / Yan Lu / Yuan Zheng / Kongkai Zhu / Minghui Zhang / Yalei Kong / Chao Zhang / Naikang Rong / Yuming Zhuang / Guoxing Fang / Jingjing Jiang / Tianyao Zhang / Xiang Han / Zili Liu / Ming Xia / Shangming Liu / Lei Zhang / Stephen D Liberles / Xiao Yu / Yunfei Xu / Fan Yang / Qian Li / Jin-Peng Sun /   要旨: Odorants are detected as smell in the nasal epithelium of mammals by two G-protein-coupled receptor families, the odorant receptors and the trace amine-associated receptors (TAARs). TAARs emerged ...Odorants are detected as smell in the nasal epithelium of mammals by two G-protein-coupled receptor families, the odorant receptors and the trace amine-associated receptors (TAARs). TAARs emerged following the divergence of jawed and jawless fish, and comprise a large monophyletic family of receptors that recognize volatile amine odorants to elicit both intraspecific and interspecific innate behaviours such as attraction and aversion. Here we report cryo-electron microscopy structures of mouse TAAR9 (mTAAR9) and mTAAR9-G or mTAAR9-G trimers in complex with β-phenylethylamine, N,N-dimethylcyclohexylamine or spermidine. The mTAAR9 structures contain a deep and tight ligand-binding pocket decorated with a conserved DWY motif, which is essential for amine odorant recognition. In the mTAAR9 structure, a unique disulfide bond connecting the N terminus to ECL2 is required for agonist-induced receptor activation. We identify key structural motifs of TAAR family members for detecting monoamines and polyamines and the shared sequence of different TAAR members that are responsible for recognition of the same odour chemical. We elucidate the molecular basis of mTAAR9 coupling to G and G by structural characterization and mutational analysis. Collectively, our results provide a structural basis for odorant detection, receptor activation and G coupling of an amine olfactory receptor. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35705.map.gz emd_35705.map.gz | 25.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35705-v30.xml emd-35705-v30.xml emd-35705.xml emd-35705.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_35705.png emd_35705.png | 122.6 KB | ||
| Filedesc metadata |  emd-35705.cif.gz emd-35705.cif.gz | 6.8 KB | ||
| その他 |  emd_35705_half_map_1.map.gz emd_35705_half_map_1.map.gz emd_35705_half_map_2.map.gz emd_35705_half_map_2.map.gz | 23.4 MB 23.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35705 http://ftp.pdbj.org/pub/emdb/structures/EMD-35705 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35705 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35705 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8itfMC  8iw1C  8iw4C  8iw7C  8iw9C  8iweC  8iwmC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35705.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35705.map.gz / 形式: CCP4 / 大きさ: 30.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.85 Å | ||||||||||||||||||||||||||||||||||||
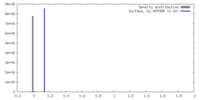
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_35705_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
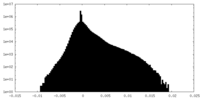
| 投影像・断面図 |
| ||||||||||||
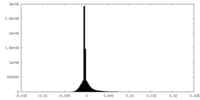
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35705_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
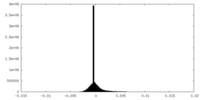
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cryo-EM structure of the DMCHA-bound mTAAR9-Gs complex
| 全体 | 名称: Cryo-EM structure of the DMCHA-bound mTAAR9-Gs complex |
|---|---|
| 要素 |
|
-超分子 #1: Cryo-EM structure of the DMCHA-bound mTAAR9-Gs complex
| 超分子 | 名称: Cryo-EM structure of the DMCHA-bound mTAAR9-Gs complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#6 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Guanine nucleotide-binding protein G(s) subunit alpha isoforms short
| 分子 | 名称: Guanine nucleotide-binding protein G(s) subunit alpha isoforms short タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 42.010664 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MMGCTLSAED KAAVERSKMI EKQLQKDKQV YRATHRLLLL GADNSGKSTI VKQMRIYHVN GYSEEECKQY KAVVYSNTIQ SIIAIIRAM GRLKIDFGDS ARADDARQLF VLAGAAEEGF MTAELAGVIK RLWKDSGVQA CFNRSREYQL NDSAAYYLND L DRIAQPNY ...文字列: MMGCTLSAED KAAVERSKMI EKQLQKDKQV YRATHRLLLL GADNSGKSTI VKQMRIYHVN GYSEEECKQY KAVVYSNTIQ SIIAIIRAM GRLKIDFGDS ARADDARQLF VLAGAAEEGF MTAELAGVIK RLWKDSGVQA CFNRSREYQL NDSAAYYLND L DRIAQPNY IPTQQDVLRT RVKTSGIFET KFQVDKVNFH MFDVGAQRDE RRKWIQCFND VTAIIFVVDS SDYNRLQEAL ND FKSIWNN RWLRTISVIL FLNKQDLLAE KVLAGKSKIE DYFPEFARYT TPEDATPEPG EDPRVTRAKY FIRDEFLRIS TAS GDGRHY CYPHFTCSVD TENARRIFND CRDIIQRMHL RQYELL |
-分子 #2: Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1
| 分子 | 名称: Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1 タイプ: protein_or_peptide / ID: 2 詳細: Author stated: residues (-9) - (-4) is His Tag, residues 341-355 is Linker, residues 356 -366 is Small Bit. コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 41.055867 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MHHHHHHGSL LQSELDQLRQ EAEQLKNQIR DARKACADAT LSQITNNIDP VGRIQMRTRR TLRGHLAKIY AMHWGTDSRL LVSASQDGK LIIWDSYTTN KVHAIPLRSS WVMTCAYAPS GNYVACGGLD NICSIYNLKT REGNVRVSRE LAGHTGYLSC C RFLDDNQI ...文字列: MHHHHHHGSL LQSELDQLRQ EAEQLKNQIR DARKACADAT LSQITNNIDP VGRIQMRTRR TLRGHLAKIY AMHWGTDSRL LVSASQDGK LIIWDSYTTN KVHAIPLRSS WVMTCAYAPS GNYVACGGLD NICSIYNLKT REGNVRVSRE LAGHTGYLSC C RFLDDNQI VTSSGDTTCA LWDIETGQQT TTFTGHTGDV MSLSLAPDTR LFVSGACDAS AKLWDVREGM CRQTFTGHES DI NAICFFP NGNAFATGSD DATCRLFDLR ADQELMTYSH DNIICGITSV SFSKSGRLLL AGYDDFNCNV WDALKADRAG VLA GHDNRV SCLGVTDDGM AVATGSWDSF LKIWNGSSGG GGSGGGGSSG VSGWRLFKKI S UniProtKB: Guanine nucleotide-binding protein G(I)/G(S)/G(T) subunit beta-1 |
-分子 #3: Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2
| 分子 | 名称: Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2 タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 6.375332 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: NTASIAQARK LVEQLKMEAN IDRIKVSKAA ADLMAYCEAH AKEDPLLTPV PASENPFR UniProtKB: Guanine nucleotide-binding protein G(I)/G(S)/G(O) subunit gamma-2 |
-分子 #4: Trace amine-associated receptor 9
| 分子 | 名称: Trace amine-associated receptor 9 / タイプ: protein_or_peptide / ID: 4 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 38.928238 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MTSDFSPEPP MELCYENVNG SCIKSSYAPW PRAILYGVLG LGALLAVFGN LLVIIAILHF KQLHTPTNFL VASLACADFL VGVTVMPFS TVRSVESCWY FGESYCKFHT CFDTSFCFAS LFHLCCISID RYIAVTDPLT YPTKFTVSVS GLCIALSWFF S VTYSFSIF ...文字列: MTSDFSPEPP MELCYENVNG SCIKSSYAPW PRAILYGVLG LGALLAVFGN LLVIIAILHF KQLHTPTNFL VASLACADFL VGVTVMPFS TVRSVESCWY FGESYCKFHT CFDTSFCFAS LFHLCCISID RYIAVTDPLT YPTKFTVSVS GLCIALSWFF S VTYSFSIF YTGANEEGIE ELVVALTCVG GCQAPLNQNW VLLCFLLFFL PTVVMVFLYG RIFLVAKYQA RKIEGTANQA QA SSESYKE RVAKRERKAA KTLGIAMAAF LVSWLPYIID AVIDAYMNFI TPAYVYEILV WCVYYNSAMN PLIYAFFYPW FRK AIKLIV SGKVFRADSS TTNLFSEEAG AG UniProtKB: Trace amine-associated receptor 9 |
-分子 #5: scFv16
| 分子 | 名称: scFv16 / タイプ: protein_or_peptide / ID: 5 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 30.363043 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MLLVNQSHQG FNKEHTSKMV SAIVLYVLLA AAAHSAFAVQ LVESGGGLVQ PGGSRKLSCS ASGFAFSSFG MHWVRQAPEK GLEWVAYIS SGSGTIYYAD TVKGRFTISR DDPKNTLFLQ MTSLRSEDTA MYYCVRSIYY YGSSPFDFWG QGTTLTVSAG G GGSGGGGS ...文字列: MLLVNQSHQG FNKEHTSKMV SAIVLYVLLA AAAHSAFAVQ LVESGGGLVQ PGGSRKLSCS ASGFAFSSFG MHWVRQAPEK GLEWVAYIS SGSGTIYYAD TVKGRFTISR DDPKNTLFLQ MTSLRSEDTA MYYCVRSIYY YGSSPFDFWG QGTTLTVSAG G GGSGGGGS GGGGSADIVM TQATSSVPVT PGESVSISCR SSKSLLHSNG NTYLYWFLQR PGQSPQLLIY RMSNLASGVP DR FSGSGSG TAFTLTISRL EAEDVGVYYC MQHLEYPLTF GAGTKLEL |
-分子 #6: Nanobody-35
| 分子 | 名称: Nanobody-35 / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.885439 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: QVQLQESGGG LVQPGGSLRL SCAASGFTFS NYKMNWVRQA PGKGLEWVSD ISQSGASISY TGSVKGRFTI SRDNAKNTLY LQMNSLKPE DTAVYYCARC PAPFTRDCFD VTSTTYAYRG QGTQVTVSS |
-分子 #7: ~{N},~{N}-dimethylcyclohexanamine
| 分子 | 名称: ~{N},~{N}-dimethylcyclohexanamine / タイプ: ligand / ID: 7 / コピー数: 1 / 式: 8IA |
|---|---|
| 分子量 | 理論値: 127.227 Da |
| Chemical component information | 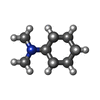 ChemComp-8IA: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 1.875 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: DIFFRACTION / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.46 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 301281 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)