+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Bi-functional malonyl-CoA reductuase from Chloroflexus aurantiacus | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | malonyl-CoA reductase (MCR) / bi-functional enzyme / NADPH-dependent reduction / OXIDOREDUCTASE | |||||||||
| 機能・相同性 | fatty acid elongation / oxidoreductase activity, acting on the CH-OH group of donors, NAD or NADP as acceptor / short chain dehydrogenase / Enoyl-(Acyl carrier protein) reductase / Short-chain dehydrogenase/reductase SDR / NAD(P)-binding domain superfamily / nucleotide binding / metal ion binding / Short-chain dehydrogenase/reductase SDR 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Chloroflexus aurantiacus (バクテリア) Chloroflexus aurantiacus (バクテリア) | |||||||||
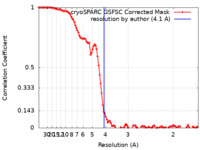
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Ahn JW / Kim S | |||||||||
| 資金援助 |  韓国, 2件 韓国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Int J Biol Macromol / 年: 2023 ジャーナル: Int J Biol Macromol / 年: 2023タイトル: Cryo-EM structure of bifunctional malonyl-CoA reductase from Chloroflexus aurantiacus reveals a dynamic domain movement for high enzymatic activity. 著者: Jae-Woo Ahn / Sangwoo Kim / Jiyeon Hong / Kyung-Jin Kim /  要旨: The platform chemical 3-hydroxypropionic acid is used to synthesize various valuable materials, including bioplastics. Bifunctional malonyl-CoA reductase is a key enzyme in 3-hydroxypropionic acid ...The platform chemical 3-hydroxypropionic acid is used to synthesize various valuable materials, including bioplastics. Bifunctional malonyl-CoA reductase is a key enzyme in 3-hydroxypropionic acid biosynthesis as it catalyzes the two-step reduction of malonyl-CoA to malonate semialdehyde to 3-hydroxypropionic acid. Here, we report the cryo-EM structure of a full-length malonyl-CoA reductase protein from Chloroflexus aurantiacus (CaMCR). The EM model of CaMCR reveals a tandem helix architecture comprising an N-terminal (CaMCR) and a C-terminal (CaMCR) domain. The CaMCR model also revealed that the enzyme undergoes a dynamic domain movement between CaMCR and CaMCR due to the presence of a flexible linker between these two domains. Increasing the flexibility and extension of the linker resulted in a twofold increase in enzyme activity, indicating that for CaMCR, domain movement is crucial for high enzyme activity. We also describe the structural features of CaMCR and CaMCR. This study reveals the protein structures underlying the molecular mechanism of CaMCR and thereby provides valuable information for future enzyme engineering to improve the productivity of 3-hydroxypropionic acid. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34840.map.gz emd_34840.map.gz | 113 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34840-v30.xml emd-34840-v30.xml emd-34840.xml emd-34840.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34840_fsc.xml emd_34840_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34840.png emd_34840.png | 142.8 KB | ||
| マスクデータ |  emd_34840_msk_1.map emd_34840_msk_1.map | 125 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-34840.cif.gz emd-34840.cif.gz | 6.6 KB | ||
| その他 |  emd_34840_half_map_1.map.gz emd_34840_half_map_1.map.gz emd_34840_half_map_2.map.gz emd_34840_half_map_2.map.gz | 115.7 MB 115.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34840 http://ftp.pdbj.org/pub/emdb/structures/EMD-34840 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34840 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34840 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34840_validation.pdf.gz emd_34840_validation.pdf.gz | 991.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34840_full_validation.pdf.gz emd_34840_full_validation.pdf.gz | 991.1 KB | 表示 | |
| XML形式データ |  emd_34840_validation.xml.gz emd_34840_validation.xml.gz | 19.2 KB | 表示 | |
| CIF形式データ |  emd_34840_validation.cif.gz emd_34840_validation.cif.gz | 24.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34840 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34840 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34840 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34840 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hjwMC  8i6zC  8i70C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34840.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34840.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_34840_msk_1.map emd_34840_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
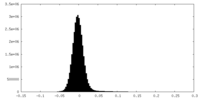
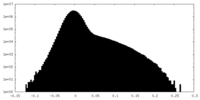
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_34840_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34840_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Dimeric state of malonyl-CoA reductase (MCR) from Chloroflexus au...
| 全体 | 名称: Dimeric state of malonyl-CoA reductase (MCR) from Chloroflexus aurantiacus |
|---|---|
| 要素 |
|
-超分子 #1: Dimeric state of malonyl-CoA reductase (MCR) from Chloroflexus au...
| 超分子 | 名称: Dimeric state of malonyl-CoA reductase (MCR) from Chloroflexus aurantiacus タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Chloroflexus aurantiacus (バクテリア) Chloroflexus aurantiacus (バクテリア) |
| 分子量 | 理論値: 264 KDa |
-分子 #1: Short-chain dehydrogenase/reductase SDR
| 分子 | 名称: Short-chain dehydrogenase/reductase SDR / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Chloroflexus aurantiacus (バクテリア) Chloroflexus aurantiacus (バクテリア) |
| 分子量 | 理論値: 132.134219 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MSGTGRLAGK IALITGGAGN IGSELTRRFL AEGATVIISG RNRAKLTALA ERMQAEAGVP AKRIDLEVMD GSDPVAVRAG IEAIVARHG QIDILVNNAG SAGAQRRLAE IPLTEAELGP GAEETLHASI ANLLGMGWHL MRIAAPHMPV GSAVINVSTI F SRAEYYGR ...文字列: MSGTGRLAGK IALITGGAGN IGSELTRRFL AEGATVIISG RNRAKLTALA ERMQAEAGVP AKRIDLEVMD GSDPVAVRAG IEAIVARHG QIDILVNNAG SAGAQRRLAE IPLTEAELGP GAEETLHASI ANLLGMGWHL MRIAAPHMPV GSAVINVSTI F SRAEYYGR IPYVTPKAAL NALSQLAARE LGARGIRVNT IFPGPIESDR IRTVFQRMDQ LKGRPEGDTA HHFLNTMRLC RA NDQGALE RRFPSVGDVA DAAVFLASAE SAALSGETIE VTHGMELPAC SETSLLARTD LRTIDASGRT TLICAGDQIE EVM ALTGML RTCGSEVIIG FRSAAALAQF EQAVNESRRL AGADFTPPIA LPLDPRDPAT IDAVFDWAGE NTGGIHAAVI LPAT SHEPA PCVIEVDDER VLNFLADEIT GTIVIASRLA RYWQSQRLTP GARARGPRVI FLSNGADQNG NVYGRIQSAA IGQLI RVWR HEAELDYQRA SAAGDHVLPP VWANQIVRFA NRSLEGLEFA CAWTAQLLHS QRHINEITLN IPANISATTG ARSASV GWA ESLIGLHLGK VALITGGSAG IGGQIGRLLA LSGARVMLAA RDRHKLEQMQ AMIQSELAEV GYTDVEDRVH IAPGCDV SS EAQLADLVER TLSAFGTVDY LINNAGIAGV EEMVIDMPVE GWRHTLFANL ISNYSLMRKL APLMKKQGSG YILNVSSY F GGEKDAAIPY PNRADYAVSK AGQRAMAEVF ARFLGPEIQI NAIAPGPVEG DRLRGTGERP GLFARRARLI LENKRLNEL HAALIAAART DERSMHELVE LLLPNDVAAL EQNPAAPTAL RELARRFRSE GDPAASSSSA LLNRSIAAKL LARLHNGGYV LPADIFANL PNPPDPFFTR AQIDREARKV RDGIMGMLYL QRMPTEFDVA MATVYYLADR NVSGETFHPS GGLRYERTPT G GELFGLPS PERLAELVGS TVYLIGEHLT EHLNLLARAY LERYGARQVV MIVETETGAE TMRRLLHDHV EAGRLMTIVA GD QIEAAID QAITRYGRPG PVVCTPFRPL PTVPLVGRKD SDWSTVLSEA EFAELCEHQL THHFRVARKI ALSDGASLAL VTP ETTATS TTEQFALANF IKTTLHAFTA TIGVESERTA QRILINQVDL TRRARAEEPR DPHERQQELE RFIEAVLLVT APLP PEADT RYAGRIHRGR AITV UniProtKB: Short-chain dehydrogenase/reductase SDR |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 20mM Tris-HCl, 150mM NaCl |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.1 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)