+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Rat GluN1-GluN2C NMDA receptor in complex with glycine and glutamate (minor class in symmetry) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | NMDA receptor / GluN2C / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報directional locomotion / pons maturation / positive regulation of Schwann cell migration / regulation of cell communication / EPHB-mediated forward signaling / Assembly and cell surface presentation of NMDA receptors / olfactory learning / conditioned taste aversion / dendritic branch / regulation of respiratory gaseous exchange ...directional locomotion / pons maturation / positive regulation of Schwann cell migration / regulation of cell communication / EPHB-mediated forward signaling / Assembly and cell surface presentation of NMDA receptors / olfactory learning / conditioned taste aversion / dendritic branch / regulation of respiratory gaseous exchange / protein localization to postsynaptic membrane / transmitter-gated monoatomic ion channel activity / suckling behavior / response to glycine / propylene metabolic process / regulation of monoatomic cation transmembrane transport / NMDA glutamate receptor activity / Synaptic adhesion-like molecules / RAF/MAP kinase cascade / voltage-gated monoatomic cation channel activity / response to glycoside / NMDA selective glutamate receptor complex / neurotransmitter receptor complex / ligand-gated sodium channel activity / glutamate binding / response to morphine / calcium ion transmembrane import into cytosol / regulation of axonogenesis / neuromuscular process controlling balance / neuromuscular process / regulation of dendrite morphogenesis / protein heterotetramerization / male mating behavior / glycine binding / regulation of synapse assembly / response to amine / parallel fiber to Purkinje cell synapse / startle response / positive regulation of reactive oxygen species biosynthetic process / monoatomic cation transmembrane transport / positive regulation of calcium ion transport into cytosol / regulation of neuron apoptotic process / associative learning / cellular response to glycine / monoatomic cation transport / excitatory synapse / positive regulation of dendritic spine maintenance / social behavior / regulation of neuronal synaptic plasticity / monoatomic ion channel complex / positive regulation of excitatory postsynaptic potential / glutamate receptor binding / Unblocking of NMDA receptors, glutamate binding and activation / long-term memory / positive regulation of synaptic transmission, glutamatergic / synaptic cleft / prepulse inhibition / phosphatase binding / monoatomic cation channel activity / calcium ion homeostasis / response to fungicide / glutamate-gated receptor activity / glutamate-gated calcium ion channel activity / cellular response to manganese ion / presynaptic active zone membrane / sensory perception of pain / dendrite membrane / ligand-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / sodium ion transmembrane transport / ionotropic glutamate receptor signaling pathway / synaptic membrane / hippocampal mossy fiber to CA3 synapse / response to amphetamine / adult locomotory behavior / excitatory postsynaptic potential / regulation of membrane potential / learning / transmitter-gated monoatomic ion channel activity involved in regulation of postsynaptic membrane potential / synaptic transmission, glutamatergic / PDZ domain binding / regulation of long-term neuronal synaptic plasticity / response to calcium ion / postsynaptic density membrane / negative regulation of protein catabolic process / neuron cellular homeostasis / calcium ion transmembrane transport / visual learning / regulation of synaptic plasticity / cerebral cortex development / calcium channel activity / response to wounding / memory / long-term synaptic potentiation / intracellular calcium ion homeostasis / terminal bouton / synaptic vesicle / synaptic vesicle membrane / calcium ion transport / rhythmic process / signaling receptor activity 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / 解像度: 4.3 Å | |||||||||
 データ登録者 データ登録者 | Zhang M / Zhang J / Guo F / Li Y / Zhu S | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Distinct structure and gating mechanism in diverse NMDA receptors with GluN2C and GluN2D subunits. 著者: Jilin Zhang / Ming Zhang / Qinrui Wang / Han Wen / Zheyi Liu / Fangjun Wang / Yuhang Wang / Fenyong Yao / Nan Song / Zengwei Kou / Yang Li / Fei Guo / Shujia Zhu /  要旨: N-methyl-D-aspartate (NMDA) receptors are heterotetramers comprising two GluN1 and two alternate GluN2 (N2A-N2D) subunits. Here we report full-length cryo-EM structures of the human N1-N2D di- ...N-methyl-D-aspartate (NMDA) receptors are heterotetramers comprising two GluN1 and two alternate GluN2 (N2A-N2D) subunits. Here we report full-length cryo-EM structures of the human N1-N2D di-heterotetramer (di-receptor), rat N1-N2C di-receptor and N1-N2A-N2C tri-heterotetramer (tri-receptor) at a best resolution of 3.0 Å. The bilobate N-terminal domain (NTD) in N2D intrinsically adopts a closed conformation, leading to a compact NTD tetramer in the N1-N2D receptor. Additionally, crosslinking the ligand-binding domain (LBD) of two N1 protomers significantly elevated the channel open probability (Po) in N1-N2D di-receptors. Surprisingly, the N1-N2C di-receptor adopted both symmetric (minor) and asymmetric (major) conformations, the latter further locked by an allosteric potentiator, PYD-106, binding to a pocket between the NTD and LBD in only one N2C protomer. Finally, the N2A and N2C subunits in the N1-N2A-N2C tri-receptor display a conformation close to one protomer in the N1-N2A and N1-N2C di-receptors, respectively. These findings provide a comprehensive structural understanding of diverse function in major NMDA receptor subtypes. #1:  ジャーナル: Nat.Struct.Mol.Biol. / 年: 2023 ジャーナル: Nat.Struct.Mol.Biol. / 年: 2023タイトル: Distinct structure and gating mechanism in diverse NMDA receptors with GluN2C and GluN2D subunits 著者: Zhang M / Zhu S | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34674.map.gz emd_34674.map.gz | 45.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34674-v30.xml emd-34674-v30.xml emd-34674.xml emd-34674.xml | 20.3 KB 20.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34674_fsc.xml emd_34674_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34674.png emd_34674.png | 50.1 KB | ||
| Filedesc metadata |  emd-34674.cif.gz emd-34674.cif.gz | 6.6 KB | ||
| その他 |  emd_34674_half_map_1.map.gz emd_34674_half_map_1.map.gz emd_34674_half_map_2.map.gz emd_34674_half_map_2.map.gz | 86.1 MB 86.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34674 http://ftp.pdbj.org/pub/emdb/structures/EMD-34674 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34674 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34674 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34674_validation.pdf.gz emd_34674_validation.pdf.gz | 959.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34674_full_validation.pdf.gz emd_34674_full_validation.pdf.gz | 958.8 KB | 表示 | |
| XML形式データ |  emd_34674_validation.xml.gz emd_34674_validation.xml.gz | 17.7 KB | 表示 | |
| CIF形式データ |  emd_34674_validation.cif.gz emd_34674_validation.cif.gz | 23.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34674 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34674 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34674 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34674 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hdkMC  7yffC  7yfgC  7yfhC  7yfiC  7yflC  7yfmC  7yfoC  7yfrC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34674.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34674.map.gz / 形式: CCP4 / 大きさ: 93 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.071 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_34674_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
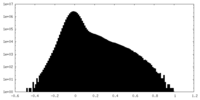
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34674_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
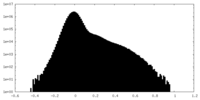
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Rat GluN1-GluN2C NMDA receptor in complex with glycine and glutamate
| 全体 | 名称: Rat GluN1-GluN2C NMDA receptor in complex with glycine and glutamate |
|---|---|
| 要素 |
|
-超分子 #1: Rat GluN1-GluN2C NMDA receptor in complex with glycine and glutamate
| 超分子 | 名称: Rat GluN1-GluN2C NMDA receptor in complex with glycine and glutamate タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Glutamate receptor ionotropic, NMDA 1
| 分子 | 名称: Glutamate receptor ionotropic, NMDA 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 89.724539 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MSTMHLLTFA LLFSCSFARA ACDPKIVNIG AVLSTRKHEQ MFREAVNQAN KRHGSWKIQL NATSVTHKPN AIQMALSVCE DLISSQVYA ILVSHPPTPN DHFTPTPVSY TAGFYRIPVL GLTTRMSIYS DKSIHLSFLR TVPPYSHQSS VWFEMMRVYN W NHIILLVS ...文字列: MSTMHLLTFA LLFSCSFARA ACDPKIVNIG AVLSTRKHEQ MFREAVNQAN KRHGSWKIQL NATSVTHKPN AIQMALSVCE DLISSQVYA ILVSHPPTPN DHFTPTPVSY TAGFYRIPVL GLTTRMSIYS DKSIHLSFLR TVPPYSHQSS VWFEMMRVYN W NHIILLVS DDHEGRAAQK RLETLLEERE SKAEKVLQFD PGTKNVTALL MEARELEARV IILSASEDDA ATVYRAAAML NM TGSGYVW LVGEREISGN ALRYAPDGII GLQLINGKNE SAHISDAVGV VAQAVHELLE KENITDPPRG CVGNTNIWKT GPL FKRVLM SSKYADGVTG RVEFNEDGDR KFANYSIMNL QNRKLVQVGI YNGTHVIPND RKIIWPGGET EKPRGYQMST RLKI VTIHQ EPFVYVKPTM SDGTCKEEFT VNGDPVKKVI CTGPNDTSPG SPRHTVPQCC YGFCIDLLIK LARTMNFTYE VHLVA DGKF GTQERVNNSN KKEWNGMMGE LLSGQADMIV APLTINNERA QYIEFSKPFK YQGLTILVKK EIPRSTLDSF MQPFQS TLW LLVGLSVHVV AVMLYLLDRF SPFGRFKVNS EEEEEDALTL SSAMWFSWGV LLNSGIGEGA PRSFSARILG MVWAGFA MI IVASYTANLA AFLVLDRPEE RITGINDPRL RNPSDKFIYA TVKQSSVDIY FRRQVELSTM YRHMEKHNYE SAAEAIQA V RDNKLHAFIW DSAVLEFEAS QKCDLVTTGE LFFRSGFGIG MRKDSPWKQN VSLSILKSHE NGFMEDLDKT WVRYQ UniProtKB: Glutamate receptor ionotropic, NMDA 1 |
-分子 #2: Glutamate receptor ionotropic, NMDA 2C
| 分子 | 名称: Glutamate receptor ionotropic, NMDA 2C / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 87.753758 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGGALGPALL LTSLLGAWAR LGAGQGEQAV TVAVVFGSSG PLQTQARTRL TSQNFLDLPL EIQPLTVGVN NTNPSSILTQ ICGLLGAAR VHGIVFEDNV DTEAVAQLLD FVSSQTHVPI LSISGGSAVV LTPKEPGSAF LQLGVSLEQQ LQVLFKVLEE Y DWSAFAVI ...文字列: MGGALGPALL LTSLLGAWAR LGAGQGEQAV TVAVVFGSSG PLQTQARTRL TSQNFLDLPL EIQPLTVGVN NTNPSSILTQ ICGLLGAAR VHGIVFEDNV DTEAVAQLLD FVSSQTHVPI LSISGGSAVV LTPKEPGSAF LQLGVSLEQQ LQVLFKVLEE Y DWSAFAVI TSLHPGHALF LEGVRAVADA SYLSWRLLDV LTLELGPGGP RARTQRLLRQ VDAPVLVAYC SREEAEVLFA EA AQAGLVG PGHVWLVPNL ALGSTDAPPA AFPVGLISVV TESWRLSLRQ KVRDGVAILA LGAHSYRRQY GTLPAPAGDC RSH PGPVSP AREAFYRHLL NVTWEGRDFS FSPGGYLVRP TMVVIALNRH RLWEMVGRWD HGVLYMKYPV WPRYSTSLQP VVDS RHLTV ATLEERPFVI VESPDPGTGG CVPNTVPCRR QSNHTFSSGD LTPYTKLCCK GFCIDILKKL AKVVKFSYDL YLVTN GKHG KRVRGVWNGM IGEVYYKRAD MAIGSLTINE ERSEIIDFSV PFVETGISVM VSRSNGTVSP SAFLEPYSPA VWVMMF VMC LTVVAITVFM FEYFSPVSYN QNLTKGKKPG GPSFTIGKSV WLLWALVFNN SVPIENPRGT TSKIMVLVWA FFAVIFL AS YTANLAAFMI QEQYIDTVSG LSDKKFQRPQ DQYPPFRFGT VPNGSTERNI RSNYRDMHTH MVKFNQRSVE DALTSLKM G KLDAFIYDAA VLNYMAGKDE GCKLVTIGSG KVFATTGYGI AMQKDSHWKR AIDLALLQLL GDGETQKLET VWLSGICQN UniProtKB: Glutamate receptor ionotropic, NMDA 2C |
-分子 #5: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 5 / コピー数: 12 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
 解析 解析 | 単粒子再構成法 |
|---|---|
| 試料の集合状態 | 2D array |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: DIRECT ELECTRON DE-10 (5k x 4k) #0 - 平均電子線量: 60.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: DIRECT ELECTRON DE-10 (5k x 4k) #1 - 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)