+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SARS-CoV-2 Omicron BA.1 spike trimer (6P) in complex with YB13-292 Fab, focused refinement of Fab region | ||||||||||||
 マップデータ マップデータ | Focused half map 2 of YB13-292 Fab complexed with Omicron S | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | Spike protein / RBD / antibody / Fab / Viral protein / VIRAL PROTEIN-IMMUNE SYSTEM complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion ...symbiont-mediated disruption of host tissue / Maturation of spike protein / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / viral translation / symbiont-mediated-mediated suppression of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / entry receptor-mediated virion attachment to host cell / membrane fusion / Attachment and Entry / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / host cell surface receptor binding / symbiont-mediated suppression of host innate immune response / receptor ligand activity / endocytosis involved in viral entry into host cell / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / identical protein binding / membrane / plasma membrane 類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
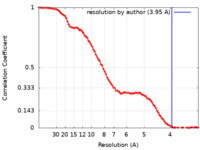
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.95 Å | ||||||||||||
 データ登録者 データ登録者 | Liu B / Gao X / Chen Q / Li Z / Su M / He J / Xiong X | ||||||||||||
| 資金援助 | 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Somatically hypermutated antibodies isolated from SARS-CoV-2 Delta infected patients cross-neutralize heterologous variants. 著者: Haisheng Yu / Banghui Liu / Yudi Zhang / Xijie Gao / Qian Wang / Haitao Xiang / Xiaofang Peng / Caixia Xie / Yaping Wang / Peiyu Hu / Jingrong Shi / Quan Shi / Pingqian Zheng / Chengqian Feng ...著者: Haisheng Yu / Banghui Liu / Yudi Zhang / Xijie Gao / Qian Wang / Haitao Xiang / Xiaofang Peng / Caixia Xie / Yaping Wang / Peiyu Hu / Jingrong Shi / Quan Shi / Pingqian Zheng / Chengqian Feng / Guofang Tang / Xiaopan Liu / Liliangzi Guo / Xiumei Lin / Jiaojiao Li / Chuanyu Liu / Yaling Huang / Naibo Yang / Qiuluan Chen / Zimu Li / Mengzhen Su / Qihong Yan / Rongjuan Pei / Xinwen Chen / Longqi Liu / Fengyu Hu / Dan Liang / Bixia Ke / Changwen Ke / Feng Li / Jun He / Meiniang Wang / Ling Chen / Xiaoli Xiong / Xiaoping Tang /  要旨: SARS-CoV-2 Omicron variants feature highly mutated spike proteins with extraordinary abilities in evading antibodies isolated earlier in the pandemic. Investigation of memory B cells from patients ...SARS-CoV-2 Omicron variants feature highly mutated spike proteins with extraordinary abilities in evading antibodies isolated earlier in the pandemic. Investigation of memory B cells from patients primarily with breakthrough infections with the Delta variant enables isolation of a number of neutralizing antibodies cross-reactive to heterologous variants of concern (VOCs) including Omicron variants (BA.1-BA.4). Structural studies identify altered complementarity determining region (CDR) amino acids and highly unusual heavy chain CDR2 insertions respectively in two representative cross-neutralizing antibodies-YB9-258 and YB13-292. These features are putatively introduced by somatic hypermutation and they are heavily involved in epitope recognition to broaden neutralization breadth. Previously, insertions/deletions were rarely reported for antiviral antibodies except for those induced by HIV-1 chronic infections. These data provide molecular mechanisms for cross-neutralization of heterologous SARS-CoV-2 variants by antibodies isolated from Delta variant infected patients with implications for future vaccination strategy. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34655.map.gz emd_34655.map.gz | 59.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34655-v30.xml emd-34655-v30.xml emd-34655.xml emd-34655.xml | 24.4 KB 24.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34655_fsc.xml emd_34655_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34655.png emd_34655.png | 26.1 KB | ||
| Filedesc metadata |  emd-34655.cif.gz emd-34655.cif.gz | 7.1 KB | ||
| その他 |  emd_34655_half_map_1.map.gz emd_34655_half_map_1.map.gz emd_34655_half_map_2.map.gz emd_34655_half_map_2.map.gz | 59.4 MB 59.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34655 http://ftp.pdbj.org/pub/emdb/structures/EMD-34655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34655 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34655 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_34655_validation.pdf.gz emd_34655_validation.pdf.gz | 798.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_34655_full_validation.pdf.gz emd_34655_full_validation.pdf.gz | 798 KB | 表示 | |
| XML形式データ |  emd_34655_validation.xml.gz emd_34655_validation.xml.gz | 15.5 KB | 表示 | |
| CIF形式データ |  emd_34655_validation.cif.gz emd_34655_validation.cif.gz | 21 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34655 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34655 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-34655 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hc8MC  8hc2C  8hc3C  8hc4C  8hc5C  8hc6C  8hc7C  8hc9C  8hcaC  8hcbC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34655.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34655.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused half map 2 of YB13-292 Fab complexed with Omicron S | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.65 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Focused map of YB13-292 Fab complexed with Omicron S
| ファイル | emd_34655_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused map of YB13-292 Fab complexed with Omicron S | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
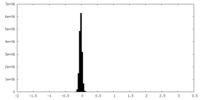
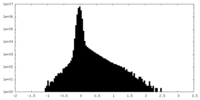
| 密度ヒストグラム |
-ハーフマップ: Focused half map 1 of YB13-292 Fab complexed with Omicron S
| ファイル | emd_34655_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused half map 1 of YB13-292 Fab complexed with Omicron S | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
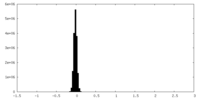
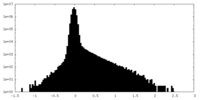
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SARS-CoV-2 Omicron BA.1 spike trimer with in complex with YB13-29...
| 全体 | 名称: SARS-CoV-2 Omicron BA.1 spike trimer with in complex with YB13-292 Fab, focused refinement of Fab region |
|---|---|
| 要素 |
|
-超分子 #1: SARS-CoV-2 Omicron BA.1 spike trimer with in complex with YB13-29...
| 超分子 | 名称: SARS-CoV-2 Omicron BA.1 spike trimer with in complex with YB13-292 Fab, focused refinement of Fab region タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 |
|---|
-超分子 #2: SARS-CoV-2 Omicron BA.1 spike protein
| 超分子 | 名称: SARS-CoV-2 Omicron BA.1 spike protein / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: YB13-292 Fab
| 超分子 | 名称: YB13-292 Fab / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Spike protein S1
| 分子 | 名称: Spike protein S1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 21.33207 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: ITNLCPFDEV FNATRFASVY AWNRKRISNC VADYSVLYNL APFFTFKCYG VSPTKLNDLC FTNVYADSFV IRGDEVRQIA PGQTGNIAD YNYKLPDDFT GCVIAWNSNK LDSKVSGNYN YLYRLFRKSN LKPFERDIST EIYQAGNKPC NGVAGFNCYF P LRSYSFRP TYGVGHQPYR VVVLSFEL UniProtKB: Spike glycoprotein |
-分子 #2: Heavy chain of YB13-292 Fab
| 分子 | 名称: Heavy chain of YB13-292 Fab / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 25.283396 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: EVQLVESGGG LVKPGGSLRL SCAASGFSFI TYNMNWVRQA PGKGLEWVSS ISSNILSSTS YIYYADSVKG RFTISRDDAA NSLFLQMNS LRVEDTAQYY CARTRSRSVR NCTSATCPVD AFDLWGQGTM VIVSSASTKG PSVFPLAPSS KSTSGGTAAL G CLVKDYFP ...文字列: EVQLVESGGG LVKPGGSLRL SCAASGFSFI TYNMNWVRQA PGKGLEWVSS ISSNILSSTS YIYYADSVKG RFTISRDDAA NSLFLQMNS LRVEDTAQYY CARTRSRSVR NCTSATCPVD AFDLWGQGTM VIVSSASTKG PSVFPLAPSS KSTSGGTAAL G CLVKDYFP EPVTVSWNSG ALTSGVHTFP AVLQSSGLYS LSSVVTVPSS SLGTQTYICN VNHKPSNTKV DKKVEPKSCD |
-分子 #3: Light chain of YB13-292 Fab
| 分子 | 名称: Light chain of YB13-292 Fab / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 23.976648 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIVLTQSPLS LPVTPGEPAS ISCRSSQSLL RSNGYNYLDW YLQKPGQSPH LLIYLGSNRA SGVPDRFSGS GSGTDFTLKI SRVEAEDVG VYYCMQALQT PYTFGQGTNL EIKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV ...文字列: DIVLTQSPLS LPVTPGEPAS ISCRSSQSLL RSNGYNYLDW YLQKPGQSPH LLIYLGSNRA SGVPDRFSGS GSGTDFTLKI SRVEAEDVG VYYCMQALQT PYTFGQGTNL EIKRTVAAPS VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL Q SGNSQESV TEQDSKDSTY SLSSTLTLSK ADYEKHKVYA CEVTHQGLSS PVTKSFNRGE C |
-分子 #4: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 4 / コピー数: 1 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)