+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3441 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Subtomogram average of the mitochondrial ATP synthase dimer from the ciliate Paramecium tetraurelia | |||||||||
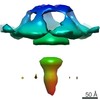
 マップデータ マップデータ | Subtomogram average of mitochondrial ATP synthase dimer from the ciliate Paramecium tetraurelia | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ATP synthase dimers / mitochondria / ciliate | |||||||||
| 生物種 |  | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 / 解像度: 26.0 Å | |||||||||
 データ登録者 データ登録者 | Muehleip AW / Kuehlbrandt W / Davies KM | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2016 ジャーナル: Proc Natl Acad Sci U S A / 年: 2016タイトル: Helical arrays of U-shaped ATP synthase dimers form tubular cristae in ciliate mitochondria. 著者: Alexander W Mühleip / Friederike Joos / Christoph Wigge / Achilleas S Frangakis / Werner Kühlbrandt / Karen M Davies /  要旨: F1Fo-ATP synthases are universal energy-converting membrane protein complexes that synthesize ATP from ADP and inorganic phosphate. In mitochondria of yeast and mammals, the ATP synthase forms V- ...F1Fo-ATP synthases are universal energy-converting membrane protein complexes that synthesize ATP from ADP and inorganic phosphate. In mitochondria of yeast and mammals, the ATP synthase forms V-shaped dimers, which assemble into rows along the highly curved ridges of lamellar cristae. Using electron cryotomography and subtomogram averaging, we have determined the in situ structure and organization of the mitochondrial ATP synthase dimer of the ciliate Paramecium tetraurelia. The ATP synthase forms U-shaped dimers with parallel monomers. Each complex has a prominent intracrista domain, which links the c-ring of one monomer to the peripheral stalk of the other. Close interaction of intracrista domains in adjacent dimers results in the formation of helical ATP synthase dimer arrays, which differ from the loose dimer rows in all other organisms observed so far. The parameters of the helical arrays match those of the cristae tubes, suggesting the unique features of the P. tetraurelia ATP synthase are directly responsible for generating the helical tubular cristae. We conclude that despite major structural differences between ATP synthase dimers of ciliates and other eukaryotes, the formation of ATP synthase dimer rows is a universal feature of mitochondria and a fundamental determinant of cristae morphology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3441.map.gz emd_3441.map.gz | 7.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3441-v30.xml emd-3441-v30.xml emd-3441.xml emd-3441.xml | 13.1 KB 13.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_3441.tif emd_3441.tif | 130.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3441 http://ftp.pdbj.org/pub/emdb/structures/EMD-3441 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3441 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3441 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3441_validation.pdf.gz emd_3441_validation.pdf.gz | 183.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3441_full_validation.pdf.gz emd_3441_full_validation.pdf.gz | 182.5 KB | 表示 | |
| XML形式データ |  emd_3441_validation.xml.gz emd_3441_validation.xml.gz | 5.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3441 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3441 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3441 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3441 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3441.map.gz / 形式: CCP4 / 大きさ: 10.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3441.map.gz / 形式: CCP4 / 大きさ: 10.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of mitochondrial ATP synthase dimer from the ciliate Paramecium tetraurelia | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.46 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mitochondrial ATP synthase dimer from Paramecium tetraurelia
| 全体 | 名称: Mitochondrial ATP synthase dimer from Paramecium tetraurelia |
|---|---|
| 要素 |
|
-超分子 #1000: Mitochondrial ATP synthase dimer from Paramecium tetraurelia
| 超分子 | 名称: Mitochondrial ATP synthase dimer from Paramecium tetraurelia タイプ: sample / ID: 1000 / 集合状態: dimer / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 1.2 MDa |
-分子 #1: Mitochondrial F1FoATPase
| 分子 | 名称: Mitochondrial F1FoATPase / タイプ: protein_or_peptide / ID: 1 / Name.synonym: Mitochondrial ATP synthase dimer / コピー数: 1 / 集合状態: Dimer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  株: d4-2 / 細胞: single-celled organism / Organelle: mitochondrion / 細胞中の位置: crista membrane |
| 分子量 | 理論値: 1.2 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 20 mM Tris, 250 mM trehalose |
|---|---|
| グリッド | 詳細: 300 mesh Quantifoil copper grid R2/2 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 10 % / チャンバー内温度: 110 K / 装置: OTHER 手法: manual blotting for 5-6 seconds with Whatman paper #4 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 80 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 64,000 times magnification |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Quantum, Gatan エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 日付 | 2013年7月3日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 実像数: 720 / 平均電子線量: 100 e/Å2 / カメラ長: 19.3 / ビット/ピクセル: 32 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 64000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER Tilt series - Axis1 - Min angle: -60 ° / Tilt series - Axis1 - Max angle: 60 ° / Tilt series - Axis1 - Angle increment: 2 ° |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | IMOD was used to align tilt series based on the position of gold fiducials, correct CTF on each projection image, and generate a tomographic volume using weighted back-projection. Subtomogram averaging was performed with PEET after initial estimate of rotations were calculated based on the orientation of particles relative to the membrane. |
|---|---|
| 最終 再構成 | アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 26.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: IMOD, PEET 詳細: Final subtomogram average was generated from 1244 subvolumes. 使用した粒子像数: 60 |
| CTF補正 | 詳細: each projection, strip-based approach |
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: C / Chain - #3 - Chain ID: D / Chain - #4 - Chain ID: E / Chain - #5 - Chain ID: F / Chain - #6 - Chain ID: G / Chain - #7 - Chain ID: H / Chain - #8 - Chain ID: I |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: RECIPROCAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: cross correlation coefficient maximization |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: S / Chain - #1 - Chain ID: W |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: RECIPROCAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: cross correlation coefficient maximization |
 ムービー
ムービー コントローラー
コントローラー









 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)























