[日本語] English
 万見
万見- EMDB-33706: Cryo-EM map of HEK293F cell-derived PEDV PT52 S T326I with three D0-up -
+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM map of HEK293F cell-derived PEDV PT52 S T326I with three D0-up | |||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 生物種 |  Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス) Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス) | |||||||||||||||||||||
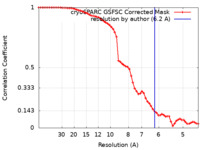
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.2 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Hsu STD / Draczkowski P / Wang YS | |||||||||||||||||||||
| 資金援助 |  台湾, 6件 台湾, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: In situ structure and dynamics of an alphacoronavirus spike protein by cryo-ET and cryo-EM. 著者: Cheng-Yu Huang / Piotr Draczkowski / Yong-Sheng Wang / Chia-Yu Chang / Yu-Chun Chien / Yun-Han Cheng / Yi-Min Wu / Chun-Hsiung Wang / Yuan-Chih Chang / Yen-Chen Chang / Tzu-Jing Yang / Yu-Xi ...著者: Cheng-Yu Huang / Piotr Draczkowski / Yong-Sheng Wang / Chia-Yu Chang / Yu-Chun Chien / Yun-Han Cheng / Yi-Min Wu / Chun-Hsiung Wang / Yuan-Chih Chang / Yen-Chen Chang / Tzu-Jing Yang / Yu-Xi Tsai / Kay-Hooi Khoo / Hui-Wen Chang / Shang-Te Danny Hsu /   要旨: Porcine epidemic diarrhea (PED) is a highly contagious swine disease caused by porcine epidemic diarrhea virus (PEDV). PED causes enteric disorders with an exceptionally high fatality in neonates, ...Porcine epidemic diarrhea (PED) is a highly contagious swine disease caused by porcine epidemic diarrhea virus (PEDV). PED causes enteric disorders with an exceptionally high fatality in neonates, bringing substantial economic losses in the pork industry. The trimeric spike (S) glycoprotein of PEDV is responsible for virus-host recognition, membrane fusion, and is the main target for vaccine development and antigenic analysis. The atomic structures of the recombinant PEDV S proteins of two different strains have been reported, but they reveal distinct N-terminal domain 0 (D0) architectures that may correspond to different functional states. The existence of the D0 is a unique feature of alphacoronavirus. Here we combined cryo-electron tomography (cryo-ET) and cryo-electron microscopy (cryo-EM) to demonstrate in situ the asynchronous S protein D0 motions on intact viral particles of a highly virulent PEDV Pintung 52 strain. We further determined the cryo-EM structure of the recombinant S protein derived from a porcine cell line, which revealed additional domain motions likely associated with receptor binding. By integrating mass spectrometry and cryo-EM, we delineated the complex compositions and spatial distribution of the PEDV S protein N-glycans, and demonstrated the functional role of a key N-glycan in modulating the D0 conformation. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33706.map.gz emd_33706.map.gz | 11.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33706-v30.xml emd-33706-v30.xml emd-33706.xml emd-33706.xml | 20.1 KB 20.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33706_fsc.xml emd_33706_fsc.xml | 6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33706.png emd_33706.png | 36.5 KB | ||
| その他 |  emd_33706_half_map_1.map.gz emd_33706_half_map_1.map.gz emd_33706_half_map_2.map.gz emd_33706_half_map_2.map.gz | 21.3 MB 21.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33706 http://ftp.pdbj.org/pub/emdb/structures/EMD-33706 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33706 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33706 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_33706_validation.pdf.gz emd_33706_validation.pdf.gz | 752.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_33706_full_validation.pdf.gz emd_33706_full_validation.pdf.gz | 751.7 KB | 表示 | |
| XML形式データ |  emd_33706_validation.xml.gz emd_33706_validation.xml.gz | 13.1 KB | 表示 | |
| CIF形式データ |  emd_33706_validation.cif.gz emd_33706_validation.cif.gz | 16.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33706 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33706 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33706 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-33706 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33706.map.gz / 形式: CCP4 / 大きさ: 23 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33706.map.gz / 形式: CCP4 / 大きさ: 23 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.2 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #1
| ファイル | emd_33706_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
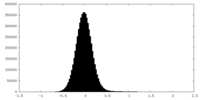
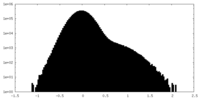
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33706_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
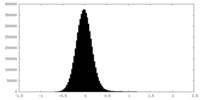
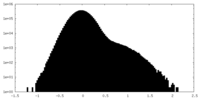
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : recombinant PEDV PT52 S protein expressed from HEK293F cells
| 全体 | 名称: recombinant PEDV PT52 S protein expressed from HEK293F cells |
|---|---|
| 要素 |
|
-超分子 #1: recombinant PEDV PT52 S protein expressed from HEK293F cells
| 超分子 | 名称: recombinant PEDV PT52 S protein expressed from HEK293F cells タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス) Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス)株: Pintung 52 |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293F Homo sapiens (ヒト) / 組換細胞: HEK293F |
-分子 #1: recombinant PEDV PT52 S T326I expressed from HEK293F cells
| 分子 | 名称: recombinant PEDV PT52 S T326I expressed from HEK293F cells タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス) Porcine epidemic diarrhea virus (ブタ流行性下痢ウイルス)株: Pintung52 |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MKSLTYFWLF LPVLSTLSLP QDVTRCSAKT NFRRFFSKFN VQAPAVVVLG GYLPIGENQG VNSTWYCAGQ HPTASGVHGI FVSHIRGGHG FEIGISQEPF DPSGYQLYLH KATNGNTNAT ARLRICQFPS IKTLGPTANN DVTIGRNCLF NKAIPAHMSE HSVVGITWDN ...文字列: MKSLTYFWLF LPVLSTLSLP QDVTRCSAKT NFRRFFSKFN VQAPAVVVLG GYLPIGENQG VNSTWYCAGQ HPTASGVHGI FVSHIRGGHG FEIGISQEPF DPSGYQLYLH KATNGNTNAT ARLRICQFPS IKTLGPTANN DVTIGRNCLF NKAIPAHMSE HSVVGITWDN DRVTVFSDKI YYFYFKNDWS RVATKCYNSG GCAMQYVYEP TYYMLNVTSA GEDGISYQPC TANCIGYAAN VFATEPNGHI PEGFSFNNWF LLSNDSTLVH GKVVSNQPLL VNCLLAIPKI YGLGQFFSFN QTIDGVCNGA AVQRAPEALR FNINDISVIL AEGSIVLHTA LGTNFSFVCS NSSNPHLATF AIPLGATQVP YYCFFKVDTY NSTVYKFLAV LPPTVREIVI TKYGDVYVNG FGYLHLGLLD AVTINFTGHG TDDDVSGFWT IASTNFVDAL IEVQGTAIQR ILYCDDPVSQ LKCSQVAFDL DDGFYPFSSR NLLSHEQPIS FVTLPSFNAH SFVNITVSAS FGGHSGANLI ASDTTINGFS SFCVDTRQFT ISLSYNVTNS YGYVSNSQDS NCPFTLQSVN DYLSFSKFCV STSLLASACT IDLFGYPEFG SGVKFTSLYF QFTKGELITG TPKPLEGVTD VSFMTLDVCT KYTIYGFKGE GIITLTNSSF LAGVYYTSDS GQLLAFKNVT SGAVYSVTPC SFSEQAAYVD DDIVGVISSL SSSTFNSTRE LPGFFYHSND GSNCTEPVLV YSNIGVCKSG SIGYVPSQSG QVKIAPTVTG NISIPTNFSM SIRTEYLQLY NTPVSVDCAT YVCNGNSRCK QLLTQYTAAC KTIESALQLS ARLESVEVNS MLTISEEALQ LATISSFNGD GYNFTNVLGV SVYDPARGRV VQKRSFIEDL LFNKVVTNGL GTVDEDYKRC SNGRSVADLV CAQYYSGVMV LPGVVDAEKL HMYSASLIGG MVLGGFTAAA ALPFSYAVQA RLNYLALQTD VLQRNQQLLA ESFNSAIGNI TSAFESVKEA SSQTSRGLNT VAHALTKVQE VVNSQGAALT QLTVQLQHNF QAISSSIDDI YSRLDPPSAD VQVDRLITGR LSALNAFVAQ TLTKYTEVQA SRKLAQQKVN ECVKSQSQRY GFCGGDGEHI FSLVQAAPQG LLFLHTVLVP SDFVDVIAIA GLCVNDEIAL TLREPGLVLF THELQNHTAT EYFVSSRRMF EPRKPTVSDF VQIESCVVTY VNLTRDQLPD VIPDYIDVNK TRDEILASLP NRTGPSLPLD VFNATYLNLT GEIADLEQRS ESLRNTTEEL QSLIYNINNT LVDLEWLNRV ETYIKWPEFG SGGYIPEAPR DGQAYVRKDG EWVLLSTFLK GQDNSADIQH SGRPLESRGP FEQKLISEED LNMHTGHHHH HH |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 構成要素:
詳細: Blot for 3 seconds before plunging. Force 0. | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | Cryo-EM map of HEK293F cell-derived PEDV PT52 S protein with three D0-down |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum |
| 詳細 | Data were recorded with stage tilt at 0 and 30 degree |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 2574 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.3000000000000003 µm 最小 デフォーカス(公称値): 1.8 µm / 倍率(公称値): 81000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー


























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)