+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3362 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Natively membrane-anchored full-length Herpes simplex virus 1 glycoprotein B | |||||||||
 マップデータ マップデータ | Subtomogram average of HSV-1 gB on membrane | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | membrane fusion / class III fusogen / pre-fusion | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報virion component => GO:0044423 / host cell Golgi membrane / host cell membrane / viral process / membrane => GO:0016020 / host cell endosome membrane / host cell endosome / host cell Golgi apparatus / viral envelope / symbiont entry into host cell ...virion component => GO:0044423 / host cell Golgi membrane / host cell membrane / viral process / membrane => GO:0016020 / host cell endosome membrane / host cell endosome / host cell Golgi apparatus / viral envelope / symbiont entry into host cell / virion attachment to host cell / host cell plasma membrane / virion membrane / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |   Human herpesvirus 1 (ヘルペスウイルス) Human herpesvirus 1 (ヘルペスウイルス) | |||||||||
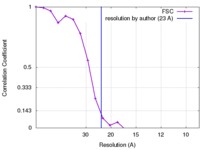
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 23.0 Å | |||||||||
 データ登録者 データ登録者 | Zeev-Ben-Mordehai T / Vasishtan D / Duran AH / Vollmer B / White P / Pandurangan AP / Siebert CA / Topf M / Grunewald K | |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2016 ジャーナル: Proc Natl Acad Sci U S A / 年: 2016タイトル: Two distinct trimeric conformations of natively membrane-anchored full-length herpes simplex virus 1 glycoprotein B. 著者: Tzviya Zeev-Ben-Mordehai / Daven Vasishtan / Anna Hernández Durán / Benjamin Vollmer / Paul White / Arun Prasad Pandurangan / C Alistair Siebert / Maya Topf / Kay Grünewald /  要旨: Many viruses are enveloped by a lipid bilayer acquired during assembly, which is typically studded with one or two types of glycoproteins. These viral surface proteins act as the primary interface ...Many viruses are enveloped by a lipid bilayer acquired during assembly, which is typically studded with one or two types of glycoproteins. These viral surface proteins act as the primary interface between the virus and the host. Entry of enveloped viruses relies on specialized fusogen proteins to help merge the virus membrane with the host membrane. In the multicomponent herpesvirus fusion machinery, glycoprotein B (gB) acts as this fusogen. Although the structure of the gB ectodomain postfusion conformation has been determined, any other conformations (e.g., prefusion, intermediate conformations) have so far remained elusive, thus restricting efforts to develop antiviral treatments and prophylactic vaccines. Here, we have characterized the full-length herpes simplex virus 1 gB in a native membrane by displaying it on cell-derived vesicles and using electron cryotomography. Alongside the known postfusion conformation, a novel one was identified. Its structure, in the context of the membrane, was determined by subvolume averaging and found to be trimeric like the postfusion conformation, but appeared more condensed. Hierarchical constrained density-fitting of domains unexpectedly revealed the fusion loops in this conformation to be apart and pointing away from the anchoring membrane. This vital observation is a substantial step forward in understanding the complex herpesvirus fusion mechanism, and opens up new opportunities for more targeted intervention of herpesvirus entry. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3362.map.gz emd_3362.map.gz | 1.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3362-v30.xml emd-3362-v30.xml emd-3362.xml emd-3362.xml | 11.9 KB 11.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3362_fsc.xml emd_3362_fsc.xml | 2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  3362_emdb_thumb.png 3362_emdb_thumb.png | 102.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3362 http://ftp.pdbj.org/pub/emdb/structures/EMD-3362 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3362 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3362 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3362_validation.pdf.gz emd_3362_validation.pdf.gz | 249.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3362_full_validation.pdf.gz emd_3362_full_validation.pdf.gz | 248.4 KB | 表示 | |
| XML形式データ |  emd_3362_validation.xml.gz emd_3362_validation.xml.gz | 6.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3362 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3362 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3362 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3362 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3362.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3362.map.gz / 形式: CCP4 / 大きさ: 1.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of HSV-1 gB on membrane | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.6 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Glycoprotein B of Herpes Simplex Virus 1
| 全体 | 名称: Glycoprotein B of Herpes Simplex Virus 1 |
|---|---|
| 要素 |
|
-超分子 #1000: Glycoprotein B of Herpes Simplex Virus 1
| 超分子 | 名称: Glycoprotein B of Herpes Simplex Virus 1 / タイプ: sample / ID: 1000 / 詳細: Sample bound to extracellular vesicles / 集合状態: Trimeric / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 300 KDa |
-分子 #1: Glycoprotein B
| 分子 | 名称: Glycoprotein B / タイプ: protein_or_peptide / ID: 1 / Name.synonym: gB / コピー数: 3 / 集合状態: Trimeric / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Human herpesvirus 1 (ヘルペスウイルス) / 別称: HSV-1 Human herpesvirus 1 (ヘルペスウイルス) / 別称: HSV-1 |
| 分子量 | 理論値: 300 KDa |
| 組換発現 | 生物種:  Mesocricetus auratus (ネズミ) / 組換細胞: BHK 21 / 組換プラスミド: pPEP98 Mesocricetus auratus (ネズミ) / 組換細胞: BHK 21 / 組換プラスミド: pPEP98 |
| 配列 | UniProtKB: Envelope glycoprotein B GO: viral process, virion attachment to host cell, symbiont entry into host cell, membrane, membrane => GO:0016020, virion component => GO:0044423, viral envelope, host cell plasma membrane, host ...GO: viral process, virion attachment to host cell, symbiont entry into host cell, membrane, membrane => GO:0016020, virion component => GO:0044423, viral envelope, host cell plasma membrane, host cell membrane, host cell endosome, host cell endosome membrane, host cell Golgi apparatus, host cell Golgi membrane, virion membrane InterPro: Herpesvirus Glycoprotein B |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 / 詳細: 25mM HEPES, 130mM NaCl |
|---|---|
| グリッド | 詳細: Holey carbon on top of 200 mesh gold grid. |
| 凍結 | 凍結剤: ETHANE-PROPANE MIXTURE / チャンバー内温度: 77 K / 装置: HOMEMADE PLUNGER / 手法: Blot for 3 sec before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 温度 | 最低: 80 K / 最高: 100 K / 平均: 85 K |
| 特殊光学系 | エネルギーフィルター - 名称: Gatan Quantum 964 エネルギーフィルター - エネルギー下限: 0.0 eV エネルギーフィルター - エネルギー上限: 20.0 eV |
| 詳細 | counting mode |
| 日付 | 2014年9月16日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 20 / 平均電子線量: 60 e/Å2 / ビット/ピクセル: 16 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 6.0 µm / 倍率(公称値): 95000 |
| 試料ステージ | 試料ホルダー: Polara holder / 試料ホルダーモデル: GATAN LIQUID NITROGEN / Tilt series - Axis1 - Min angle: -52 ° / Tilt series - Axis1 - Max angle: 53 ° |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称: Chimera, Flex-EM, ADP-EM, GMFit, TEMPy |
| 詳細 | Individual domains of 2GUM were fitted separately, using a hierarchical fitting approach |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT 当てはまり具合の基準: CCC, SCCC, Atom Protrusion Score |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)