+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3302 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the Salipro-POT complex at 6.48 A | |||||||||
 マップデータ マップデータ | Reconstruction of Salipro-POT | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | POT / salipro / lipid / disk / membrane / protein / transporter / peptider | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報dipeptide transmembrane transport / tripeptide transmembrane transport / tripeptide transmembrane transporter activity / peptide:proton symporter activity / dipeptide transmembrane transporter activity / identical protein binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Shewanella oneidensis (バクテリア) Shewanella oneidensis (バクテリア) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 6.48 Å | |||||||||
 データ登録者 データ登録者 | Frauenfeld J / Loeving R / Armache JP / Sonnen A / Guettou F / Moberg P / Zhu L / Jegerschoeld C / Flayhan A / Briggs J ...Frauenfeld J / Loeving R / Armache JP / Sonnen A / Guettou F / Moberg P / Zhu L / Jegerschoeld C / Flayhan A / Briggs J / Garoff H / Loew C / Cheng Y / Nordlund P | |||||||||
 引用 引用 |  ジャーナル: Nat Methods / 年: 2016 ジャーナル: Nat Methods / 年: 2016タイトル: A saposin-lipoprotein nanoparticle system for membrane proteins. 著者: Jens Frauenfeld / Robin Löving / Jean-Paul Armache / Andreas F-P Sonnen / Fatma Guettou / Per Moberg / Lin Zhu / Caroline Jegerschöld / Ali Flayhan / John A G Briggs / Henrik Garoff / ...著者: Jens Frauenfeld / Robin Löving / Jean-Paul Armache / Andreas F-P Sonnen / Fatma Guettou / Per Moberg / Lin Zhu / Caroline Jegerschöld / Ali Flayhan / John A G Briggs / Henrik Garoff / Christian Löw / Yifan Cheng / Pär Nordlund /    要旨: A limiting factor in membrane protein research is the ability to solubilize and stabilize such proteins. Detergents are used most often for solubilizing membrane proteins, but they are associated ...A limiting factor in membrane protein research is the ability to solubilize and stabilize such proteins. Detergents are used most often for solubilizing membrane proteins, but they are associated with protein instability and poor compatibility with structural and biophysical studies. Here we present a saposin-lipoprotein nanoparticle system, Salipro, which allows for the reconstitution of membrane proteins in a lipid environment that is stabilized by a scaffold of saposin proteins. We demonstrate the applicability of the method on two purified membrane protein complexes as well as by the direct solubilization and nanoparticle incorporation of a viral membrane protein complex from the virus membrane. Our approach facilitated high-resolution structural studies of the bacterial peptide transporter PeptTSo2 by single-particle cryo-electron microscopy (cryo-EM) and allowed us to stabilize the HIV envelope glycoprotein in a functional state. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3302.map.gz emd_3302.map.gz | 45.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3302-v30.xml emd-3302-v30.xml emd-3302.xml emd-3302.xml | 9.1 KB 9.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-3302.png EMD-3302.png | 763.3 KB | ||
| その他 |  emd_3302_additional_1.map.gz emd_3302_additional_1.map.gz emd_3302_half_map_1.map.gz emd_3302_half_map_1.map.gz emd_3302_half_map_2.map.gz emd_3302_half_map_2.map.gz | 2.6 MB 45.8 MB 45.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3302 http://ftp.pdbj.org/pub/emdb/structures/EMD-3302 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3302 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3302 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3302_validation.pdf.gz emd_3302_validation.pdf.gz | 232.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3302_full_validation.pdf.gz emd_3302_full_validation.pdf.gz | 231.3 KB | 表示 | |
| XML形式データ |  emd_3302_validation.xml.gz emd_3302_validation.xml.gz | 6.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3302 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3302 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3302 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3302 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3302.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3302.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of Salipro-POT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.2156 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
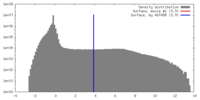
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-添付マップデータ: emd 3302 additional 1.map
| ファイル | emd_3302_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-添付マップデータ: emd 3302 half map 1.map
| ファイル | emd_3302_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-添付マップデータ: emd 3302 half map 2.map
| ファイル | emd_3302_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Salipro-POT
| 全体 | 名称: Salipro-POT |
|---|---|
| 要素 |
|
-超分子 #1000: Salipro-POT
| 超分子 | 名称: Salipro-POT / タイプ: sample / ID: 1000 / 集合状態: One tetramer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 224 MDa / 理論値: 250 MDa |
-分子 #1: Proton-coupled oligopeptide transporter
| 分子 | 名称: Proton-coupled oligopeptide transporter / タイプ: protein_or_peptide / ID: 1 / Name.synonym: PeptTSo2, / コピー数: 1 / 集合状態: Tetramer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Shewanella oneidensis (バクテリア) Shewanella oneidensis (バクテリア) |
| 分子量 | 実験値: 224 MDa / 理論値: 250 MDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: Proton:oligopeptide symporter POT family |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.7 mg/mL |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 120 K / 装置: FEI VITROBOT MARK I / 手法: Blot for 6 before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 日付 | 2014年9月15日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 実像数: 288 / 平均電子線量: 10 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 41132 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 0.0026 µm / 最小 デフォーカス(公称値): 0.0018 µm / 倍率(公称値): 31000 |
| 試料ステージ | 試料ホルダーモデル: OTHER |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Processed with Relion 1.3 |
|---|---|
| CTF補正 | 詳細: each particle |
| 最終 再構成 | 想定した対称性 - 点群: C4 (4回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 6.48 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Relion / 使用した粒子像数: 9913 |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)