+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Coxsackievirus B1 muture virion in complex with nAbs 8A10 and 5F5 (CVB1-M:8A10:5F5) | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Coxsackievirus B1 / Neutralizing antibody / Cryo-EM / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / viral capsid / host cell / nucleoside-triphosphate phosphatase ...symbiont-mediated suppression of host cytoplasmic pattern recognition receptor signaling pathway via inhibition of RIG-I activity / picornain 2A / symbiont-mediated suppression of host mRNA export from nucleus / symbiont genome entry into host cell via pore formation in plasma membrane / picornain 3C / T=pseudo3 icosahedral viral capsid / host cell cytoplasmic vesicle membrane / viral capsid / host cell / nucleoside-triphosphate phosphatase / channel activity / monoatomic ion transmembrane transport / DNA replication / RNA helicase activity / endocytosis involved in viral entry into host cell / symbiont-mediated suppression of host gene expression / symbiont-mediated activation of host autophagy / RNA-directed RNA polymerase / cysteine-type endopeptidase activity / viral RNA genome replication / RNA-directed RNA polymerase activity / DNA-templated transcription / symbiont entry into host cell / virion attachment to host cell / host cell nucleus / structural molecule activity / ATP hydrolysis activity / proteolysis / RNA binding / zinc ion binding / ATP binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Coxsackievirus B1 (コクサッキーウイルス) / Coxsackievirus B1 (コクサッキーウイルス) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.03 Å | |||||||||
 データ登録者 データ登録者 | Zheng Q / Zhu R / Sun H / Cheng T / Li S / Xia N | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Cell Host Microbe / 年: 2022 ジャーナル: Cell Host Microbe / 年: 2022タイトル: Structural basis for the synergistic neutralization of coxsackievirus B1 by a triple-antibody cocktail. 著者: Qingbing Zheng / Rui Zhu / Zhichao Yin / Longfa Xu / Hui Sun / Hai Yu / Yuanyuan Wu / Yichao Jiang / Qiongzi Huang / Yang Huang / Dongqing Zhang / Liqin Liu / Hongwei Yang / Maozhou He / ...著者: Qingbing Zheng / Rui Zhu / Zhichao Yin / Longfa Xu / Hui Sun / Hai Yu / Yuanyuan Wu / Yichao Jiang / Qiongzi Huang / Yang Huang / Dongqing Zhang / Liqin Liu / Hongwei Yang / Maozhou He / Zhenhong Zhou / Yanan Jiang / Zhenqin Chen / Huan Zhao / Yuqiong Que / Zhibo Kong / Lizhi Zhou / Tingting Li / Jun Zhang / Wenxin Luo / Ying Gu / Tong Cheng / Shaowei Li / Ningshao Xia /  要旨: Coxsackievirus B1 (CVB1) is an emerging pathogen associated with severe neonatal diseases including aseptic meningitis, myocarditis, and pancreatitis and also with the development of type 1 diabetes. ...Coxsackievirus B1 (CVB1) is an emerging pathogen associated with severe neonatal diseases including aseptic meningitis, myocarditis, and pancreatitis and also with the development of type 1 diabetes. We characterize the binding and therapeutic efficacies of three CVB1-specific neutralizing antibodies (nAbs) identified for their ability to inhibit host receptor engagement. High-resolution cryo-EM structures showed that these antibodies recognize different epitopes but with an overlapping region in the capsid VP2 protein and specifically the highly variable EF loop. Moreover, they perturb capsid-receptor interactions by binding various viral particle forms. Antibody combinations achieve synergetic neutralization via a stepwise capsid transition and virion disruption, indicating dynamic changes in the virion in response to multiple nAbs targeting the receptor-binding site. Furthermore, this three-antibody cocktail protects against lethal challenge in neonatal mice and limits pancreatitis and viral replication in a non-obese diabetic mouse model. These results illustrate the utility of nAbs for rational design of therapeutics against picornaviruses such as CVB. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
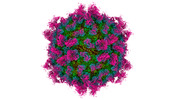
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_32983.map.gz emd_32983.map.gz | 631.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-32983-v30.xml emd-32983-v30.xml emd-32983.xml emd-32983.xml | 19.7 KB 19.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_32983.png emd_32983.png | 85.2 KB | ||
| Filedesc metadata |  emd-32983.cif.gz emd-32983.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-32983 http://ftp.pdbj.org/pub/emdb/structures/EMD-32983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32983 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-32983 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_32983_validation.pdf.gz emd_32983_validation.pdf.gz | 656.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_32983_full_validation.pdf.gz emd_32983_full_validation.pdf.gz | 656.3 KB | 表示 | |
| XML形式データ |  emd_32983_validation.xml.gz emd_32983_validation.xml.gz | 8.5 KB | 表示 | |
| CIF形式データ |  emd_32983_validation.cif.gz emd_32983_validation.cif.gz | 9.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32983 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32983 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32983 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-32983 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7x3cMC  7x2gC  7x2iC  7x2oC  7x2tC  7x2wC  7x35C  7x37C  7x38C  7x3dC  7x3eC  7x3fC  7x3yC  7x40C  7x42C  7x46C  7x47C  7x49C  7x4kC  7x4mC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_32983.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_32983.map.gz / 形式: CCP4 / 大きさ: 669.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.12 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Cryo-EM structure of Coxsackievirus B1 muture virion in complex w...
+超分子 #1: Cryo-EM structure of Coxsackievirus B1 muture virion in complex w...
+分子 #1: 8A10 light chain
+分子 #2: 8A10 heavy chain
+分子 #3: Virion protein 1
+分子 #4: VP2
+分子 #5: VP3
+分子 #6: Capsid protein VP4
+分子 #7: 5F5 light chain
+分子 #8: 5F5 heavy chain
+分子 #9: PALMITIC ACID
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F30 |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: INTEGRATING / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.4 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Tecnai F30 / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)





















