+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3242 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Dodecameric Chaetomium thermophilum Rvb1/Rvb2 complex in compact conformation | |||||||||
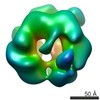
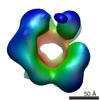
 マップデータ マップデータ | Reconstruction of full-length chaetomium thermophilum dodecameric Rvb1/Rvb2 complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | AAA+ proteins / cryo-electron microscopy / Rvb1 / Rvb2 / Chaetomium thermophilum | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ATP-dependent activity, acting on DNA / helicase activity / chromatin organization / DNA helicase / DNA repair / ATP hydrolysis activity / ATP binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Chaetomium thermophilum (菌類) Chaetomium thermophilum (菌類) | |||||||||
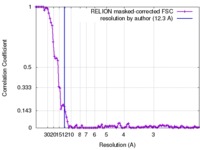
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 12.3 Å | |||||||||
 データ登録者 データ登録者 | Silva-Martin N / Dauden MI / Glatt S / Hoffmann NA / Kastritis P / Bork P / Beck M / Mueller CW | |||||||||
 引用 引用 |  ジャーナル: PLoS One / 年: 2016 ジャーナル: PLoS One / 年: 2016タイトル: The Combination of X-Ray Crystallography and Cryo-Electron Microscopy Provides Insight into the Overall Architecture of the Dodecameric Rvb1/Rvb2 Complex. 著者: Noella Silva-Martin / María I Daudén / Sebastian Glatt / Niklas A Hoffmann / Panagiotis Kastritis / Peer Bork / Martin Beck / Christoph W Müller /  要旨: The Rvb1/Rvb2 complex is an essential component of many cellular pathways. The Rvb1/Rvb2 complex forms a dodecameric assembly where six copies of each subunit form two heterohexameric rings. However, ...The Rvb1/Rvb2 complex is an essential component of many cellular pathways. The Rvb1/Rvb2 complex forms a dodecameric assembly where six copies of each subunit form two heterohexameric rings. However, due to conformational variability, the way the two rings pack together is still not fully understood. Here, we present the crystal structure and two cryo-electron microscopy reconstructions of the dodecameric, full-length Rvb1/Rvb2 complex, all showing that the interaction between the two heterohexameric rings is mediated through the Rvb1/Rvb2-specific domain II. Two conformations of the Rvb1/Rvb2 dodecamer are present in solution: a stretched conformation also present in the crystal, and a compact conformation. Novel asymmetric features observed in the reconstruction of the compact conformation provide additional insight into the plasticity of the Rvb1/Rvb2 complex. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3242.map.gz emd_3242.map.gz | 9.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3242-v30.xml emd-3242-v30.xml emd-3242.xml emd-3242.xml | 11.1 KB 11.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3242_fsc.xml emd_3242_fsc.xml | 10.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_3242.tif emd_3242.tif | 68.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3242 http://ftp.pdbj.org/pub/emdb/structures/EMD-3242 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3242 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3242 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3242_validation.pdf.gz emd_3242_validation.pdf.gz | 224.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3242_full_validation.pdf.gz emd_3242_full_validation.pdf.gz | 224 KB | 表示 | |
| XML形式データ |  emd_3242_validation.xml.gz emd_3242_validation.xml.gz | 11.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3242 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3242 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3242 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3242 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3242.map.gz / 形式: CCP4 / 大きさ: 96.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3242.map.gz / 形式: CCP4 / 大きさ: 96.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of full-length chaetomium thermophilum dodecameric Rvb1/Rvb2 complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.084 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Full-length Chaetomium thermophilum Rvb1/Rvb2 dodecameric complex...
| 全体 | 名称: Full-length Chaetomium thermophilum Rvb1/Rvb2 dodecameric complex in compact conformation |
|---|---|
| 要素 |
|
-超分子 #1000: Full-length Chaetomium thermophilum Rvb1/Rvb2 dodecameric complex...
| 超分子 | 名称: Full-length Chaetomium thermophilum Rvb1/Rvb2 dodecameric complex in compact conformation タイプ: sample / ID: 1000 集合状態: Dodecamer formed by two hetero-hexamers of Rvb1/Rvb2 Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 621 KDa |
-分子 #1: Rvb1
| 分子 | 名称: Rvb1 / タイプ: protein_or_peptide / ID: 1 / Name.synonym: RuvBL1, Pontin, TIP49a / コピー数: 6 / 集合状態: Double hetero-hexamer with Rvb2 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) / 別称: thermophilic filamentous fungus Chaetomium thermophilum (菌類) / 別称: thermophilic filamentous fungus |
| 分子量 | 理論値: 50.384 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: RuvB-like helicase InterPro: AAA+ ATPase domain, P-loop containing nucleoside triphosphate hydrolase, RuvB-like, TIP49, P-loop domain |
-分子 #2: Rvb2
| 分子 | 名称: Rvb2 / タイプ: protein_or_peptide / ID: 2 / Name.synonym: RuvBL2, Reptin, TIP49b / コピー数: 6 / 集合状態: Double hetero-hexamer with Rvb2 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Chaetomium thermophilum (菌類) / 別称: thermophilic filamentous fungus Chaetomium thermophilum (菌類) / 別称: thermophilic filamentous fungus |
| 分子量 | 理論値: 53.1458 KDa |
| 組換発現 | 生物種:  |
| 配列 | UniProtKB: RuvB-like helicase InterPro: AAA+ ATPase domain, P-loop containing nucleoside triphosphate hydrolase, RuvB-like, TIP49, P-loop domain |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.35 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Tris-HCl pH 7.5, 125 mM NaCl, 2 mM 2-mercaptoethanol and 5 mM MgCl2 |
| グリッド | 詳細: glow-discharged molybdenum grids 400 mesh, 1.2/1.5, Quantifoil |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 120 K / 装置: FEI VITROBOT MARK III 手法: 9 seconds blotting time, 15 seconds waiting time, blot offset -3 |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 日付 | 2014年11月3日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 1629 / 平均電子線量: 40 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 4.2 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 75000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)