+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-31420 | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Marburg virus nucleoprotein-RNA complex | |||||||||||||||||||||
 マップデータ マップデータ | ||||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
 キーワード キーワード | nucleoprotein / VIRAL PROTEIN | |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報viral RNA genome packaging / helical viral capsid / viral budding via host ESCRT complex / viral nucleocapsid / host cell cytoplasm / ribonucleoprotein complex / RNA binding 類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  Lake Victoria marburgvirus (strain Angola/2005) (ウイルス) / synthetic construct (人工物) Lake Victoria marburgvirus (strain Angola/2005) (ウイルス) / synthetic construct (人工物) | |||||||||||||||||||||
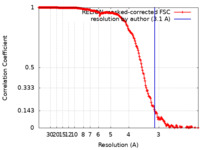
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Fujita FY / Sugita Y | |||||||||||||||||||||
| 資金援助 |  日本, 6件 日本, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Structural insight into Marburg virus nucleoprotein-RNA complex formation. 著者: Yoko Fujita-Fujiharu / Yukihiko Sugita / Yuki Takamatsu / Kazuya Houri / Manabu Igarashi / Yukiko Muramoto / Masahiro Nakano / Yugo Tsunoda / Ichiro Taniguchi / Stephan Becker / Takeshi Noda /   要旨: The nucleoprotein (NP) of Marburg virus (MARV), a close relative of Ebola virus (EBOV), encapsidates the single-stranded, negative-sense viral genomic RNA (vRNA) to form the helical NP-RNA complex. ...The nucleoprotein (NP) of Marburg virus (MARV), a close relative of Ebola virus (EBOV), encapsidates the single-stranded, negative-sense viral genomic RNA (vRNA) to form the helical NP-RNA complex. The NP-RNA complex constitutes the core structure for the assembly of the nucleocapsid that is responsible for viral RNA synthesis. Although appropriate interactions among NPs and RNA are required for the formation of nucleocapsid, the structural basis of the helical assembly remains largely elusive. Here, we show the structure of the MARV NP-RNA complex determined using cryo-electron microscopy at a resolution of 3.1 Å. The structures of the asymmetric unit, a complex of an NP and six RNA nucleotides, was very similar to that of EBOV, suggesting that both viruses share common mechanisms for the nucleocapsid formation. Structure-based mutational analysis of both MARV and EBOV NPs identified key residues for helical assembly and subsequent viral RNA synthesis. Importantly, most of the residues identified were conserved in both viruses. These findings provide a structural basis for understanding the nucleocapsid formation and contribute to the development of novel antivirals against MARV and EBOV. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_31420.map.gz emd_31420.map.gz | 405.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-31420-v30.xml emd-31420-v30.xml emd-31420.xml emd-31420.xml | 24.7 KB 24.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_31420_fsc.xml emd_31420_fsc.xml | 18.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_31420.png emd_31420.png | 137 KB | ||
| マスクデータ |  emd_31420_msk_1.map emd_31420_msk_1.map | 512 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-31420.cif.gz emd-31420.cif.gz | 6.5 KB | ||
| その他 |  emd_31420_additional_1.map.gz emd_31420_additional_1.map.gz emd_31420_half_map_1.map.gz emd_31420_half_map_1.map.gz emd_31420_half_map_2.map.gz emd_31420_half_map_2.map.gz | 281.9 MB 405 MB 404.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-31420 http://ftp.pdbj.org/pub/emdb/structures/EMD-31420 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31420 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-31420 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_31420_validation.pdf.gz emd_31420_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_31420_full_validation.pdf.gz emd_31420_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_31420_validation.xml.gz emd_31420_validation.xml.gz | 26.6 KB | 表示 | |
| CIF形式データ |  emd_31420_validation.cif.gz emd_31420_validation.cif.gz | 35.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31420 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31420 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31420 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-31420 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7f1mMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10733 (タイトル: Marburgvirus nucleoprotein RNA complex / Data size: 3.3 TB EMPIAR-10733 (タイトル: Marburgvirus nucleoprotein RNA complex / Data size: 3.3 TBData #1: 2469 Falcon3 movies in TIFF [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_31420.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_31420.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.13 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_31420_msk_1.map emd_31420_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: #2
| ファイル | emd_31420_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_31420_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_31420_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Marburgvirus nucleoprotein RNA complex
| 全体 | 名称: Marburgvirus nucleoprotein RNA complex |
|---|---|
| 要素 |
|
-超分子 #1: Marburgvirus nucleoprotein RNA complex
| 超分子 | 名称: Marburgvirus nucleoprotein RNA complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Nucleoprotein
| 分子 | 名称: Nucleoprotein / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Lake Victoria marburgvirus (strain Angola/2005) (ウイルス) Lake Victoria marburgvirus (strain Angola/2005) (ウイルス)株: Angola/2005 |
| 分子量 | 理論値: 44.165332 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MDLHSLLELG TKPTAPHVRN KKVILFDTNH QVSICNQIID AINSGIDLGD LLEGGLLTLC VEHYYNSDKD KFNTSPIAKY LRDAGYEFD VIKNADATRF LDVIPNEPHY SPLILALKTL ESTESQRGRI GLFLSFCSLF LPKLVVGDRA SIEKALRQVT V HQEQGIVT ...文字列: MDLHSLLELG TKPTAPHVRN KKVILFDTNH QVSICNQIID AINSGIDLGD LLEGGLLTLC VEHYYNSDKD KFNTSPIAKY LRDAGYEFD VIKNADATRF LDVIPNEPHY SPLILALKTL ESTESQRGRI GLFLSFCSLF LPKLVVGDRA SIEKALRQVT V HQEQGIVT YPNHWLTTGH MKVIFGILRS SFILKFVLIY QGVNLVTGHD AYDSIISNSV GQTRFSGLLI VKTVLEFILQ KT DSGVTLH PLVRTSKVKN EVASFKQALS NLARHGEYAP FARVLNLSGI NNLEHGLYPQ LSAIALGVAT AHGSTLAGVN VGE QYQQLR EAAYDAEVKL QRRHEHQEIQ AIAEDDEERK ILEQFHLQKT EITHSQTLAV LSQKREKLAR LAAEIENNI UniProtKB: Nucleoprotein |
-分子 #2: RNA (5'-R(P*UP*UP*UP*UP*UP*U)-3')
| 分子 | 名称: RNA (5'-R(P*UP*UP*UP*UP*UP*U)-3') / タイプ: rna / ID: 2 / コピー数: 2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 1.792037 KDa |
| 配列 | 文字列: UUUUUU |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 濃度 | 1.47 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 200 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV 詳細: Apply 1.25 micro littere of sample from each side of the grid and blot for 7 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 撮影したグリッド数: 1 / 実像数: 2469 / 平均電子線量: 30.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 0.5 µm / 倍率(補正後): 59000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 0.01 mm / 最大 デフォーカス(公称値): 2.25 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 59000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)