+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3070 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mammalian ribosome bound to the native Sec61 protein-conducting channel in the idle state | |||||||||
 マップデータ マップデータ | Subtomogram average of non-solubilized ribosome-Sec61 complexes in the idle state. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Ribosome / Sec61 / Translocon / Endoplasmic Reticulum / Cryoelectron Tomography / Subtomogram Analysis | |||||||||
| 生物種 |  | |||||||||
| 手法 | サブトモグラム平均法 / クライオ電子顕微鏡法 / 解像度: 9.4 Å | |||||||||
 データ登録者 データ登録者 | Pfeffer S / Burbaum L / Unverdorben P / Pech M / Chen Y / Zimmermann R / Beckmann R / Foerster F | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2015 ジャーナル: Nat Commun / 年: 2015タイトル: Structure of the native Sec61 protein-conducting channel. 著者: Stefan Pfeffer / Laura Burbaum / Pia Unverdorben / Markus Pech / Yuxiang Chen / Richard Zimmermann / Roland Beckmann / Friedrich Förster /  要旨: In mammalian cells, secretory and membrane proteins are translocated across or inserted into the endoplasmic reticulum (ER) membrane by the universally conserved protein-conducting channel Sec61, ...In mammalian cells, secretory and membrane proteins are translocated across or inserted into the endoplasmic reticulum (ER) membrane by the universally conserved protein-conducting channel Sec61, which has been structurally studied in isolated, detergent-solubilized states. Here we structurally and functionally characterize native, non-solubilized ribosome-Sec61 complexes on rough ER vesicles using cryo-electron tomography and ribosome profiling. Surprisingly, the 9-Å resolution subtomogram average reveals Sec61 in a laterally open conformation, even though the channel is not in the process of inserting membrane proteins into the lipid bilayer. In contrast to recent mechanistic models for polypeptide translocation and insertion, our results indicate that the laterally open conformation of Sec61 is the only conformation present in the ribosome-bound translocon complex, independent of its functional state. Consistent with earlier functional studies, our structure suggests that the ribosome alone, even without a nascent chain, is sufficient for lateral opening of Sec61 in a lipid environment. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3070.map.gz emd_3070.map.gz | 6.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3070-v30.xml emd-3070-v30.xml emd-3070.xml emd-3070.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-3070.tif EMD-3070.tif | 209.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3070 http://ftp.pdbj.org/pub/emdb/structures/EMD-3070 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3070 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3070 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3070_validation.pdf.gz emd_3070_validation.pdf.gz | 258.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3070_full_validation.pdf.gz emd_3070_full_validation.pdf.gz | 257.2 KB | 表示 | |
| XML形式データ |  emd_3070_validation.xml.gz emd_3070_validation.xml.gz | 6.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3070 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3070 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3070 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3070 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3070.map.gz / 形式: CCP4 / 大きさ: 39.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3070.map.gz / 形式: CCP4 / 大きさ: 39.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Subtomogram average of non-solubilized ribosome-Sec61 complexes in the idle state. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.62 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
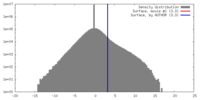
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Mammalian ribosome bound to the native protein translocon on cani...
| 全体 | 名称: Mammalian ribosome bound to the native protein translocon on canine pancreatic ER vesicles |
|---|---|
| 要素 |
|
-超分子 #1000: Mammalian ribosome bound to the native protein translocon on cani...
| 超分子 | 名称: Mammalian ribosome bound to the native protein translocon on canine pancreatic ER vesicles タイプ: sample / ID: 1000 / Number unique components: 2 |
|---|
-超分子 #1: Membrane-bound 80S ribosome
| 超分子 | 名称: Membrane-bound 80S ribosome / タイプ: complex / ID: 1 / 組換発現: No / Ribosome-details: ribosome-eukaryote: ALL |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: ER protein translocon
| 分子 | 名称: ER protein translocon / タイプ: protein_or_peptide / ID: 1 / 組換発現: No |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.6 / 詳細: 20mM Hepes, 50mM KCl; 2mM MgCl2 |
| グリッド | 詳細: Lacey carbon molybdenum grid |
| 凍結 | 凍結剤: ETHANE-PROPANE MIXTURE / チャンバー内湿度: 70 % / 装置: FEI VITROBOT MARK IV / 手法: Blot 3 seconds before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: Gatan |
| 日付 | 2014年6月18日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / 平均電子線量: 30 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.0 µm / 最小 デフォーカス(公称値): 3.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER Tilt series - Axis1 - Min angle: -20 ° / Tilt series - Axis1 - Max angle: 20 ° |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Tomogram reconstruction and template matching against a single particle cryo-EM reconstruction of the 80S ribosome were accomplished using PyTom. Subtomograms extracted from cross correlation peaks in the tomogram were classified using constrained principal component analysis focusing on the large ribosomal subunit and the ER membrane. For the retained coordinates, 1 x binned subtomograms were reconstructed individually from the weighted back-projections using the full tilt range, iteratively aligned and classified in two consecutive steps focusing (I) on the translocon and (II) on the ribosomal tRNA binding sites. For the retained coordinates, unbinned subtomograms were reconstructed individually from the weighted back-projections using only a reduced tilt range (-20 deg to +20 deg) and iteratively aligned using a 'conventional' subtomogram alignment procedure. |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 9.4 Å / 解像度の算出法: OTHER ソフトウェア - 名称: PyTom, tom_toolbox, av3_toolbox 使用したサブトモグラム数: 12528 |
| CTF補正 | 詳細: Each tilt image |
 ムービー
ムービー コントローラー
コントローラー












 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)