+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-3014 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Electron cryo-microscopy of Cowpea Mosaic Virus (CPMV) empty virus like particle (eVLP) | |||||||||
 マップデータ マップデータ | Cowpea Mosaic virus empty virus like particles (CPMV eVLPs) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | CPMV / eVLP / virus / comoviridae / picornavirales. | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報transport of virus in host, cell to cell / host cell plasmodesma / T=3 icosahedral viral capsid / symbiont-mediated suppression of host innate immune response / GTP binding / host cell nucleus / structural molecule activity / DNA binding / RNA binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   Cowpea mosaic virus (ササゲモザイクウイルス) Cowpea mosaic virus (ササゲモザイクウイルス) | |||||||||
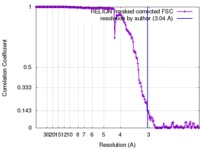
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.04 Å | |||||||||
 データ登録者 データ登録者 | Hesketh EL / Meshcheriakova Y / Dent KC / Saxena P / Thompson R / Reddy V / Cockburn JJ / Lomonossoff GP / Ranson NA | |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2015 ジャーナル: Nat Commun / 年: 2015タイトル: Mechanisms of assembly and genome packaging in an RNA virus revealed by high-resolution cryo-EM. 著者: Emma L Hesketh / Yulia Meshcheriakova / Kyle C Dent / Pooja Saxena / Rebecca F Thompson / Joseph J Cockburn / George P Lomonossoff / Neil A Ranson /  要旨: Cowpea mosaic virus is a plant-infecting member of the Picornavirales and is of major interest in the development of biotechnology applications. Despite the availability of >100 crystal structures of ...Cowpea mosaic virus is a plant-infecting member of the Picornavirales and is of major interest in the development of biotechnology applications. Despite the availability of >100 crystal structures of Picornavirales capsids, relatively little is known about the mechanisms of capsid assembly and genome encapsidation. Here we have determined cryo-electron microscopy reconstructions for the wild-type virus and an empty virus-like particle, to 3.4 Å and 3.0 Å resolution, respectively, and built de novo atomic models of their capsids. These new structures reveal the C-terminal region of the small coat protein subunit, which is essential for virus assembly and which was missing from previously determined crystal structures, as well as residues that bind to the viral genome. These observations allow us to develop a new model for genome encapsidation and capsid assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_3014.map.gz emd_3014.map.gz | 43.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-3014-v30.xml emd-3014-v30.xml emd-3014.xml emd-3014.xml | 9.7 KB 9.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_3014_fsc.xml emd_3014_fsc.xml | 17.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  radial.tif radial.tif | 382 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-3014 http://ftp.pdbj.org/pub/emdb/structures/EMD-3014 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3014 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-3014 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_3014_validation.pdf.gz emd_3014_validation.pdf.gz | 311.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_3014_full_validation.pdf.gz emd_3014_full_validation.pdf.gz | 310.8 KB | 表示 | |
| XML形式データ |  emd_3014_validation.xml.gz emd_3014_validation.xml.gz | 14.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3014 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3014 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3014 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-3014 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_3014.map.gz / 形式: CCP4 / 大きさ: 210.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_3014.map.gz / 形式: CCP4 / 大きさ: 210.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cowpea Mosaic virus empty virus like particles (CPMV eVLPs) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.04 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Cowpea mosaic virus empty virus like particle (CPMV eVLP)
| 全体 | 名称: Cowpea mosaic virus empty virus like particle (CPMV eVLP) |
|---|---|
| 要素 |
|
-超分子 #1000: Cowpea mosaic virus empty virus like particle (CPMV eVLP)
| 超分子 | 名称: Cowpea mosaic virus empty virus like particle (CPMV eVLP) タイプ: sample / ID: 1000 / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 3.87 MDa |
-超分子 #1: Cowpea mosaic virus
| 超分子 | 名称: Cowpea mosaic virus / タイプ: virus / ID: 1 / Name.synonym: CPMV 詳細: Empty virus like particles (eVLPs) produced by expressing a coat precursor protein VP60 which expressed both large and small proteins and the 24K protease in N. benthamiana NCBI-ID: 12264 / 生物種: Cowpea mosaic virus / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: Yes / Syn species name: CPMV |
|---|---|
| 宿主 | 生物種:  |
| 分子量 | 理論値: 3.87 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 280 Å / T番号(三角分割数): 3 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.2 mg/mL |
|---|---|
| グリッド | 詳細: 200 mesh quantifoil grids with 2 um holes, glow discharged |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 94 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 90 K / 最高: 90 K |
| 日付 | 2014年11月1日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 1135 / 平均電子線量: 45 e/Å2 詳細: Images are averages of 16 images recorded on a Falcon II direct detector |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 134615 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 134615 |
| 試料ステージ | 試料ホルダー: Liquid nitrogen cooled 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)