+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
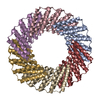
| タイトル | C4HR1_8r_shift5: Extendable repeat protein fiber | |||||||||
 マップデータ マップデータ | Sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Oligomer / repeat protein / DE NOVO PROTEIN | |||||||||
| 生物種 | unidentified (未定義) | |||||||||
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 7.62 Å | |||||||||
 データ登録者 データ登録者 | Bethel NP / Borst AJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Chem / 年: 2023 ジャーナル: Nat Chem / 年: 2023タイトル: Precisely patterned nanofibres made from extendable protein multiplexes. 著者: Neville P Bethel / Andrew J Borst / Fabio Parmeggiani / Matthew J Bick / T J Brunette / Hannah Nguyen / Alex Kang / Asim K Bera / Lauren Carter / Marcos C Miranda / Ryan D Kibler / Mila Lamb ...著者: Neville P Bethel / Andrew J Borst / Fabio Parmeggiani / Matthew J Bick / T J Brunette / Hannah Nguyen / Alex Kang / Asim K Bera / Lauren Carter / Marcos C Miranda / Ryan D Kibler / Mila Lamb / Xinting Li / Banumathi Sankaran / David Baker /   要旨: Molecular systems with coincident cyclic and superhelical symmetry axes have considerable advantages for materials design as they can be readily lengthened or shortened by changing the length of the ...Molecular systems with coincident cyclic and superhelical symmetry axes have considerable advantages for materials design as they can be readily lengthened or shortened by changing the length of the constituent monomers. Among proteins, alpha-helical coiled coils have such symmetric, extendable architectures, but are limited by the relatively fixed geometry and flexibility of the helical protomers. Here we describe a systematic approach to generating modular and rigid repeat protein oligomers with coincident C to C and superhelical symmetry axes that can be readily extended by repeat propagation. From these building blocks, we demonstrate that a wide range of unbounded fibres can be systematically designed by introducing hydrophilic surface patches that force staggering of the monomers; the geometry of such fibres can be precisely tuned by varying the number of repeat units in the monomer and the placement of the hydrophilic patches. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_29851.map.gz emd_29851.map.gz | 10.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-29851-v30.xml emd-29851-v30.xml emd-29851.xml emd-29851.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_29851_fsc.xml emd_29851_fsc.xml | 8.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_29851.png emd_29851.png | 56.5 KB | ||
| Filedesc metadata |  emd-29851.cif.gz emd-29851.cif.gz | 4.8 KB | ||
| その他 |  emd_29851_additional_1.map.gz emd_29851_additional_1.map.gz emd_29851_half_map_1.map.gz emd_29851_half_map_1.map.gz emd_29851_half_map_2.map.gz emd_29851_half_map_2.map.gz | 10.6 MB 59.2 MB 59.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-29851 http://ftp.pdbj.org/pub/emdb/structures/EMD-29851 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29851 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-29851 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_29851_validation.pdf.gz emd_29851_validation.pdf.gz | 1.1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_29851_full_validation.pdf.gz emd_29851_full_validation.pdf.gz | 1.1 MB | 表示 | |
| XML形式データ |  emd_29851_validation.xml.gz emd_29851_validation.xml.gz | 16.6 KB | 表示 | |
| CIF形式データ |  emd_29851_validation.cif.gz emd_29851_validation.cif.gz | 21.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29851 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29851 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29851 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-29851 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_29851.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_29851.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.885 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Unsharpened map
| ファイル | emd_29851_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_29851_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_29851_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : C4HR1_8r_shift5
| 全体 | 名称: C4HR1_8r_shift5 |
|---|---|
| 要素 |
|
-超分子 #1: C4HR1_8r_shift5
| 超分子 | 名称: C4HR1_8r_shift5 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 分子量 | 理論値: 42.54 KDa |
-分子 #1: C4HR1_8r_shift5
| 分子 | 名称: C4HR1_8r_shift5 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: unidentified (未定義) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MMRDELIRWH LMHPMEQLRF LRELVENPER FREFWRRLEE EPGAVELFLE NIHLLHPIVQ IYLLELLVEN PELFRLFFEV LERCPGAVEL FLENWRLLPP IVQRIFLRLL VENPELFRLF FEVLERCPGA VKLFLENIKN LPPLIQKYLL RLLVENPELF RLFFEMLERC ...文字列: MMRDELIRWH LMHPMEQLRF LRELVENPER FREFWRRLEE EPGAVELFLE NIHLLHPIVQ IYLLELLVEN PELFRLFFEV LERCPGAVEL FLENWRLLPP IVQRIFLRLL VENPELFRLF FEVLERCPGA VKLFLENIKN LPPLIQKYLL RLLVENPELF RLFFEMLERC PGAVELFLEI IHELPPEIQK YFLELLVENP ELFRLFFEVL ERCPGAVKLF LNNIYLLHPL VQIHLLRLLV ENPELFRLFF EMLERCPGAV EQFLINLYLL HPIVQLVFLE LLVENPELFR LFFEVLKRCP GALELFKFII ELLPPIVQRH FKRLLEENPE LKELVKEVEE EGSLEHHHHH H |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | helical array |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS GLACIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.8 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)