+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2888 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-Molecular electron tomography of human non-immune soluble pentameric IgM | |||||||||
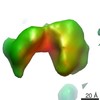
 マップデータ マップデータ | Reconstruction of human non-iimune pentameric IgM | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Malaria / Rosetting / IgM | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 電子線トモグラフィー法 / クライオ電子顕微鏡法 / 解像度: 20.0 Å | |||||||||
 データ登録者 データ登録者 | Akhouri RR / Goel S / Furusho H / Skoglund U / Wahlgren M | |||||||||
 引用 引用 |  ジャーナル: Cell Rep / 年: 2016 ジャーナル: Cell Rep / 年: 2016タイトル: Architecture of Human IgM in Complex with P. falciparum Erythrocyte Membrane Protein 1. 著者: Reetesh Raj Akhouri / Suchi Goel / Hirotoshi Furusho / Ulf Skoglund / Mats Wahlgren /   要旨: Plasmodium falciparum virulence is associated with sequestration of infected erythrocytes. Microvascular binding mediated by PfEMP1 in complex with non-immune immunoglobulin M (IgM) is common among ...Plasmodium falciparum virulence is associated with sequestration of infected erythrocytes. Microvascular binding mediated by PfEMP1 in complex with non-immune immunoglobulin M (IgM) is common among parasites that cause both severe childhood malaria and pregnancy-associated malaria. Here, we present cryo-molecular electron tomography structures of human IgM, PfEMP1 and their complex. Three-dimensional reconstructions of IgM reveal that it has a dome-like core, randomly oriented Fab2s units, and the overall shape of a turtle. PfEMP1 is a C- shaped molecule with a flexible N terminus followed by an arc-shaped backbone and a bulky C terminus that interacts with IgM. Our data demonstrate that the PfEMP1 binding pockets on IgM overlap with those of C1q, and the bulkiness of PfEMP1 limits the capacity of IgM to interact with PfEMP1. We suggest that P. falciparum exploits IgM to cluster PfEMP1 into an organized matrix to augment its affinity to host cell receptors. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2888.map.gz emd_2888.map.gz | 1.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2888-v30.xml emd-2888-v30.xml emd-2888.xml emd-2888.xml | 9.8 KB 9.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2888.tif emd_2888.tif | 83.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2888 http://ftp.pdbj.org/pub/emdb/structures/EMD-2888 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2888 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2888 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2888_validation.pdf.gz emd_2888_validation.pdf.gz | 162.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2888_full_validation.pdf.gz emd_2888_full_validation.pdf.gz | 161.5 KB | 表示 | |
| XML形式データ |  emd_2888_validation.xml.gz emd_2888_validation.xml.gz | 4.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2888 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2888 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2888 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2888 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2888.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2888.map.gz / 形式: CCP4 / 大きさ: 1.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of human non-iimune pentameric IgM | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 これらの図は立方格子座標系で作成されたものです | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.534 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
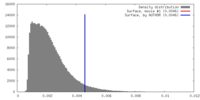
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Human non-immune pentameric IgM
| 全体 | 名称: Human non-immune pentameric IgM |
|---|---|
| 要素 |
|
-超分子 #1000: Human non-immune pentameric IgM
| 超分子 | 名称: Human non-immune pentameric IgM / タイプ: sample / ID: 1000 詳細: the sample was polydisperse using Dynamic Light Scattering. 集合状態: Monomer / Number unique components: 1 |
|---|---|
| 分子量 | 実験値: 1.047 MDa / 理論値: 1.0 MDa |
-分子 #1: Immunoglobulin M
| 分子 | 名称: Immunoglobulin M / タイプ: protein_or_peptide / ID: 1 / Name.synonym: IgM / コピー数: 1 / 集合状態: monomer / 組換発現: No / データベース: NCBI |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Soluble in serum Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: Soluble in serum |
| 分子量 | 実験値: 1.047 MDa / 理論値: 1.0 MDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 電子線トモグラフィー法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 20mM Tris, 200mM NaCl |
| グリッド | 詳細: C-Flat (Copper grid with thin Carbon support) |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 78 K / 装置: FEI VITROBOT MARK IV / 手法: Blot for 4 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 温度 | 最低: 78 K / 最高: 100 K |
| 特殊光学系 | エネルギーフィルター - 名称: FEI |
| 日付 | 2013年10月24日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 実像数: 281 / 平均電子線量: 40 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 0.0015 µm / 最小 デフォーカス(公称値): 0.0007 µm / 倍率(公称値): 37000 |
| 試料ステージ | 試料ホルダー: LN2 cooled 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER Tilt series - Axis1 - Min angle: -70 ° / Tilt series - Axis1 - Max angle: 70 ° / Tilt series - Axis1 - Angle increment: 0.5 ° |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | CTF correction on each tilt |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 20.0 Å / ソフトウェア - 名称: COMET / 使用した粒子像数: 281 |
| CTF補正 | 詳細: each frame |
 ムービー
ムービー コントローラー
コントローラー




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)