+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of human ABCA7 in BPL/Ch Nanodiscs | |||||||||
 マップデータ マップデータ | Cryo-EM Structure of human ABCA7 in Brain Polar Lipid/Cholesterol nanodiscs - Final Post processed map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ABC transporter / Lipid Transporter / ABC exporter / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報plasma membrane raft organization / apolipoprotein A-I receptor activity / positive regulation of engulfment of apoptotic cell / phospholipid transporter activity / negative regulation of amyloid precursor protein biosynthetic process / ABC transporters in lipid homeostasis / phosphatidylserine floppase activity / floppase activity / amyloid-beta clearance by cellular catabolic process / positive regulation of phospholipid efflux ...plasma membrane raft organization / apolipoprotein A-I receptor activity / positive regulation of engulfment of apoptotic cell / phospholipid transporter activity / negative regulation of amyloid precursor protein biosynthetic process / ABC transporters in lipid homeostasis / phosphatidylserine floppase activity / floppase activity / amyloid-beta clearance by cellular catabolic process / positive regulation of phospholipid efflux / phospholipid efflux / phosphatidylcholine floppase activity / positive regulation of amyloid-beta clearance / negative regulation of endocytosis / high-density lipoprotein particle assembly / positive regulation of protein localization to cell surface / P-type phospholipid transporter / regulation of amyloid precursor protein catabolic process / apolipoprotein A-I-mediated signaling pathway / cholesterol efflux / phospholipid translocation / amyloid-beta formation / negative regulation of amyloid-beta formation / negative regulation of PERK-mediated unfolded protein response / regulation of lipid metabolic process / glial cell projection / positive regulation of cholesterol efflux / ATPase-coupled transmembrane transporter activity / protein localization to nucleus / phagocytosis / ABC-type transporter activity / phagocytic cup / negative regulation of MAPK cascade / positive regulation of phagocytosis / visual learning / memory / ruffle membrane / cell junction / early endosome membrane / positive regulation of ERK1 and ERK2 cascade / Golgi membrane / intracellular membrane-bounded organelle / cell surface / endoplasmic reticulum / Golgi apparatus / ATP hydrolysis activity / ATP binding / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Alam A / Le LTM / Thompson JR | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: EMBO J / 年: 2023 ジャーナル: EMBO J / 年: 2023タイトル: Cryo-EM structures of human ABCA7 provide insights into its phospholipid translocation mechanisms. 著者: Le Thi My Le / James Robert Thompson / Sepehr Dehghani-Ghahnaviyeh / Shashank Pant / Phuoc Xuan Dang / Jarrod Bradley French / Takahisa Kanikeyo / Emad Tajkhorshid / Amer Alam /  要旨: Phospholipid extrusion by ABC subfamily A (ABCA) exporters is central to cellular physiology, although the specifics of the underlying substrate interactions and transport mechanisms remain poorly ...Phospholipid extrusion by ABC subfamily A (ABCA) exporters is central to cellular physiology, although the specifics of the underlying substrate interactions and transport mechanisms remain poorly resolved at the molecular level. Here we report cryo-EM structures of lipid-embedded human ABCA7 in an open state and in a nucleotide-bound, closed state at resolutions between 3.6 and 4.0 Å. The former reveals an ordered patch of bilayer lipids traversing the transmembrane domain (TMD), while the latter reveals a lipid-free, closed TMD with a small extracellular opening. These structures offer a structural framework for both substrate entry and exit from the ABCA7 TMD and highlight conserved rigid-body motions that underlie the associated conformational transitions. Combined with functional analysis and molecular dynamics (MD) simulations, our data also shed light on lipid partitioning into the ABCA7 TMD and localized membrane perturbations that underlie ABCA7 function and have broader implications for other ABCA family transporters. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28041.map.gz emd_28041.map.gz | 202.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28041-v30.xml emd-28041-v30.xml emd-28041.xml emd-28041.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_28041.png emd_28041.png | 105.8 KB | ||
| Filedesc metadata |  emd-28041.cif.gz emd-28041.cif.gz | 7.6 KB | ||
| その他 |  emd_28041_additional_1.map.gz emd_28041_additional_1.map.gz emd_28041_half_map_1.map.gz emd_28041_half_map_1.map.gz emd_28041_half_map_2.map.gz emd_28041_half_map_2.map.gz | 129 MB 171.3 MB 171.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28041 http://ftp.pdbj.org/pub/emdb/structures/EMD-28041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28041 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28041 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_28041_validation.pdf.gz emd_28041_validation.pdf.gz | 918 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_28041_full_validation.pdf.gz emd_28041_full_validation.pdf.gz | 917.6 KB | 表示 | |
| XML形式データ |  emd_28041_validation.xml.gz emd_28041_validation.xml.gz | 15.4 KB | 表示 | |
| CIF形式データ |  emd_28041_validation.cif.gz emd_28041_validation.cif.gz | 18.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28041 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28041 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28041 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-28041 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28041.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28041.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Cryo-EM Structure of human ABCA7 in Brain Polar Lipid/Cholesterol nanodiscs - Final Post processed map | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.895 Å | ||||||||||||||||||||||||||||||||||||
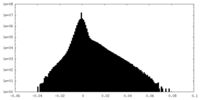
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: Local resolution filtered map
| ファイル | emd_28041_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution filtered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
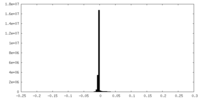
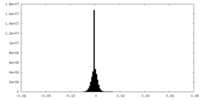
| 密度ヒストグラム |
-ハーフマップ: Refined Map half 1
| ファイル | emd_28041_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refined Map half 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
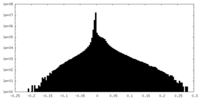
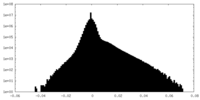
| 密度ヒストグラム |
-ハーフマップ: Refined Map half 2
| ファイル | emd_28041_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Refined Map half 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
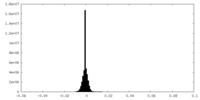
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human ABCA7 in Porcine BPL/Ch/MSP1D1 nanodiscs
| 全体 | 名称: Human ABCA7 in Porcine BPL/Ch/MSP1D1 nanodiscs |
|---|---|
| 要素 |
|
-超分子 #1: Human ABCA7 in Porcine BPL/Ch/MSP1D1 nanodiscs
| 超分子 | 名称: Human ABCA7 in Porcine BPL/Ch/MSP1D1 nanodiscs / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: Human ABCA7 recombinantly expressed in HEK293 TREX cell line and reconstituted in MSP1D1 nanodiscs comprising 4:1 mixture of Porcine Brain Polar Lipids:Cholesterol |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 234.35 KDa |
-分子 #1: Phospholipid-transporting ATPase ABCA7
| 分子 | 名称: Phospholipid-transporting ATPase ABCA7 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO / EC番号: P-type phospholipid transporter |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 234.598578 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAFWTQLMLL LWKNFMYRRR QPVQLLVELL WPLFLFFILV AVRHSHPPLE HHECHFPNKP LPSAGTVPWL QGLICNVNNT CFPQLTPGE EPGRLSNFND SLVSRLLADA RTVLGGASAH RTLAGLGKLI ATLRAARSTA QPQPTKQSPL EPPMLDVAEL L TSLLRTES ...文字列: MAFWTQLMLL LWKNFMYRRR QPVQLLVELL WPLFLFFILV AVRHSHPPLE HHECHFPNKP LPSAGTVPWL QGLICNVNNT CFPQLTPGE EPGRLSNFND SLVSRLLADA RTVLGGASAH RTLAGLGKLI ATLRAARSTA QPQPTKQSPL EPPMLDVAEL L TSLLRTES LGLALGQAQE PLHSLLEAAE DLAQELLALR SLVELRALLQ RPRGTSGPLE LLSEALCSVR GPSSTVGPSL NW YEASDLM ELVGQEPESA LPDSSLSPAC SELIGALDSH PLSRLLWRRL KPLILGKLLF APDTPFTRKL MAQVNRTFEE LTL LRDVRE VWEMLGPRIF TFMNDSSNVA MLQRLLQMQD EGRRQPRPGG RDHMEALRSF LDPGSGGYSW QDAHADVGHL VGTL GRVTE CLSLDKLEAA PSEAALVSRA LQLLAEHRFW AGVVFLGPED SSDPTEHPTP DLGPGHVRIK IRMDIDVVTR TNKIR DRFW DPGPAADPLT DLRYVWGGFV YLQDLVERAA VRVLSGANPR AGLYLQQMPY PCYVDDVFLR VLSRSLPLFL TLAWIY SVT LTVKAVVREK ETRLRDTMRA MGLSRAVLWL GWFLSCLGPF LLSAALLVLV LKLGDILPYS HPGVVFLFLA AFAVATV TQ SFLLSAFFSR ANLAAACGGL AYFSLYLPYV LCVAWRDRLP AGGRVAASLL SPVAFGFGCE SLALLEEQGE GAQWHNVG T RPTADVFSLA QVSGLLLLDA ALYGLATWYL EAVCPGQYGI PEPWNFPFRR SYWCGPRPPK SPAPCPTPLD PKVLVEEAP PGLSPGVSVR SLEKRFPGSP QPALRGLSLD FYQGHITAFL GHNGAGKTTT LSILSGLFPP SGGSAFILGH DVRSSMAAIR PHLGVCPQY NVLFDMLTVD EHVWFYGRLK GLSAAVVGPE QDRLLQDVGL VSKQSVQTRH LSGGMQRKLS VAIAFVGGSQ V VILDEPTA GVDPASRRGI WELLLKYREG RTLILSTHHL DEAELLGDRV AVVAGGRLCC CGSPLFLRRH LGSGYYLTLV KA RLPLTTN EKADTDMEGS VDTRQEKKNG SQGSRVGTPQ LLALVQHWVP GARLVEELPH ELVLVLPYTG AHDGSFATLF REL DTRLAE LRLTGYGISD TSLEEIFLKV VEECAADTDM EDGSCGQHLC TGIAGLDVTL RLKMPPQETA LENGEPAGSA PETD QGSGP DAVGRVQGWA LTRQQLQALL LKRFLLARRS RRGLFAQIVL PALFVGLALV FSLIVPPFGH YPALRLSPTM YGAQV SFFS EDAPGDPGRA RLLEALLQEA GLEEPPVQHS SHRFSAPEVP AEVAKVLASG NWTPESPSPA CQCSRPGARR LLPDCP AAA GGPPPPQAVT GSGEVVQNLT GRNLSDFLVK TYPRLVRQGL KTKKWVNEVR YGGFSLGGRD PGLPSGQELG RSVEELW AL LSPLPGGALD RVLKNLTAWA HSLDAQDSLK IWFNNKGWHS MVAFVNRASN AILRAHLPPG PARHAHSITT LNHPLNLT K EQLSEGALMA SSVDVLVSIC VVFAMSFVPA SFTLVLIEER VTRAKHLQLM GGLSPTLYWL GNFLWDMCNY LVPACIVVL IFLAFQQRAY VAPANLPALL LLLLLYGWSI TPLMYPASFF FSVPSTAYVV LTCINLFIGI NGSMATFVLE LFSDQKLQEV SRILKQVFL IFPHFCLGRG LIDMVRNQAM ADAFERLGDR QFQSPLRWEV VGKNLLAMVI QGPLFLLFTL LLQHRSQLLP Q PRVRSLPL LGEEDEDVAR ERERVVQGAT QGDVLVLRNL TKVYRGQRMP AVDRLCLGIP PGECFGLLGV NGAGKTSTFR MV TGDTLAS RGEAVLAGHS VAREPSAAHL SMGYCPQSDA IFELLTGREH LELLARLRGV PEAQVAQTAG SGLARLGLSW YAD RPAGTY SGGNKRKLAT ALALVGDPAV VFLDEPTTGM DPSARRFLWN SLLAVVREGR SVMLTSHSME ECEALCSRLA IMVN GRFRC LGSPQHLKGR FAAGHTLTLR VPAARSQPAA AFVAAEFPGA ELREAHGGRL RFQLPPGGRC ALARVFGELA VHGAE HGVE DFSVSQTMLE EVFLYFSKDQ GKDEDTEEQK EAGVGVDPAP GLQHPKRVSQ FLDDPSTAET VL UniProtKB: Phospholipid-transporting ATPase ABCA7 |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 4 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #4: UNKNOWN LIGAND
| 分子 | 名称: UNKNOWN LIGAND / タイプ: ligand / ID: 4 / コピー数: 18 / 式: UNL |
|---|---|
| 分子量 | 理論値: 622.834 Da |
| Chemical component information | 
ChemComp-UNL: |
-分子 #5: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 5 / コピー数: 2 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information |  ChemComp-AGS: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 25mM HEPES pH 7.5, 150mM NaCl, 5mM ATP gammaS, 10mM Magnesium Chloride |
| グリッド | モデル: Quantifoil R1.2/1.3 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均露光時間: 60.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.4 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)