+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2791 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Near-atomic resolution reconstruction of Nudaurelia capensis omega virus (NwV) using a mid-range electron microscope operated at 200 kV | |||||||||
 マップデータ マップデータ | Reconstruction of Nudaurelia capensis omega virus (NwV). Binned in Fourier space from an original pixel size of 1.25 | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Single-particle electron microscopy / Direct detectors / Near-atomic resolution | |||||||||
| 生物種 |   Nudaurelia capensis omega virus (ウイルス) Nudaurelia capensis omega virus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Campbell MG / Kearney BM / Cheng A / Potter CS / Johnson JE / Carragher B / Veesler D | |||||||||
 引用 引用 |  ジャーナル: J Struct Biol / 年: 2014 ジャーナル: J Struct Biol / 年: 2014タイトル: Near-atomic resolution reconstructions using a mid-range electron microscope operated at 200 kV. 要旨: A new era has begun for single particle cryo-electron microscopy (cryoEM) which can now compete with X-ray crystallography for determination of protein structures. The development of direct detectors ...A new era has begun for single particle cryo-electron microscopy (cryoEM) which can now compete with X-ray crystallography for determination of protein structures. The development of direct detectors constitutes a revolution that has led to a wave of near-atomic resolution cryoEM reconstructions. However, regardless of the sample studied, virtually all high-resolution reconstructions reported to date have been achieved using high-end microscopes. We demonstrate that the new generation of direct detectors coupled to a widely used mid-range electron microscope also enables obtaining cryoEM maps of sufficient quality for de novo modeling of protein structures of different sizes and symmetries. We provide an outline of the strategy used to achieve a 3.7 Å resolution reconstruction of Nudaurelia capensis ω virus and a 4.2 Å resolution reconstruction of the Thermoplasma acidophilum T20S proteasome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2791.map.gz emd_2791.map.gz | 51.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2791-v30.xml emd-2791-v30.xml emd-2791.xml emd-2791.xml | 10 KB 10 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2791.png emd_2791.png | 2.1 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2791 http://ftp.pdbj.org/pub/emdb/structures/EMD-2791 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2791 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2791 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2791_validation.pdf.gz emd_2791_validation.pdf.gz | 232.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2791_full_validation.pdf.gz emd_2791_full_validation.pdf.gz | 231.7 KB | 表示 | |
| XML形式データ |  emd_2791_validation.xml.gz emd_2791_validation.xml.gz | 8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2791 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2791 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2791 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2791 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2791.map.gz / 形式: CCP4 / 大きさ: 500 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2791.map.gz / 形式: CCP4 / 大きさ: 500 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Reconstruction of Nudaurelia capensis omega virus (NwV). Binned in Fourier space from an original pixel size of 1.25 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.56 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
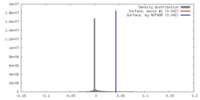
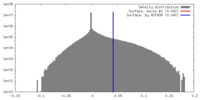
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Near-atomic resolution reconstruction of NwV using a mid-range el...
| 全体 | 名称: Near-atomic resolution reconstruction of NwV using a mid-range electron microscope operated at 200 kV |
|---|---|
| 要素 |
|
-超分子 #1000: Near-atomic resolution reconstruction of NwV using a mid-range el...
| 超分子 | 名称: Near-atomic resolution reconstruction of NwV using a mid-range electron microscope operated at 200 kV タイプ: sample / ID: 1000 / 集合状態: Icosahedral / Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 16.75 MDa |
-超分子 #1: Nudaurelia capensis omega virus
| 超分子 | 名称: Nudaurelia capensis omega virus / タイプ: virus / ID: 1 / Name.synonym: NwV / NCBI-ID: 12541 / 生物種: Nudaurelia capensis omega virus / ウイルスタイプ: VIRUS-LIKE PARTICLE / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No / Syn species name: NwV |
|---|---|
| 宿主 | 生物種:  Lepidoptera (昆虫) / 別称: INVERTEBRATES Lepidoptera (昆虫) / 別称: INVERTEBRATES |
| Host system | 生物種:  組換細胞: SF21 / 組換プラスミド: baculovirus |
| 分子量 | 理論値: 16.75 MDa |
| ウイルス殻 | Shell ID: 1 / 直径: 410 Å / T番号(三角分割数): 4 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 60 mg/mL |
|---|---|
| 緩衝液 | pH: 8 / 詳細: 250 mM NaCl, 50 mM Tris |
| グリッド | 詳細: 1.2/1.3 C-Flat grid, plasma cleaned |
| 凍結 | 凍結剤: ETHANE / チャンバー内温度: 95 K / 装置: GATAN CRYOPLUNGE 3 / 詳細: Vitrification carried out at room temperature / 手法: Blot for 3 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 29,000 times magnification |
| 詳細 | Collected in counting mode |
| 日付 | 2013年11月16日 |
| 撮影 | カテゴリ: CCD / フィルム・検出器のモデル: GATAN K2 (4k x 4k) / 実像数: 625 / 平均電子線量: 38 e/Å2 詳細: Each movie was acquired over 5 s and was comprised of 25 frames |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 40000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダー: Liquid nitrogen cooled / 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Initial data processing was performed using the Appion pipeline (including UCSF doseefgpu_driftcorr). Subsequent statistical refinement procedures and motion correction were performed using Relion. |
|---|---|
| CTF補正 | 詳細: Each particle |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 3.7 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Relion / 使用した粒子像数: 14884 |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)