[English] 日本語
 Yorodumi
Yorodumi- EMDB-27823: Cryo-EM structure of Rous sarcoma virus strand transfer complex -
+ Open data
Open data
- Basic information
Basic information
| Entry |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Title | Cryo-EM structure of Rous sarcoma virus strand transfer complex | |||||||||
 Map data Map data | Flipped map | |||||||||
 Sample Sample |
| |||||||||
 Keywords Keywords | intasome / integrase-viral DNA complex / strand transfer complex / VIRAL PROTEIN-DNA complex | |||||||||
| Function / homology |  Function and homology information Function and homology informationribonuclease H / Hydrolases; Acting on peptide bonds (peptidases); Aspartic endopeptidases / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / RNA stem-loop binding / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / Transferases; Transferring phosphorus-containing groups; Nucleotidyltransferases ...ribonuclease H / Hydrolases; Acting on peptide bonds (peptidases); Aspartic endopeptidases / DNA integration / viral genome integration into host DNA / RNA-directed DNA polymerase / establishment of integrated proviral latency / RNA stem-loop binding / RNA-directed DNA polymerase activity / RNA-DNA hybrid ribonuclease activity / Transferases; Transferring phosphorus-containing groups; Nucleotidyltransferases / viral nucleocapsid / DNA recombination / DNA-directed DNA polymerase / aspartic-type endopeptidase activity / Hydrolases; Acting on ester bonds / DNA-directed DNA polymerase activity / viral translational frameshifting / symbiont entry into host cell / proteolysis / DNA binding / zinc ion binding Similarity search - Function | |||||||||
| Biological species |  Rous sarcoma virus - Prague C Rous sarcoma virus - Prague C | |||||||||
| Method | single particle reconstruction / cryo EM / Resolution: 3.36 Å | |||||||||
 Authors Authors | Pandey KK / Bera S / Shi K / Aihara H / Grandgenett DP | |||||||||
| Funding support |  United States, 2 items United States, 2 items
| |||||||||
 Citation Citation |  Journal: J Biol Chem / Year: 2023 Journal: J Biol Chem / Year: 2023Title: Molecular determinants for Rous sarcoma virus intasome assemblies involved in retroviral integration. Authors: Sibes Bera / Ke Shi / Hideki Aihara / Duane P Grandgenett / Krishan K Pandey /  Abstract: Integration of retroviral DNA into the host genome involves the formation of integrase (IN)-DNA complexes termed intasomes. Further characterization of these complexes is needed to understand their ...Integration of retroviral DNA into the host genome involves the formation of integrase (IN)-DNA complexes termed intasomes. Further characterization of these complexes is needed to understand their assembly process. Here, we report the single-particle cryo-EM structure of the Rous sarcoma virus (RSV) strand transfer complex (STC) intasome produced with IN and a preassembled viral/target DNA substrate at 3.36 Å resolution. The conserved intasome core region consisting of IN subunits contributing active sites interacting with viral/target DNA has a resolution of 3 Å. Our structure demonstrated the flexibility of the distal IN subunits relative to the IN subunits in the conserved intasome core, similar to results previously shown with the RSV octameric cleaved synaptic complex intasome produced with IN and viral DNA only. An extensive analysis of higher resolution STC structure helped in the identification of nucleoprotein interactions important for intasome assembly. Using structure-function studies, we determined the mechanisms of several IN-DNA interactions critical for assembly of both RSV intasomes. We determined the role of IN residues R244, Y246, and S124 in cleaved synaptic complex and STC intasome assemblies and their catalytic activities, demonstrating differential effects. Taken together, these studies advance our understanding of different RSV intasome structures and molecular determinants involved in their assembly. | |||||||||
| History |
|
- Structure visualization
Structure visualization
| Supplemental images |
|---|
- Downloads & links
Downloads & links
-EMDB archive
| Map data |  emd_27823.map.gz emd_27823.map.gz | 66.6 MB |  EMDB map data format EMDB map data format | |
|---|---|---|---|---|
| Header (meta data) |  emd-27823-v30.xml emd-27823-v30.xml emd-27823.xml emd-27823.xml | 20.2 KB 20.2 KB | Display Display |  EMDB header EMDB header |
| Images |  emd_27823.png emd_27823.png | 76.6 KB | ||
| Filedesc metadata |  emd-27823.cif.gz emd-27823.cif.gz | 6.7 KB | ||
| Others |  emd_27823_half_map_1.map.gz emd_27823_half_map_1.map.gz emd_27823_half_map_2.map.gz emd_27823_half_map_2.map.gz | 120.1 MB 120.2 MB | ||
| Archive directory |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27823 http://ftp.pdbj.org/pub/emdb/structures/EMD-27823 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27823 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27823 | HTTPS FTP |
-Validation report
| Summary document |  emd_27823_validation.pdf.gz emd_27823_validation.pdf.gz | 679.1 KB | Display |  EMDB validaton report EMDB validaton report |
|---|---|---|---|---|
| Full document |  emd_27823_full_validation.pdf.gz emd_27823_full_validation.pdf.gz | 678.6 KB | Display | |
| Data in XML |  emd_27823_validation.xml.gz emd_27823_validation.xml.gz | 14.2 KB | Display | |
| Data in CIF |  emd_27823_validation.cif.gz emd_27823_validation.cif.gz | 16.8 KB | Display | |
| Arichive directory |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27823 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27823 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27823 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27823 | HTTPS FTP |
-Related structure data
| Related structure data |  8e14MC M: atomic model generated by this map C: citing same article ( |
|---|---|
| Similar structure data | Similarity search - Function & homology  F&H Search F&H Search |
- Links
Links
| EMDB pages |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| Related items in Molecule of the Month |
- Map
Map
| File |  Download / File: emd_27823.map.gz / Format: CCP4 / Size: 129.7 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) Download / File: emd_27823.map.gz / Format: CCP4 / Size: 129.7 MB / Type: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Flipped map | ||||||||||||||||||||||||||||||||||||
| Projections & slices | Image control
Images are generated by Spider. | ||||||||||||||||||||||||||||||||||||
| Voxel size | X=Y=Z: 1.16 Å | ||||||||||||||||||||||||||||||||||||
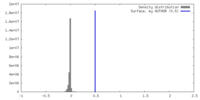
| Density |
| ||||||||||||||||||||||||||||||||||||
| Symmetry | Space group: 1 | ||||||||||||||||||||||||||||||||||||
| Details | EMDB XML:
|
-Supplemental data
-Half map: Half map A
| File | emd_27823_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half map A | ||||||||||||
| Projections & Slices |
| ||||||||||||
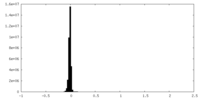
| Density Histograms |
-Half map: Half map B
| File | emd_27823_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Annotation | Half map B | ||||||||||||
| Projections & Slices |
| ||||||||||||
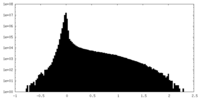
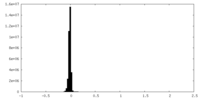
| Density Histograms |
- Sample components
Sample components
-Entire : RSV strand transfer complex
| Entire | Name: RSV strand transfer complex |
|---|---|
| Components |
|
-Supramolecule #1: RSV strand transfer complex
| Supramolecule | Name: RSV strand transfer complex / type: complex / ID: 1 / Parent: 0 / Macromolecule list: #1-#4 |
|---|---|
| Source (natural) | Organism:  Rous sarcoma virus - Prague C Rous sarcoma virus - Prague C |
| Molecular weight | Theoretical: 257 KDa |
-Macromolecule #1: integrase
| Macromolecule | Name: integrase / type: protein_or_peptide / ID: 1 / Number of copies: 8 / Enantiomer: LEVO EC number: Hydrolases; Acting on peptide bonds (peptidases); Aspartic endopeptidases |
|---|---|
| Source (natural) | Organism:  Rous sarcoma virus - Prague C / Strain: Prague C Rous sarcoma virus - Prague C / Strain: Prague C |
| Molecular weight | Theoretical: 30.926582 KDa |
| Recombinant expression | Organism:  |
| Sequence | String: PLREAKDLHT ALHIGPRALS KACNISMQQA REVVQTCPHC NSAPALEAGV NPRGLGPLQI WQTDFTLEPR MAPRSWLAVT VDTASSAIV VTQHGRVTSV AVQHHWATAI AVLGRPKAIK TDNGSCFTSK STREWLARWG IAHTTGIPGN SQGQAMVERA N RLLKDKIR ...String: PLREAKDLHT ALHIGPRALS KACNISMQQA REVVQTCPHC NSAPALEAGV NPRGLGPLQI WQTDFTLEPR MAPRSWLAVT VDTASSAIV VTQHGRVTSV AVQHHWATAI AVLGRPKAIK TDNGSCFTSK STREWLARWG IAHTTGIPGN SQGQAMVERA N RLLKDKIR VLAEGDGFMK RIPTSKQGEL LAKAMYALNH FERGENTKTP IQKHWRPTVL TEGPPVKIRI ETGEWEKGWN VL VWGRGYA AVKNRDTDKV IWVPSRKVKP DITQKDEVTK K UniProtKB: Gag-Pol polyprotein |
-Macromolecule #2: DNA (42-MER)
| Macromolecule | Name: DNA (42-MER) / type: dna / ID: 2 / Number of copies: 2 / Classification: DNA |
|---|---|
| Source (natural) | Organism:  Rous sarcoma virus - Prague C Rous sarcoma virus - Prague C |
| Molecular weight | Theoretical: 13.047456 KDa |
| Sequence | String: (DG)(DA)(DG)(DT)(DA)(DT)(DT)(DG)(DC)(DA) (DT)(DA)(DA)(DG)(DA)(DC)(DA)(DA)(DC)(DA) (DG)(DT)(DG)(DC)(DA)(DC)(DG)(DA)(DA) (DA)(DG)(DA)(DA)(DG)(DA)(DA)(DG)(DA)(DC) (DA) (DC)(DT) |
-Macromolecule #3: DNA (5'-D(*AP*AP*TP*GP*TP*TP*GP*TP*CP*TP*TP*AP*TP*GP*CP*AP*AP*TP*...
| Macromolecule | Name: DNA (5'-D(*AP*AP*TP*GP*TP*TP*GP*TP*CP*TP*TP*AP*TP*GP*CP*AP*AP*TP*AP*CP*TP*C)-3') type: dna / ID: 3 / Number of copies: 2 / Classification: DNA |
|---|---|
| Source (natural) | Organism:  Rous sarcoma virus - Prague C Rous sarcoma virus - Prague C |
| Molecular weight | Theoretical: 6.716363 KDa |
| Sequence | String: (DA)(DA)(DT)(DG)(DT)(DT)(DG)(DT)(DC)(DT) (DT)(DA)(DT)(DG)(DC)(DA)(DA)(DT)(DA)(DC) (DT)(DC) |
-Macromolecule #4: DNA (5'-D(*AP*GP*TP*GP*TP*CP*TP*TP*CP*TP*TP*CP*TP*TP*TP*C)-3')
| Macromolecule | Name: DNA (5'-D(*AP*GP*TP*GP*TP*CP*TP*TP*CP*TP*TP*CP*TP*TP*TP*C)-3') type: dna / ID: 4 / Number of copies: 2 / Classification: DNA |
|---|---|
| Source (natural) | Organism:  Rous sarcoma virus - Prague C Rous sarcoma virus - Prague C |
| Molecular weight | Theoretical: 4.821123 KDa |
| Sequence | String: (DA)(DG)(DT)(DG)(DT)(DC)(DT)(DT)(DC)(DT) (DT)(DC)(DT)(DT)(DT)(DC) |
-Macromolecule #5: ZINC ION
| Macromolecule | Name: ZINC ION / type: ligand / ID: 5 / Number of copies: 2 / Formula: ZN |
|---|---|
| Molecular weight | Theoretical: 65.409 Da |
-Experimental details
-Structure determination
| Method | cryo EM |
|---|---|
 Processing Processing | single particle reconstruction |
| Aggregation state | particle |
- Sample preparation
Sample preparation
| Concentration | 0.5 mg/mL |
|---|---|
| Buffer | pH: 7.5 |
| Grid | Model: Quantifoil R1.2/1.3 / Material: COPPER / Support film - Material: CARBON / Support film - topology: HOLEY / Support film - Film thickness: 12 / Pretreatment - Type: PLASMA CLEANING |
| Vitrification | Cryogen name: ETHANE / Chamber humidity: 100 % / Chamber temperature: 277 K / Instrument: FEI VITROBOT MARK IV |
- Electron microscopy
Electron microscopy
| Microscope | TFS KRIOS |
|---|---|
| Specialist optics | Energy filter - Name: GIF Bioquantum / Energy filter - Slit width: 20 eV |
| Image recording | Film or detector model: FEI FALCON IV (4k x 4k) / Detector mode: SUPER-RESOLUTION / Number real images: 3000 / Average electron dose: 50.0 e/Å2 |
| Electron beam | Acceleration voltage: 300 kV / Electron source:  FIELD EMISSION GUN FIELD EMISSION GUN |
| Electron optics | Illumination mode: FLOOD BEAM / Imaging mode: BRIGHT FIELD / Cs: 0.01 mm / Nominal defocus max: 2.4 µm / Nominal defocus min: 0.8 µm / Nominal magnification: 59000 |
| Sample stage | Specimen holder model: FEI TITAN KRIOS AUTOGRID HOLDER / Cooling holder cryogen: NITROGEN |
| Experimental equipment |  Model: Titan Krios / Image courtesy: FEI Company |
 Movie
Movie Controller
Controller




 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)