+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Saccharomyces cerevisiae Ufd1/Npl4/Cdc48 complex unbound but in the presence of SUMO-ubiquitin(K48polyUb)-mEOS and ATP | |||||||||
 マップデータ マップデータ | composite map of substrate unbound class of particles. This class represents the portion of particles where Ufd1/Npl4/Cdc48 was not bound to the SUMO-ubiquitin(K48polyUb)-mEOS substrate. Composite map represents the composite map of overall and focused refinements under this deposition. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ATPASE / ATPASE COMPLEX / UBIQUITIN / SUMO / SMT3 / QUALITY CONTROL / MOTOR PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報SCF complex disassembly in response to cadmium stress / mitotic DNA replication termination / Ovarian tumor domain proteases / Cdc48p-Npl4p-Vms1p AAA ATPase complex / Doa10p ubiquitin ligase complex / stress-induced homeostatically regulated protein degradation pathway / endoplasmic reticulum membrane fusion / protein localization to vacuole / sister chromatid biorientation / Hrd1p ubiquitin ligase ERAD-L complex ...SCF complex disassembly in response to cadmium stress / mitotic DNA replication termination / Ovarian tumor domain proteases / Cdc48p-Npl4p-Vms1p AAA ATPase complex / Doa10p ubiquitin ligase complex / stress-induced homeostatically regulated protein degradation pathway / endoplasmic reticulum membrane fusion / protein localization to vacuole / sister chromatid biorientation / Hrd1p ubiquitin ligase ERAD-L complex / ribophagy / DNA replication termination / RQC complex / mitochondria-associated ubiquitin-dependent protein catabolic process / cytoplasm protein quality control by the ubiquitin-proteasome system / positive regulation of mitochondrial fusion / HSF1 activation / nuclear protein quality control by the ubiquitin-proteasome system / protein-containing complex disassembly / protein transport to vacuole involved in ubiquitin-dependent protein catabolic process via the multivesicular body sorting pathway / endosome to plasma membrane protein transport / protein phosphatase regulator activity / Translesion Synthesis by POLH / piecemeal microautophagy of the nucleus / mating projection tip / mitotic spindle disassembly / Protein methylation / VCP-NPL4-UFD1 AAA ATPase complex / replisome / vesicle-fusing ATPase / ribosome-associated ubiquitin-dependent protein catabolic process / K48-linked polyubiquitin modification-dependent protein binding / retrograde protein transport, ER to cytosol / nuclear outer membrane-endoplasmic reticulum membrane network / nonfunctional rRNA decay / KEAP1-NFE2L2 pathway / Neddylation / protein quality control for misfolded or incompletely synthesized proteins / polyubiquitin modification-dependent protein binding / autophagosome maturation / mRNA transport / ATP metabolic process / ERAD pathway / Neutrophil degranulation / rescue of stalled ribosome / ubiquitin binding / macroautophagy / positive regulation of protein localization to nucleus / ubiquitin-dependent protein catabolic process / nuclear membrane / proteasome-mediated ubiquitin-dependent protein catabolic process / ubiquitin protein ligase binding / endoplasmic reticulum membrane / perinuclear region of cytoplasm / ATP hydrolysis activity / mitochondrion / ATP binding / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
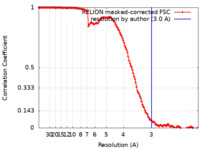
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | |||||||||
 データ登録者 データ登録者 | Lee HG / Lima CD | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: SUMO enhances unfolding of SUMO-polyubiquitin-modified substrates by the Ufd1/Npl4/Cdc48 complex. 著者: Hyein G Lee / Abigail A Lemmon / Christopher D Lima /  要旨: The Ufd1/Npl4/Cdc48 complex is a universal protein segregase that plays key roles in eukaryotic cellular processes. Its functions orchestrating the clearance or removal of polyubiquitylated targets ...The Ufd1/Npl4/Cdc48 complex is a universal protein segregase that plays key roles in eukaryotic cellular processes. Its functions orchestrating the clearance or removal of polyubiquitylated targets are established; however, prior studies suggest that the complex also targets substrates modified by the ubiquitin-like protein SUMO. Here, we show that interactions between Ufd1 and SUMO enhance unfolding of substrates modified by SUMO-polyubiquitin hybrid chains by the budding yeast Ufd1/Npl4/Cdc48 complex compared to substrates modified by polyubiquitin chains, a difference that is accentuated when the complex has a choice between these substrates. Incubating Ufd1/Npl4/Cdc48 with a substrate modified by a SUMO-polyubiquitin hybrid chain produced a series of single-particle cryo-EM structures that reveal features of interactions between Ufd1/Npl4/Cdc48 and ubiquitin prior to and during unfolding of ubiquitin. These results are consistent with cellular functions for SUMO and ubiquitin modifications and support a physical model wherein Ufd1/Npl4/Cdc48, SUMO, and ubiquitin conjugation pathways converge to promote clearance of proteins modified with SUMO and polyubiquitin. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27273.map.gz emd_27273.map.gz | 25.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27273-v30.xml emd-27273-v30.xml emd-27273.xml emd-27273.xml | 42.4 KB 42.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27273_fsc.xml emd_27273_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27273.png emd_27273.png | 81.8 KB | ||
| Filedesc metadata |  emd-27273.cif.gz emd-27273.cif.gz | 7.7 KB | ||
| その他 |  emd_27273_additional_1.map.gz emd_27273_additional_1.map.gz emd_27273_additional_10.map.gz emd_27273_additional_10.map.gz emd_27273_additional_2.map.gz emd_27273_additional_2.map.gz emd_27273_additional_3.map.gz emd_27273_additional_3.map.gz emd_27273_additional_4.map.gz emd_27273_additional_4.map.gz emd_27273_additional_5.map.gz emd_27273_additional_5.map.gz emd_27273_additional_6.map.gz emd_27273_additional_6.map.gz emd_27273_additional_7.map.gz emd_27273_additional_7.map.gz emd_27273_additional_8.map.gz emd_27273_additional_8.map.gz emd_27273_additional_9.map.gz emd_27273_additional_9.map.gz emd_27273_half_map_1.map.gz emd_27273_half_map_1.map.gz emd_27273_half_map_2.map.gz emd_27273_half_map_2.map.gz | 13.3 MB 171.4 MB 14.7 MB 18.5 MB 10.6 MB 171.5 MB 171.5 MB 171.6 MB 171.4 MB 171.7 MB 171.4 MB 171.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27273 http://ftp.pdbj.org/pub/emdb/structures/EMD-27273 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27273 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27273 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_27273_validation.pdf.gz emd_27273_validation.pdf.gz | 740.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_27273_full_validation.pdf.gz emd_27273_full_validation.pdf.gz | 740.4 KB | 表示 | |
| XML形式データ |  emd_27273_validation.xml.gz emd_27273_validation.xml.gz | 21.5 KB | 表示 | |
| CIF形式データ |  emd_27273_validation.cif.gz emd_27273_validation.cif.gz | 28.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27273 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27273 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27273 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-27273 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27273.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27273.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | composite map of substrate unbound class of particles. This class represents the portion of particles where Ufd1/Npl4/Cdc48 was not bound to the SUMO-ubiquitin(K48polyUb)-mEOS substrate. Composite map represents the composite map of overall and focused refinements under this deposition. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.064 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: focused refinement of the D1/D2 domains of Cdc48...
+追加マップ: focused refinement half map 1 of the D1/D2...
+追加マップ: focused refinement of cdc48 of substrate unbound class...
+追加マップ: overall refinement of substrate unbound class of particles,...
+追加マップ: focused refinement of the central densities of Ufd1/Npl4...
+追加マップ: focused refinement half map 2 of the D1/D2...
+追加マップ: focused refinement half map 1 of Cdc48 of...
+追加マップ: focused refinement half map 1 of the central...
+追加マップ: focused refinement half map 2 of Cdc48 of...
+追加マップ: focused refinement half map 2 of the central...
+ハーフマップ: overall refinement half map 2 of substrate unbound class of particles
+ハーフマップ: overall refinement half map 1 of substrate unbound class of particles
- 試料の構成要素
試料の構成要素
-全体 : Saccharomyces cerevisiae Ufd1/Npl4/Cdc48 complex unbound but in t...
| 全体 | 名称: Saccharomyces cerevisiae Ufd1/Npl4/Cdc48 complex unbound but in the presence of SUMO-ubiquitin(K48polyUb)-mEOS and ATP |
|---|---|
| 要素 |
|
-超分子 #1: Saccharomyces cerevisiae Ufd1/Npl4/Cdc48 complex unbound but in t...
| 超分子 | 名称: Saccharomyces cerevisiae Ufd1/Npl4/Cdc48 complex unbound but in the presence of SUMO-ubiquitin(K48polyUb)-mEOS and ATP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#3 詳細: This class represents the portion of particles where Ufd1/Npl4/Cdc48 was not bound to the SUMO-ubiquitin(K48polyUb)-mEOS substrate. Composite map represents the composite map of overall and ...詳細: This class represents the portion of particles where Ufd1/Npl4/Cdc48 was not bound to the SUMO-ubiquitin(K48polyUb)-mEOS substrate. Composite map represents the composite map of overall and focused refinements under this deposition. |
|---|
-超分子 #2: Cdc48 hexamer
| 超分子 | 名称: Cdc48 hexamer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #3: Ufd1/Npl4 tower
| 超分子 | 名称: Ufd1/Npl4 tower / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Cell division control protein 48
| 分子 | 名称: Cell division control protein 48 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号: vesicle-fusing ATPase |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 92.389195 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMGEEHKP LLDASGVDPR EEDKTATAIL RRKKKDNMLL VDDAINDDNS VIAINSNTMD KLELFRGDTV LVKGKKRKDT VLIVLIDDE LEDGACRINR VVRNNLRIRL GDLVTIHPCP DIKYATRISV LPIADTIEGI TGNLFDVFLK PYFVEAYRPV R KGDHFVVR ...文字列: GSHMGEEHKP LLDASGVDPR EEDKTATAIL RRKKKDNMLL VDDAINDDNS VIAINSNTMD KLELFRGDTV LVKGKKRKDT VLIVLIDDE LEDGACRINR VVRNNLRIRL GDLVTIHPCP DIKYATRISV LPIADTIEGI TGNLFDVFLK PYFVEAYRPV R KGDHFVVR GGMRQVEFKV VDVEPEEYAV VAQDTIIHWE GEPINREDEE NNMNEVGYDD IGGCRKQMAQ IREMVELPLR HP QLFKAIG IKPPRGVLMY GPPGTGKTLM ARAVANETGA FFFLINGPEV MSKMAGESES NLRKAFEEAE KNAPAIIFID EID SIAPKR DKTNGEVERR VVSQLLTLMD GMKARSNVVV IAATNRPNSI DPALRRFGRF DREVDIGIPD ATGRLEVLRI HTKN MKLAD DVDLEALAAE THGYVGADIA SLCSEAAMQQ IREKMDLIDL DEDEIDAEVL DSLGVTMDNF RFALGNSNPS ALRET VVES VNVTWDDVGG LDEIKEELKE TVEYPVLHPD QYTKFGLSPS KGVLFYGPPG TGKTLLAKAV ATEVSANFIS VKGPEL LSM WYGESESNIR DIFDKARAAA PTVVFLDELD SIAKARGGSL GDAGGASDRV VNQLLTEMDG MNAKKNVFVI GATNRPD QI DPAILRPGRL DQLIYVPLPD ENARLSILNA QLRKTPLEPG LELTAIAKAT QGFSGADLLY IVQRAAKYAI KDSIEAHR Q HEAEKEVKVE GEDVEMTDEG AKAEQEPEVD PVPYITKEHF AEAMKTAKRS VSDAELRRYE AYSQQMKASR GQFSNFNFN DAPLGTTATD NANSNNSAPS GAGAAFGSNA EEDDDLYS UniProtKB: Cell division control protein 48 |
-分子 #2: Nuclear protein localization protein 4
| 分子 | 名称: Nuclear protein localization protein 4 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 66.144336 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSHMLIRFRS KNGTHRVSCQ ENDLFGTVIE KLVGNLDPNA DVDTFTVCEK PGQGIHAVSE LADRTVMDLG LKHGDMLILN YSDKPANEK DGVNVEIGSV GIDSKGIRQH RYGPLRIKEL AVDEELEKED GLIPRQKSKL CKHGDRGMCE YCSPLPPWDK E YHEKNKIK ...文字列: GSHMLIRFRS KNGTHRVSCQ ENDLFGTVIE KLVGNLDPNA DVDTFTVCEK PGQGIHAVSE LADRTVMDLG LKHGDMLILN YSDKPANEK DGVNVEIGSV GIDSKGIRQH RYGPLRIKEL AVDEELEKED GLIPRQKSKL CKHGDRGMCE YCSPLPPWDK E YHEKNKIK HISFHSYLKK LNENANKKEN GSSYISPLSE PDFRINKRCH NGHEPWPRGI CSKCQPSAIT LQQQEFRMVD HV EFQKSEI INEFIQAWRY TGMQRFGYMY GSYSKYDNTP LGIKAVVEAI YEPPQHDEQD GLTMDVEQVK NEMLQIDRQA QEM GLSRIG LIFTDLSDAG AGDGSVFCKR HKDSFFLSSL EVIMAARHQT RHPNVSKYSE QGFFSSKFVT CVISGNLEGE IDIS SYQVS TEAEALVTAD MISGSTFPSM AYINDTTDER YVPEIFYMKS NEYGITVKEN AKPAFPVDYL LVTLTHGFPN TDTET NSKF VSSTGFPWSN RQAMGQSQDY QELKKYLFNV ASSGDFNLLH EKISNFHLLL YINSLQILSP DEWKLLIESA VKNEWE ESL LKLVSSAGWQ TLVMILQESG UniProtKB: Nuclear protein localization protein 4 |
-分子 #3: Ubiquitin fusion degradation protein 1
| 分子 | 名称: Ubiquitin fusion degradation protein 1 / タイプ: protein_or_peptide / ID: 3 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 40.001449 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: GSMFSGFSSF GGGNGFVNMP QTFEEFFRCY PIAMMNDRIR KDDANFGGKI FLPPSALSKL SMLNIRYPML FKLTANETGR VTHGGVLEF IAEEGRVYLP QWMMETLGIQ PGSLLQISST DVPLGQFVKL EPQSVDFLDI SDPKAVLENV LRNFSTLTVD D VIEISYNG ...文字列: GSMFSGFSSF GGGNGFVNMP QTFEEFFRCY PIAMMNDRIR KDDANFGGKI FLPPSALSKL SMLNIRYPML FKLTANETGR VTHGGVLEF IAEEGRVYLP QWMMETLGIQ PGSLLQISST DVPLGQFVKL EPQSVDFLDI SDPKAVLENV LRNFSTLTVD D VIEISYNG KTFKIKILEV KPESSSKSIC VIETDLVTDF APPVGYVEPD YKALKAQQDK EKKNSFGKGQ VLDPSVLGQG SM STRIDYA GIANSSRNKL SKFVGQGQNI SGKAPKAEPK QDIKDMKITF DGEPAKLDLP EGQLFFGFPM VLPKEDEESA AGS KSSEQN FQGQGISLRK SNKRKTKSDH DSSKSKAPKS PEVIEID UniProtKB: Ubiquitin fusion degradation protein 1 |
-分子 #4: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 6 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #5: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 5 / コピー数: 6 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information |  ChemComp-ADP: |
-分子 #6: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 6 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM HEPES pH 8.0, 150 mM NaCl, 0.1 mM TCEP, 1 mM MgCl2, 5 mM ATP. Added 0.05% CHAPSO before vitrification. |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: 8 s wait, 4 s blot before plunging. |
| 詳細 | Ufd1/Npl4/Cdc48 was pre-incubated with SUMO-ubiquitin(K48polyUb)-mEOS and ATP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 70.577 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.2 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー
































 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)