+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2672 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | 35O22 Fab in complex with BG505 SOSIP.664 Trimer | |||||||||
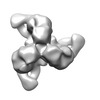
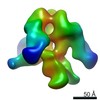
 マップデータ マップデータ | Negative stain reconstruction of 35O22 Fab bound to BG505 SOSIP trimer | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | HIV-1 / Env / glycoprotein / bnAb / antibody / Fab | |||||||||
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) /  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 19.0 Å | |||||||||
 データ登録者 データ登録者 | Lee JH / Pancera M / Kwong PD / Connors M / Ward AB | |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2014 ジャーナル: Nature / 年: 2014タイトル: Broad and potent HIV-1 neutralization by a human antibody that binds the gp41-gp120 interface. 著者: Jinghe Huang / Byong H Kang / Marie Pancera / Jeong Hyun Lee / Tommy Tong / Yu Feng / Hiromi Imamichi / Ivelin S Georgiev / Gwo-Yu Chuang / Aliaksandr Druz / Nicole A Doria-Rose / Leo Laub / ...著者: Jinghe Huang / Byong H Kang / Marie Pancera / Jeong Hyun Lee / Tommy Tong / Yu Feng / Hiromi Imamichi / Ivelin S Georgiev / Gwo-Yu Chuang / Aliaksandr Druz / Nicole A Doria-Rose / Leo Laub / Kwinten Sliepen / Marit J van Gils / Alba Torrents de la Peña / Ronald Derking / Per-Johan Klasse / Stephen A Migueles / Robert T Bailer / Munir Alam / Pavel Pugach / Barton F Haynes / Richard T Wyatt / Rogier W Sanders / James M Binley / Andrew B Ward / John R Mascola / Peter D Kwong / Mark Connors /   要旨: The isolation of human monoclonal antibodies is providing important insights into the specificities that underlie broad neutralization of HIV-1 (reviewed in ref. 1). Here we report a broad and ...The isolation of human monoclonal antibodies is providing important insights into the specificities that underlie broad neutralization of HIV-1 (reviewed in ref. 1). Here we report a broad and extremely potent HIV-specific monoclonal antibody, termed 35O22, which binds a novel HIV-1 envelope glycoprotein (Env) epitope. 35O22 neutralized 62% of 181 pseudoviruses with a half-maximum inhibitory concentration (IC50) <50 μg ml(-1). The median IC50 of neutralized viruses was 0.033 μg ml(-1), among the most potent thus far described. 35O22 did not bind monomeric forms of Env tested, but did bind the trimeric BG505 SOSIP.664. Mutagenesis and a reconstruction by negative-stain electron microscopy of the Fab in complex with trimer revealed that it bound to a conserved epitope, which stretched across gp120 and gp41. The specificity of 35O22 represents a novel site of vulnerability on HIV Env, which serum analysis indicates to be commonly elicited by natural infection. Binding to this new site of vulnerability may thus be an important complement to current monoclonal-antibody-based approaches to immunotherapies, prophylaxis and vaccine design. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2672.map.gz emd_2672.map.gz | 13.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2672-v30.xml emd-2672-v30.xml emd-2672.xml emd-2672.xml | 13.9 KB 13.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2672.png emd_2672.png | 83.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2672 http://ftp.pdbj.org/pub/emdb/structures/EMD-2672 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2672 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2672 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2672_validation.pdf.gz emd_2672_validation.pdf.gz | 206.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2672_full_validation.pdf.gz emd_2672_full_validation.pdf.gz | 205.5 KB | 表示 | |
| XML形式データ |  emd_2672_validation.xml.gz emd_2672_validation.xml.gz | 5.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2672 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2672 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2672 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2672 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2672.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2672.map.gz / 形式: CCP4 / 大きさ: 15.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Negative stain reconstruction of 35O22 Fab bound to BG505 SOSIP trimer | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.05 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
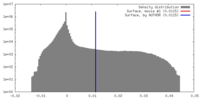
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Fab fragment of bnAb 35O22 bound to recombinant gp140 Env trimer ...
| 全体 | 名称: Fab fragment of bnAb 35O22 bound to recombinant gp140 Env trimer BG505 SOSIP.664 |
|---|---|
| 要素 |
|
-超分子 #1000: Fab fragment of bnAb 35O22 bound to recombinant gp140 Env trimer ...
| 超分子 | 名称: Fab fragment of bnAb 35O22 bound to recombinant gp140 Env trimer BG505 SOSIP.664 タイプ: sample / ID: 1000 / 集合状態: One Fab binds per gp140 monomer / Number unique components: 2 |
|---|---|
| 分子量 | 理論値: 570 KDa |
-分子 #1: Soluble HIV-1 Envelope glycoprotein
| 分子 | 名称: Soluble HIV-1 Envelope glycoprotein / タイプ: protein_or_peptide / ID: 1 / Name.synonym: BG505 SOSIP.664 / コピー数: 1 / 集合状態: Trimer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)別称: HIV-1 |
| 分子量 | 理論値: 420 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293T Homo sapiens (ヒト) / 組換細胞: HEK293T |
-分子 #2: Fab fragment of bnAb 35O22
| 分子 | 名称: Fab fragment of bnAb 35O22 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) / 別称: Human Homo sapiens (ヒト) / 別称: Human |
| 組換発現 | 生物種:  Homo sapiens (ヒト) / 組換細胞: HEK293T Homo sapiens (ヒト) / 組換細胞: HEK293T |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 50 mM Tris, 150 mM NaCl |
| 染色 | タイプ: NEGATIVE 詳細: 3 uL of protein applied, blotted then applied 2% UF for 45 seconds. |
| グリッド | 詳細: 400 Cu mesh grid with carbon support, plasma cleaned. |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法 #1
電子顕微鏡法 #1
| Microscopy ID | 1 |
|---|---|
| 顕微鏡 | FEI TECNAI 12 |
| 温度 | 平均: 293 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism corrected at 52,000x mag. |
| 日付 | 2013年11月27日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 実像数: 139 / 平均電子線量: 30 e/Å2 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 52000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
- 電子顕微鏡法 #2
電子顕微鏡法 #2
| Microscopy ID | 2 |
|---|---|
| 顕微鏡 | FEI TECNAI 12 |
| 温度 | 平均: 293 K |
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism corrected at 52,000x mag. |
| 日付 | 2014年1月22日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 実像数: 139 / 平均電子線量: 30 e/Å2 |
| Tilt angle min | 0 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 倍率(補正後): 52000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 0.7 µm / 倍率(公称値): 52000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 19.0 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: EMAN2, SPARX / 使用した粒子像数: 4746 |
|---|
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - #0 - Chain ID: H / Chain - #1 - Chain ID: L |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Three copies of the Fab were fit into the EM map |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - #0 - Chain ID: A / Chain - #1 - Chain ID: B / Chain - #2 - Chain ID: E / Chain - #3 - Chain ID: F / Chain - #4 - Chain ID: I / Chain - #5 - Chain ID: J |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)