+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of JetD from Pseudomonas aeruginosa | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Wadjet / Bacterial defense systems / JetD / Anti-plasmid defense system / EptD / MksG / Toprim domain / Nuclease / Topoisomerase / DNA BINDING PROTEIN | |||||||||
| 機能・相同性 | Uncharacterised conserved protein UCP028408 / Wadjet protein JetD, C-terminal / Domain of unknown function DUF3322 / Wadjet protein JetD, C-terminal / Uncharacterized protein conserved in bacteria N-term (DUF3322) / DUF3322 and DUF2220 domain-containing protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Pseudomonas aeruginosa PA14 (緑膿菌) Pseudomonas aeruginosa PA14 (緑膿菌) | |||||||||
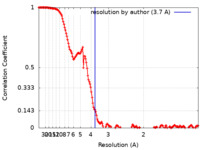
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Deep A / Gu Y | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Mol Cell / 年: 2022 ジャーナル: Mol Cell / 年: 2022タイトル: The SMC-family Wadjet complex protects bacteria from plasmid transformation by recognition and cleavage of closed-circular DNA. 著者: Amar Deep / Yajie Gu / Yong-Qi Gao / Kaori M Ego / Mark A Herzik / Huilin Zhou / Kevin D Corbett /  要旨: Self versus non-self discrimination is a key element of innate and adaptive immunity across life. In bacteria, CRISPR-Cas and restriction-modification systems recognize non-self nucleic acids through ...Self versus non-self discrimination is a key element of innate and adaptive immunity across life. In bacteria, CRISPR-Cas and restriction-modification systems recognize non-self nucleic acids through their sequence and their methylation state, respectively. Here, we show that the Wadjet defense system recognizes DNA topology to protect its host against plasmid transformation. By combining cryoelectron microscopy with cross-linking mass spectrometry, we show that Wadjet forms a complex similar to the bacterial condensin complex MukBEF, with a novel nuclease subunit similar to a type II DNA topoisomerase. Wadjet specifically cleaves closed-circular DNA in a reaction requiring ATP hydrolysis by the structural maintenance of chromosome (SMC) ATPase subunit JetC, suggesting that the complex could use DNA loop extrusion to sense its substrate's topology, then specifically activate the nuclease subunit JetD to cleave plasmid DNA. Overall, our data reveal how bacteria have co-opted a DNA maintenance machine to specifically recognize and destroy foreign DNAs through topology sensing. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25913.map.gz emd_25913.map.gz | 169.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25913-v30.xml emd-25913-v30.xml emd-25913.xml emd-25913.xml | 28 KB 28 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25913_fsc.xml emd_25913_fsc.xml | 15.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25913.png emd_25913.png | 104.8 KB | ||
| Filedesc metadata |  emd-25913.cif.gz emd-25913.cif.gz | 6.1 KB | ||
| その他 |  emd_25913_additional_1.map.gz emd_25913_additional_1.map.gz emd_25913_additional_2.map.gz emd_25913_additional_2.map.gz emd_25913_additional_3.map.gz emd_25913_additional_3.map.gz emd_25913_additional_4.map.gz emd_25913_additional_4.map.gz emd_25913_additional_5.map.gz emd_25913_additional_5.map.gz emd_25913_additional_6.map.gz emd_25913_additional_6.map.gz emd_25913_half_map_1.map.gz emd_25913_half_map_1.map.gz emd_25913_half_map_2.map.gz emd_25913_half_map_2.map.gz | 318.3 MB 318.3 MB 170.2 MB 318.1 MB 318.1 MB 170 MB 318.7 MB 318.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25913 http://ftp.pdbj.org/pub/emdb/structures/EMD-25913 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25913 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25913 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25913_validation.pdf.gz emd_25913_validation.pdf.gz | 1 MB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25913_full_validation.pdf.gz emd_25913_full_validation.pdf.gz | 1 MB | 表示 | |
| XML形式データ |  emd_25913_validation.xml.gz emd_25913_validation.xml.gz | 24.1 KB | 表示 | |
| CIF形式データ |  emd_25913_validation.cif.gz emd_25913_validation.cif.gz | 31.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25913 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25913 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25913 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25913 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25913.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25913.map.gz / 形式: CCP4 / 大きさ: 343 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.65 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #6
| ファイル | emd_25913_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
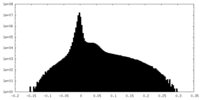
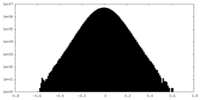
| 密度ヒストグラム |
-追加マップ: #5
| ファイル | emd_25913_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
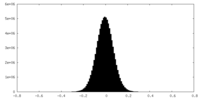
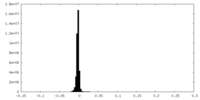
| 密度ヒストグラム |
-追加マップ: #4
| ファイル | emd_25913_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
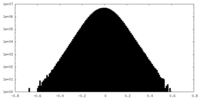
| 密度ヒストグラム |
-追加マップ: #3
| ファイル | emd_25913_additional_4.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
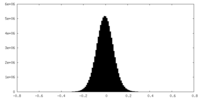
| 密度ヒストグラム |
-追加マップ: #2
| ファイル | emd_25913_additional_5.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
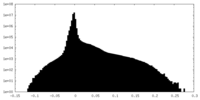
| 密度ヒストグラム |
-追加マップ: #1
| ファイル | emd_25913_additional_6.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
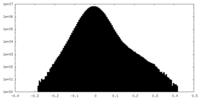
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_25913_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_25913_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : JetD
| 全体 | 名称: JetD |
|---|---|
| 要素 |
|
-超分子 #1: JetD
| 超分子 | 名称: JetD / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all / 詳細: A Pseudomonas aeruginosa protein. |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PA14 (緑膿菌) Pseudomonas aeruginosa PA14 (緑膿菌) |
| 分子量 | 理論値: 85.6 kDa/nm |
-分子 #1: JetD
| 分子 | 名称: JetD / タイプ: protein_or_peptide / ID: 1 詳細: Pseudomonas aeruginosa PA14 JetD protein with N-terminal TEV cleavable 6XHis tag. コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Pseudomonas aeruginosa PA14 (緑膿菌) / 株: UCBPP-PA14 Pseudomonas aeruginosa PA14 (緑膿菌) / 株: UCBPP-PA14 |
| 分子量 | 理論値: 45.132164 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKSSHHHHHH ENLYFQSNAK SPTDIGRNLA RQWQRSSVRL ERLLNPGSWP QSLNIGKPSA RLFTDNIQAV LRHVENWRSV NVGKVDWEP VSYRAGLAPI SLPMRWHLRS PSEWIAASDD PRVSQEYAQL EYLVEQVDSA FHTLLVAQRS LWLTRTPEEV V ATAQLATQ ...文字列: MKSSHHHHHH ENLYFQSNAK SPTDIGRNLA RQWQRSSVRL ERLLNPGSWP QSLNIGKPSA RLFTDNIQAV LRHVENWRSV NVGKVDWEP VSYRAGLAPI SLPMRWHLRS PSEWIAASDD PRVSQEYAQL EYLVEQVDSA FHTLLVAQRS LWLTRTPEEV V ATAQLATQ LAPGCAQGRP LRLLAEHGVD TKFFERNATL LTKLLDERFE GAASEQGLTT FLDAFEESSH WVLVVPLQPG LL PFKRLRL TTSELAETPL PGSRLLVVEN EQCVHLLPET LPDTLAVLGA GLDLHWLASA HLAGKQIGYW GDMDTWGLLM LAR ARLHQP AVEALLMEQE LFEQHSQGNT VVEPAKALES APPGLLADEA DFYRYLLVQE RGRLEQEYLP KAQVELAIRK WAR UniProtKB: DUF3322 and DUF2220 domain-containing protein |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.10 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Prepared using deionized water and filtered strelized. | ||||||||||||
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 12 sec. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 平均電子線量: 62.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL |
|---|---|
| 得られたモデル |  PDB-7til: |
 ムービー
ムービー コントローラー
コントローラー










 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)