+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-25827 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AtTPC1 D454N with 1 mM EDTA state I | |||||||||
 マップデータ マップデータ | Volume from non-uniform refinement, post-processed with Deepemhancer (inputs are the two provided half maps) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ion channel / voltage activation / VGIC / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of jasmonic acid biosynthetic process / seed germination / regulation of stomatal movement / plant-type vacuole / vacuole / vacuolar membrane / monoatomic ion channel complex / voltage-gated calcium channel activity / calcium-mediated signaling / calcium ion transport ...regulation of jasmonic acid biosynthetic process / seed germination / regulation of stomatal movement / plant-type vacuole / vacuole / vacuolar membrane / monoatomic ion channel complex / voltage-gated calcium channel activity / calcium-mediated signaling / calcium ion transport / calcium ion binding / Golgi apparatus / identical protein binding / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
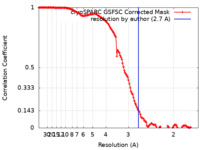
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Dickinson MS / Stroud RM | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2022 ジャーナル: Proc Natl Acad Sci U S A / 年: 2022タイトル: Molecular basis of multistep voltage activation in plant two-pore channel 1. 著者: Miles Sasha Dickinson / Jinping Lu / Meghna Gupta / Irene Marten / Rainer Hedrich / Robert M Stroud /   要旨: Voltage-gated ion channels confer excitability to biological membranes, initiating and propagating electrical signals across large distances on short timescales. Membrane excitation requires channels ...Voltage-gated ion channels confer excitability to biological membranes, initiating and propagating electrical signals across large distances on short timescales. Membrane excitation requires channels that respond to changes in electric field and couple the transmembrane voltage to gating of a central pore. To address the mechanism of this process in a voltage-gated ion channel, we determined structures of the plant two-pore channel 1 at different stages along its activation coordinate. These high-resolution structures of activation intermediates, when compared with the resting-state structure, portray a mechanism in which the voltage-sensing domain undergoes dilation and in-membrane plane rotation about the gating charge-bearing helix, followed by charge translocation across the charge transfer seal. These structures, in concert with patch-clamp electrophysiology, show that residues in the pore mouth sense inhibitory Ca and are allosterically coupled to the voltage sensor. These conformational changes provide insight into the mechanism of voltage-sensor domain activation in which activation occurs vectorially over a series of elementary steps. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25827.map.gz emd_25827.map.gz | 188.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25827-v30.xml emd-25827-v30.xml emd-25827.xml emd-25827.xml | 18.6 KB 18.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25827_fsc.xml emd_25827_fsc.xml | 12.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25827.png emd_25827.png | 118.8 KB | ||
| Filedesc metadata |  emd-25827.cif.gz emd-25827.cif.gz | 6.2 KB | ||
| その他 |  emd_25827_additional_1.map.gz emd_25827_additional_1.map.gz emd_25827_half_map_1.map.gz emd_25827_half_map_1.map.gz emd_25827_half_map_2.map.gz emd_25827_half_map_2.map.gz | 30.1 MB 203.5 MB 203.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25827 http://ftp.pdbj.org/pub/emdb/structures/EMD-25827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25827 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25827 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25827_validation.pdf.gz emd_25827_validation.pdf.gz | 724.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25827_full_validation.pdf.gz emd_25827_full_validation.pdf.gz | 723.9 KB | 表示 | |
| XML形式データ |  emd_25827_validation.xml.gz emd_25827_validation.xml.gz | 21.6 KB | 表示 | |
| CIF形式データ |  emd_25827_validation.cif.gz emd_25827_validation.cif.gz | 27.8 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25827 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25827 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25827 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25827 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25827.map.gz / 形式: CCP4 / 大きさ: 219.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25827.map.gz / 形式: CCP4 / 大きさ: 219.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Volume from non-uniform refinement, post-processed with Deepemhancer (inputs are the two provided half maps) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.835 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: #1
| ファイル | emd_25827_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 1 from non-uniform refinement
| ファイル | emd_25827_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 1 from non-uniform refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map 2 from non-uniform refinement
| ファイル | emd_25827_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map 2 from non-uniform refinement | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : AtTPC1 D454N with 1 mM EDTA state I
| 全体 | 名称: AtTPC1 D454N with 1 mM EDTA state I |
|---|---|
| 要素 |
|
-超分子 #1: AtTPC1 D454N with 1 mM EDTA state I
| 超分子 | 名称: AtTPC1 D454N with 1 mM EDTA state I / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 168 KDa |
-分子 #1: Two pore calcium channel protein 1
| 分子 | 名称: Two pore calcium channel protein 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 84.939844 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MEDPLIGRDS LGGGGTDRVR RSEAITHGTP FQKAAALVDL AEDGIGLPVE ILDQSSFGES ARYYFIFTRL DLIWSLNYFA LLFLNFFEQ PLWCEKNPKP SCKDRDYYYL GELPYLTNAE SIIYEVITLA ILLVHTFFPI SYEGSRIFWT SRLNLVKVAC V VILFVDVL ...文字列: MEDPLIGRDS LGGGGTDRVR RSEAITHGTP FQKAAALVDL AEDGIGLPVE ILDQSSFGES ARYYFIFTRL DLIWSLNYFA LLFLNFFEQ PLWCEKNPKP SCKDRDYYYL GELPYLTNAE SIIYEVITLA ILLVHTFFPI SYEGSRIFWT SRLNLVKVAC V VILFVDVL VDFLYLSPLA FDFLPFRIAP YVRVIIFILS IRELRDTLVL LSGMLGTYLN ILALWMLFLL FASWIAFVMF ED TQQGLTV FTSYGATLYQ MFILFTTSNN PDVWIPAYKS SRWSSVFFVL YVLIGVYFVT NLILAVVYDS FKEQLAKQVS GMD QMKRRM LEKAFGLIDS DKNGEIDKNQ CIKLFEQLTN YRTLPKISKE EFGLIFDELD DTRDFKINKD EFADLCQAIA LRFQ KEEVP SLFEHFPQIY HSALSQQLRA FVRSPNFGYA ISFILIINFI AVVVETTLNI EESSAQKPWQ VAEFVFGWIY VLEMA LKIY TYGFENYWRE GANRFDFLVT WVIVIGETAT FITPDENTFF SNGEWIRYLL LARMLRLIRL LMNVQRYRAF IATFIT LIP SLMPYLGTIF CVLCIYCSIG VQVFGGLVNA GNKKLFETEL AEDDYLLFNF NDYPNGMVTL FNLLVMGNWQ VWMESYK DL TGTWWSITYF VSFYVITILL LLNLVVAFVL EAFFTELDLE EEEKCQGQDS QEKRNRRRSA GSKSRSQRVD TLLHHMLG D ELSKPECSTS DT UniProtKB: Two pore calcium channel protein 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 50 mM Tris, 200 mM NaCl, 0.06% glycodiosgenin, 1 mM EDTA |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 66.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: FLEXIBLE FIT |
|---|---|
| 得られたモデル |  PDB-7tdf: |
 ムービー
ムービー コントローラー
コントローラー



















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)