+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of VRK1 C-terminal tail bound to nucleosome core particle | ||||||||||||
 マップデータ マップデータ | Map of crosslinked VRK1-nucleosome complex | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | nucleosome / chromatin / NUCLEAR PROTEIN / STRUCTURAL PROTEIN-DNA-TRANSFERASE complex | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報histone H2AX kinase activity / Cajal body organization / Golgi disassembly / Nuclear Envelope Breakdown / positive regulation of protein localization to chromatin / mitotic nuclear membrane disassembly / regulation of neuron migration / Golgi stack / Initiation of Nuclear Envelope (NE) Reformation / nucleosomal DNA binding ...histone H2AX kinase activity / Cajal body organization / Golgi disassembly / Nuclear Envelope Breakdown / positive regulation of protein localization to chromatin / mitotic nuclear membrane disassembly / regulation of neuron migration / Golgi stack / Initiation of Nuclear Envelope (NE) Reformation / nucleosomal DNA binding / negative regulation of megakaryocyte differentiation / protein localization to CENP-A containing chromatin / Cajal body / Chromatin modifying enzymes / Replacement of protamines by nucleosomes in the male pronucleus / CENP-A containing nucleosome / Packaging Of Telomere Ends / Recognition and association of DNA glycosylase with site containing an affected purine / Cleavage of the damaged purine / Recognition and association of DNA glycosylase with site containing an affected pyrimidine / Cleavage of the damaged pyrimidine / Deposition of new CENPA-containing nucleosomes at the centromere / telomere organization / Inhibition of DNA recombination at telomere / Meiotic synapsis / Interleukin-7 signaling / RNA Polymerase I Promoter Opening / Assembly of the ORC complex at the origin of replication / Regulation of endogenous retroelements by the Human Silencing Hub (HUSH) complex / innate immune response in mucosa / SUMOylation of chromatin organization proteins / DNA methylation / Condensation of Prophase Chromosomes / Chromatin modifications during the maternal to zygotic transition (MZT) / HCMV Late Events / SIRT1 negatively regulates rRNA expression / ERCC6 (CSB) and EHMT2 (G9a) positively regulate rRNA expression / PRC2 methylates histones and DNA / Regulation of endogenous retroelements by KRAB-ZFP proteins / Defective pyroptosis / Regulation of endogenous retroelements by Piwi-interacting RNAs (piRNAs) / HDACs deacetylate histones / Nonhomologous End-Joining (NHEJ) / RNA Polymerase I Promoter Escape / Transcriptional regulation by small RNAs / Formation of the beta-catenin:TCF transactivating complex / RUNX1 regulates genes involved in megakaryocyte differentiation and platelet function / Activated PKN1 stimulates transcription of AR (androgen receptor) regulated genes KLK2 and KLK3 / HDMs demethylate histones / G2/M DNA damage checkpoint / NoRC negatively regulates rRNA expression / DNA Damage/Telomere Stress Induced Senescence / B-WICH complex positively regulates rRNA expression / PKMTs methylate histone lysines / Meiotic recombination / Pre-NOTCH Transcription and Translation / Metalloprotease DUBs / RMTs methylate histone arginines / Activation of anterior HOX genes in hindbrain development during early embryogenesis / Transcriptional regulation of granulopoiesis / HCMV Early Events / antimicrobial humoral immune response mediated by antimicrobial peptide / neuron projection development / kinase activity / structural constituent of chromatin / antibacterial humoral response / UCH proteinases / nucleosome / heterochromatin formation / E3 ubiquitin ligases ubiquitinate target proteins / nucleosome assembly / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / chromatin organization / HATs acetylate histones / RUNX1 regulates transcription of genes involved in differentiation of HSCs / Factors involved in megakaryocyte development and platelet production / MLL4 and MLL3 complexes regulate expression of PPARG target genes in adipogenesis and hepatic steatosis / Processing of DNA double-strand break ends / Senescence-Associated Secretory Phenotype (SASP) / protein autophosphorylation / Oxidative Stress Induced Senescence / Estrogen-dependent gene expression / histone binding / eukaryotic translation initiation factor 2alpha kinase activity / chromosome, telomeric region / 3-phosphoinositide-dependent protein kinase activity / DNA-dependent protein kinase activity / ribosomal protein S6 kinase activity / histone H3S10 kinase activity / histone H2AXS139 kinase activity / histone H3S28 kinase activity / histone H4S1 kinase activity / histone H2BS14 kinase activity / histone H3T3 kinase activity / histone H2AS121 kinase activity / Rho-dependent protein serine/threonine kinase activity / histone H2BS36 kinase activity / histone H3S57 kinase activity / histone H2AT120 kinase activity / AMP-activated protein kinase activity 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | ||||||||||||
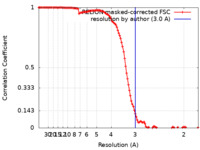
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.0 Å | ||||||||||||
 データ登録者 データ登録者 | Spangler CJ / Budziszewski GR | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2022 ジャーナル: Nucleic Acids Res / 年: 2022タイトル: Multivalent DNA and nucleosome acidic patch interactions specify VRK1 mitotic localization and activity. 著者: Gabrielle R Budziszewski / Yani Zhao / Cathy J Spangler / Katarzyna M Kedziora / Michael R Williams / Dalal N Azzam / Aleksandra Skrajna / Yuka Koyama / Andrew P Cesmat / Holly C Simmons / ...著者: Gabrielle R Budziszewski / Yani Zhao / Cathy J Spangler / Katarzyna M Kedziora / Michael R Williams / Dalal N Azzam / Aleksandra Skrajna / Yuka Koyama / Andrew P Cesmat / Holly C Simmons / Eyla C Arteaga / Joshua D Strauss / Dmitri Kireev / Robert K McGinty /  要旨: A key role of chromatin kinases is to phosphorylate histone tails during mitosis to spatiotemporally regulate cell division. Vaccinia-related kinase 1 (VRK1) is a serine-threonine kinase that ...A key role of chromatin kinases is to phosphorylate histone tails during mitosis to spatiotemporally regulate cell division. Vaccinia-related kinase 1 (VRK1) is a serine-threonine kinase that phosphorylates histone H3 threonine 3 (H3T3) along with other chromatin-based targets. While structural studies have defined how several classes of histone-modifying enzymes bind to and function on nucleosomes, the mechanism of chromatin engagement by kinases is largely unclear. Here, we paired cryo-electron microscopy with biochemical and cellular assays to demonstrate that VRK1 interacts with both linker DNA and the nucleosome acidic patch to phosphorylate H3T3. Acidic patch binding by VRK1 is mediated by an arginine-rich flexible C-terminal tail. Homozygous missense and nonsense mutations of this acidic patch recognition motif in VRK1 are causative in rare adult-onset distal spinal muscular atrophy. We show that these VRK1 mutations interfere with nucleosome acidic patch binding, leading to mislocalization of VRK1 during mitosis, thus providing a potential new molecular mechanism for pathogenesis. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25777.map.gz emd_25777.map.gz | 9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25777-v30.xml emd-25777-v30.xml emd-25777.xml emd-25777.xml | 21.1 KB 21.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_25777_fsc.xml emd_25777_fsc.xml | 11.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_25777.png emd_25777.png | 127.6 KB | ||
| Filedesc metadata |  emd-25777.cif.gz emd-25777.cif.gz | 7.1 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25777 http://ftp.pdbj.org/pub/emdb/structures/EMD-25777 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25777 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25777 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7tanMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25777.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25777.map.gz / 形式: CCP4 / 大きさ: 137.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Map of crosslinked VRK1-nucleosome complex | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.91 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Structure of VRK1 C-terminal tail bound to nucleosome core particle
+超分子 #1: Structure of VRK1 C-terminal tail bound to nucleosome core particle
+超分子 #2: Histone H3.2, Histone H4, Histone H2A type 1, Histone H2B type 1-...
+超分子 #3: DNA
+分子 #1: Histone H3.2
+分子 #2: Histone H4
+分子 #3: Histone H2A type 1
+分子 #4: Histone H2B type 1-C/E/F/G/I
+分子 #7: Serine/threonine-protein kinase VRK1
+分子 #5: WIDOM 601 DNA (185-MER)
+分子 #6: WIDOM 601 DNA (185-MER)
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE-PROPANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 8000 / 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)