+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the peroxisomal retro-translocon formed by a heterotrimeric ubiquitin ligase complex | |||||||||
 マップデータ マップデータ | Structure of the peroxisomal retro-translocon formed by a heterotrimeric ubiquitin ligase complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | peroxisome / retro-translocon / ubiquitin ligase / TRANSLOCASE | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報RING-type E3 ubiquitin transferase (cysteine targeting) / peroxisomal importomer complex / protein import into peroxisome matrix, receptor recycling / peroxisomal membrane / protein monoubiquitination / RING-type E3 ubiquitin transferase / ubiquitin-protein transferase activity / transferase activity / protein ubiquitination / zinc ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Thermothelomyces thermophilus ATCC 42464 (菌類) / synthetic construct (人工物) Thermothelomyces thermophilus ATCC 42464 (菌類) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å | |||||||||
 データ登録者 データ登録者 | Peiqiang F / Tom R | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2022 ジャーナル: Nature / 年: 2022タイトル: A peroxisomal ubiquitin ligase complex forms a retrotranslocation channel. 著者: Peiqiang Feng / Xudong Wu / Satchal K Erramilli / Joao A Paulo / Pawel Knejski / Steven P Gygi / Anthony A Kossiakoff / Tom A Rapoport /  要旨: Peroxisomes are ubiquitous organelles that house various metabolic reactions and are essential for human health. Luminal peroxisomal proteins are imported from the cytosol by mobile receptors, which ...Peroxisomes are ubiquitous organelles that house various metabolic reactions and are essential for human health. Luminal peroxisomal proteins are imported from the cytosol by mobile receptors, which then recycle back to the cytosol by a poorly understood process. Recycling requires receptor modification by a membrane-embedded ubiquitin ligase complex comprising three RING finger domain-containing proteins (Pex2, Pex10 and Pex12). Here we report a cryo-electron microscopy structure of the ligase complex, which together with biochemical and in vivo experiments reveals its function as a retrotranslocation channel for peroxisomal import receptors. Each subunit of the complex contributes five transmembrane segments that co-assemble into an open channel. The three ring finger domains form a cytosolic tower, with ring finger 2 (RF2) positioned above the channel pore. We propose that the N terminus of a recycling receptor is inserted from the peroxisomal lumen into the pore and monoubiquitylated by RF2 to enable extraction into the cytosol. If recycling is compromised, receptors are polyubiquitylated by the concerted action of RF10 and RF12 and degraded. This polyubiquitylation pathway also maintains the homeostasis of other peroxisomal import factors. Our results clarify a crucial step during peroxisomal protein import and reveal why mutations in the ligase complex cause human disease. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25750.map.gz emd_25750.map.gz | 62.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25750-v30.xml emd-25750-v30.xml emd-25750.xml emd-25750.xml | 17.4 KB 17.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_25750.png emd_25750.png | 55.8 KB | ||
| Filedesc metadata |  emd-25750.cif.gz emd-25750.cif.gz | 6.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25750 http://ftp.pdbj.org/pub/emdb/structures/EMD-25750 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25750 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25750 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25750_validation.pdf.gz emd_25750_validation.pdf.gz | 554.8 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25750_full_validation.pdf.gz emd_25750_full_validation.pdf.gz | 554.4 KB | 表示 | |
| XML形式データ |  emd_25750_validation.xml.gz emd_25750_validation.xml.gz | 6.1 KB | 表示 | |
| CIF形式データ |  emd_25750_validation.cif.gz emd_25750_validation.cif.gz | 6.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25750 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25750 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25750 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25750 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7t92MC  7t9xC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25750.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25750.map.gz / 形式: CCP4 / 大きさ: 67 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Structure of the peroxisomal retro-translocon formed by a heterotrimeric ubiquitin ligase complex | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||||||||||||||||||
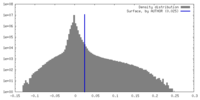
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : Peroxisomal heterotrimeric ubiquitin ligase complex bound with Fab
+超分子 #1: Peroxisomal heterotrimeric ubiquitin ligase complex bound with Fab
+超分子 #2: Peroxin12, Peroxin2, Peroxin10
+超分子 #3: Fab heavy chain, Fab light chain
+分子 #1: Peroxin-12
+分子 #2: Peroxin-2
+分子 #3: Peroxin-10
+分子 #4: Fab heavy chain
+分子 #5: Fab light chain
+分子 #6: ZINC ION
+分子 #7: 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphocholine
+分子 #8: CHOLESTEROL
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 4 K / 装置: FEI VITROBOT MARK I |
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 平均電子線量: 52.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.1 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 121644 |
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
 ムービー
ムービー コントローラー
コントローラー






 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)