+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of the HCMV Pentamer gH/gL/UL128/UL130/UL131A in complex with THBD and neutralizing fabs MSL-109 and 13H11 | |||||||||
 マップデータ マップデータ | Composite map obtained by combining focused, sharpened maps. Map used for model building and refinements. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Human Cytomegalovirus / glycoprotein complex / antibody complex / thrombomodulin / VIRAL PROTEIN / VIRAL PROTEIN-Immune System complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報blood coagulation, common pathway / apicolateral plasma membrane / serine-type endopeptidase complex / zymogen activation / vacuolar membrane / negative regulation of platelet activation / negative regulation of blood coagulation / negative regulation of fibrinolysis / response to cAMP / response to X-ray ...blood coagulation, common pathway / apicolateral plasma membrane / serine-type endopeptidase complex / zymogen activation / vacuolar membrane / negative regulation of platelet activation / negative regulation of blood coagulation / negative regulation of fibrinolysis / response to cAMP / response to X-ray / Common Pathway of Fibrin Clot Formation / Cell surface interactions at the vascular wall / female pregnancy / blood coagulation / transmembrane signaling receptor activity / signaling receptor activity / host cell endosome / response to lipopolysaccharide / host cell Golgi apparatus / entry receptor-mediated virion attachment to host cell / fusion of virus membrane with host plasma membrane / external side of plasma membrane / viral envelope / calcium ion binding / symbiont entry into host cell / host cell plasma membrane / virion membrane / cell surface / proteolysis / extracellular space / membrane / plasma membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Human betaherpesvirus 5 (ヘルペスウイルス) Human betaherpesvirus 5 (ヘルペスウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Kschonsak M / Johnson MC | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Structural basis for HCMV Pentamer receptor recognition and antibody neutralization. 著者: Marc Kschonsak / Matthew C Johnson / Rachel Schelling / Evan M Green / Lionel Rougé / Hoangdung Ho / Nidhi Patel / Cem Kilic / Edward Kraft / Christopher P Arthur / Alexis L Rohou / Laetitia ...著者: Marc Kschonsak / Matthew C Johnson / Rachel Schelling / Evan M Green / Lionel Rougé / Hoangdung Ho / Nidhi Patel / Cem Kilic / Edward Kraft / Christopher P Arthur / Alexis L Rohou / Laetitia Comps-Agrar / Nadia Martinez-Martin / Laurent Perez / Jian Payandeh / Claudio Ciferri /   要旨: Human cytomegalovirus (HCMV) represents the viral leading cause of congenital birth defects and uses the gH/gL/UL128-130-131A complex (Pentamer) to enter different cell types, including epithelial ...Human cytomegalovirus (HCMV) represents the viral leading cause of congenital birth defects and uses the gH/gL/UL128-130-131A complex (Pentamer) to enter different cell types, including epithelial and endothelial cells. Upon infection, Pentamer elicits the most potent neutralizing response against HCMV, representing a key vaccine candidate. Despite its relevance, the structural basis for Pentamer receptor recognition and antibody neutralization is largely unknown. Here, we determine the structures of Pentamer bound to neuropilin 2 (NRP2) and a set of potent neutralizing antibodies against HCMV. Moreover, we identify thrombomodulin (THBD) as a functional HCMV receptor and determine the structures of the Pentamer-THBD complex. Unexpectedly, both NRP2 and THBD also promote dimerization of Pentamer. Our results provide a framework for understanding HCMV receptor engagement, cell entry, antibody neutralization, and outline strategies for antiviral therapies against HCMV. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_25686.map.gz emd_25686.map.gz | 13 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-25686-v30.xml emd-25686-v30.xml emd-25686.xml emd-25686.xml | 59.3 KB 59.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
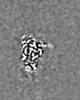
| 画像 |  emd_25686.png emd_25686.png | 83.3 KB | ||
| Filedesc metadata |  emd-25686.cif.gz emd-25686.cif.gz | 9.7 KB | ||
| その他 |  emd_25686_additional_1.map.gz emd_25686_additional_1.map.gz emd_25686_additional_10.map.gz emd_25686_additional_10.map.gz emd_25686_additional_11.map.gz emd_25686_additional_11.map.gz emd_25686_additional_12.map.gz emd_25686_additional_12.map.gz emd_25686_additional_13.map.gz emd_25686_additional_13.map.gz emd_25686_additional_14.map.gz emd_25686_additional_14.map.gz emd_25686_additional_15.map.gz emd_25686_additional_15.map.gz emd_25686_additional_16.map.gz emd_25686_additional_16.map.gz emd_25686_additional_2.map.gz emd_25686_additional_2.map.gz emd_25686_additional_3.map.gz emd_25686_additional_3.map.gz emd_25686_additional_4.map.gz emd_25686_additional_4.map.gz emd_25686_additional_5.map.gz emd_25686_additional_5.map.gz emd_25686_additional_6.map.gz emd_25686_additional_6.map.gz emd_25686_additional_7.map.gz emd_25686_additional_7.map.gz emd_25686_additional_8.map.gz emd_25686_additional_8.map.gz emd_25686_additional_9.map.gz emd_25686_additional_9.map.gz | 226.2 MB 165.7 MB 165.7 MB 8.2 MB 226.4 MB 165.7 MB 165.7 MB 12.3 MB 165.5 MB 13.7 MB 165.6 MB 226.3 MB 165.6 MB 165.7 MB 8.5 MB 226.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-25686 http://ftp.pdbj.org/pub/emdb/structures/EMD-25686 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25686 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-25686 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_25686_validation.pdf.gz emd_25686_validation.pdf.gz | 374 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_25686_full_validation.pdf.gz emd_25686_full_validation.pdf.gz | 373.6 KB | 表示 | |
| XML形式データ |  emd_25686_validation.xml.gz emd_25686_validation.xml.gz | 7 KB | 表示 | |
| CIF形式データ |  emd_25686_validation.cif.gz emd_25686_validation.cif.gz | 8.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25686 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25686 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25686 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-25686 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7t4rMC  7t4qC  7t4sC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_25686.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_25686.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Composite map obtained by combining focused, sharpened maps. Map used for model building and refinements. | ||||||||||||||||||||||||||||||||||||
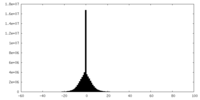
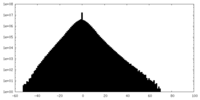
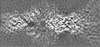
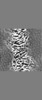
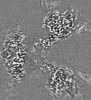
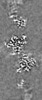
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
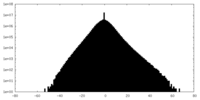
| ボクセルのサイズ | X=Y=Z: 1.0726 Å | ||||||||||||||||||||||||||||||||||||
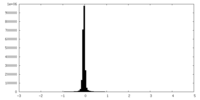
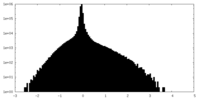
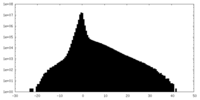
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: Non-sharpened, full map of overall map (not focused).
+追加マップ: Half map 1 of focused gH-gL (A) region.
+追加マップ: Half map 2 of focused gH-gL (A) region.
+追加マップ: Density modified map of focused gH-gL (A) map...
+追加マップ: Non-sharpened, full map of focused gH-gL (B) region.
+追加マップ: Half map 1 of focused gH-gL (B) region.
+追加マップ: Half map 2 of focused gH-gL (B) region.
+追加マップ: Density modified map of overall map (not focused)...
+追加マップ: Half map 1 of overall map (not focused).
+追加マップ: Density modified map of focused THBD-ULs map used...
+追加マップ: Half map 2 of overall map (not focused).
+追加マップ: Non-sharpened, full map of focused THBD-ULs region.
+追加マップ: Half map 1 of focused THBD-ULs region.
+追加マップ: Half map 2 of focused THBD-ULs region.
+追加マップ: Density modified map of focused gH-gL (B) map...
+追加マップ: Non-sharpened, full map of focused gH-gL (A) region.
- 試料の構成要素
試料の構成要素
+全体 : Complex of 2x HCMV Pentamer gH, gL, UL128, UL130, UL131A bound to...
+超分子 #1: Complex of 2x HCMV Pentamer gH, gL, UL128, UL130, UL131A bound to...
+分子 #1: Thrombomodulin
+分子 #2: Envelope glycoprotein H
+分子 #3: Envelope glycoprotein L
+分子 #4: Envelope protein UL128
+分子 #5: Envelope glycoprotein UL130
+分子 #6: Envelope protein UL131A
+分子 #7: Fab 13H11 heavy chain
+分子 #8: Fab 13H11 light chain
+分子 #9: Fab MSL-109 light chain
+分子 #10: Fab MSL-109 heavy chain
+分子 #11: 2-acetamido-2-deoxy-beta-D-glucopyranose
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.6 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||
| グリッド | モデル: UltrAuFoil R0.6/1 / 材質: GOLD / メッシュ: 300 詳細: The grid was incubated with a thiol reactive self-assembling reaction mixture of 4mM monothiolalkane(C11)PEG6-OH (11-mercaptoundecyl) hexaethyleneglycol, (SPT-0011P6, SensoPath Technologies, ...詳細: The grid was incubated with a thiol reactive self-assembling reaction mixture of 4mM monothiolalkane(C11)PEG6-OH (11-mercaptoundecyl) hexaethyleneglycol, (SPT-0011P6, SensoPath Technologies, Inc., Bozeman, MT)[23]. Grids were incubated with this self-assembled, monolayer (SAM) solution for 24 hours. Prior to grid freezing, grids were removed from the SAM solution and rinsed with EtOH. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: LEICA EM GP / 詳細: blot for 3.5s before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 10926 / 平均電子線量: 64.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー


















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)