+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2565 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM of SecA-70S complex | |||||||||
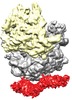
 マップデータ マップデータ | reconstruction of monomeric SecA bound to 70S ribosome | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | SecA / ribosome | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 10.3 Å | |||||||||
 データ登録者 データ登録者 | Singh R / Kraft C / Jaiswal R / Sejwal K / Kasaragod V / Kuper J / Buerger J / Mielke T / Luirink J / Bhushan S | |||||||||
 引用 引用 |  ジャーナル: J Biol Chem / 年: 2014 ジャーナル: J Biol Chem / 年: 2014タイトル: Cryo-electron microscopic structure of SecA protein bound to the 70S ribosome. 著者: Rajkumar Singh / Christian Kraft / Rahul Jaiswal / Kushal Sejwal / Vikram Babu Kasaragod / Jochen Kuper / Jörg Bürger / Thorsten Mielke / Joen Luirink / Shashi Bhushan /    要旨: SecA is an ATP-dependent molecular motor pumping secretory and outer membrane proteins across the cytoplasmic membrane in bacteria. SecA associates with the protein-conducting channel, the ...SecA is an ATP-dependent molecular motor pumping secretory and outer membrane proteins across the cytoplasmic membrane in bacteria. SecA associates with the protein-conducting channel, the heterotrimeric SecYEG complex, in a so-called posttranslational manner. A recent study further showed binding of a monomeric state of SecA to the ribosome. However, the true oligomeric state of SecA remains controversial because SecA can also form functional dimers, and high-resolution crystal structures exist for both the monomer and the dimer. Here we present the cryo-electron microscopy structures of Escherichia coli SecA bound to the ribosome. We show that not only a monomeric SecA binds to the ribosome but also that two copies of SecA can be observed that form an elongated dimer. Two copies of SecA completely surround the tunnel exit, providing a unique environment to the nascent polypeptides emerging from the ribosome. We identified the N-terminal helix of SecA required for a stable association with the ribosome. The structures indicate a possible function of the dimeric form of SecA at the ribosome. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2565.map.gz emd_2565.map.gz | 5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2565-v30.xml emd-2565-v30.xml emd-2565.xml emd-2565.xml | 9.9 KB 9.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  EMD-2565-dimerr.tif EMD-2565-dimerr.tif | 902.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2565 http://ftp.pdbj.org/pub/emdb/structures/EMD-2565 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2565 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2565 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2565_validation.pdf.gz emd_2565_validation.pdf.gz | 219.7 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2565_full_validation.pdf.gz emd_2565_full_validation.pdf.gz | 218.8 KB | 表示 | |
| XML形式データ |  emd_2565_validation.xml.gz emd_2565_validation.xml.gz | 7.2 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2565 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2565 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2565 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2565 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2565.map.gz / 形式: CCP4 / 大きさ: 94.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2565.map.gz / 形式: CCP4 / 大きさ: 94.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | reconstruction of monomeric SecA bound to 70S ribosome | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.24 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : SecA-70S ribosome
| 全体 | 名称: SecA-70S ribosome |
|---|---|
| 要素 |
|
-超分子 #1000: SecA-70S ribosome
| 超分子 | 名称: SecA-70S ribosome / タイプ: sample / ID: 1000 / 集合状態: Monomeric SecA bound to the 70S ribosome / Number unique components: 2 |
|---|---|
| 分子量 | 実験値: 2 MDa / 理論値: 2 MDa |
-超分子 #1: 70S ribosome
| 超分子 | 名称: 70S ribosome / タイプ: complex / ID: 1 / Name.synonym: 70S / 組換発現: No / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 2 MDa / 理論値: 2 MDa |
-超分子 #2: SecA
| 超分子 | 名称: SecA / タイプ: complex / ID: 2 / Name.synonym: SecA / 組換発現: No / Ribosome-details: ribosome-prokaryote: ALL |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 100 KDa / 理論値: 100 KDa |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 詳細: 40 mM Hepes pH 7.6, 50 mM K-acetate, 25 mM Mg-Acetate, 5mM DTT, 0.1% protease inhibitor pill/ml, 0.1 U/ml RNAsin, 125 mM sucrose |
|---|---|
| グリッド | 詳細: 2-nm carbon-coated holey grids |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / 装置: FEI VITROBOT MARK III / 手法: Blot for 5 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI POLARA 300 |
|---|---|
| 日付 | 2011年6月11日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK 4489 FILM / デジタル化 - スキャナー: OTHER / デジタル化 - サンプリング間隔: 0.25 µm / 実像数: 300 / 平均電子線量: 25 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 39000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
- 画像解析
画像解析
| 詳細 | Spider |
|---|---|
| CTF補正 | 詳細: Each Micrographs |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 10.3 Å / 解像度の算出法: OTHER / ソフトウェア - 名称: Spider / 使用した粒子像数: 83000 |
| 最終 角度割当 | 詳細: Spider |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | The domains were separately fitted by manual docking using coot and chimera |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)






















