+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2558 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative-stain electron microscopy of E. coli ClpB (BAP form bound to ClpP) | |||||||||
 マップデータ マップデータ | Asymmetric reconstruction of wild-type E.coli ClpB (BAP form bound to ClpP) | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | chaperone / disaggregase / ClpB / BAP / wild type / asymmetric / coiled-coil domain | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 21.0 Å | |||||||||
 データ登録者 データ登録者 | Carroni M / Kummer E / Oguchi Y / Clare DK / Wendler P / Sinning I / Kopp J / Mogk A / Bukau B / Saibil HR | |||||||||
 引用 引用 |  ジャーナル: Elife / 年: 2014 ジャーナル: Elife / 年: 2014タイトル: Head-to-tail interactions of the coiled-coil domains regulate ClpB activity and cooperation with Hsp70 in protein disaggregation. 著者: Marta Carroni / Eva Kummer / Yuki Oguchi / Petra Wendler / Daniel K Clare / Irmgard Sinning / Jürgen Kopp / Axel Mogk / Bernd Bukau / Helen R Saibil /   要旨: The hexameric AAA+ chaperone ClpB reactivates aggregated proteins in cooperation with the Hsp70 system. Essential for disaggregation, the ClpB middle domain (MD) is a coiled-coil propeller that binds ...The hexameric AAA+ chaperone ClpB reactivates aggregated proteins in cooperation with the Hsp70 system. Essential for disaggregation, the ClpB middle domain (MD) is a coiled-coil propeller that binds Hsp70. Although the ClpB subunit structure is known, positioning of the MD in the hexamer and its mechanism of action are unclear. We obtained electron microscopy (EM) structures of the BAP variant of ClpB that binds the protease ClpP, clearly revealing MD density on the surface of the ClpB ring. Mutant analysis and asymmetric reconstructions show that MDs adopt diverse positions in a single ClpB hexamer. Adjacent, horizontally oriented MDs form head-to-tail contacts and repress ClpB activity by preventing Hsp70 interaction. Tilting of the MD breaks this contact, allowing Hsp70 binding, and releasing the contact in adjacent subunits. Our data suggest a wavelike activation of ClpB subunits around the ring.DOI: http://dx.doi.org/10.7554/eLife.02481.001. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2558.map.gz emd_2558.map.gz | 61.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2558-v30.xml emd-2558-v30.xml emd-2558.xml emd-2558.xml | 11.5 KB 11.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_2558_fsc.xml emd_2558_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_2558.tiff emd_2558.tiff | 396.3 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2558 http://ftp.pdbj.org/pub/emdb/structures/EMD-2558 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2558 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2558 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2558_validation.pdf.gz emd_2558_validation.pdf.gz | 216.4 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2558_full_validation.pdf.gz emd_2558_full_validation.pdf.gz | 215.5 KB | 表示 | |
| XML形式データ |  emd_2558_validation.xml.gz emd_2558_validation.xml.gz | 10.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2558 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2558 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2558 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2558 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2558.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2558.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Asymmetric reconstruction of wild-type E.coli ClpB (BAP form bound to ClpP) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : ClpB ATPgammaS. BAP variant bound to ClpP.
| 全体 | 名称: ClpB ATPgammaS. BAP variant bound to ClpP. |
|---|---|
| 要素 |
|
-超分子 #1000: ClpB ATPgammaS. BAP variant bound to ClpP.
| 超分子 | 名称: ClpB ATPgammaS. BAP variant bound to ClpP. / タイプ: sample / ID: 1000 詳細: Only the ClpB part was reconstructed and the molecular weight only refers to this part. 集合状態: Homohexamer. (One homohexamer of BAP bound to one homoheptamer of ClpP) Number unique components: 1 |
|---|---|
| 分子量 | 理論値: 500 KDa |
-分子 #1: ClpB
| 分子 | 名称: ClpB / タイプ: protein_or_peptide / ID: 1 / 詳細: The protein is engineered to bind to ClpP. / コピー数: 6 / 集合状態: Hexamer / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 実験値: 80 KDa / 理論値: 80 KDa |
| 組換発現 | 生物種:  |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 詳細: 20 mM Tris-HCl, pH 7.5, 20 mM KCl, 15 mM MgCl2, 1 mM DTT, 2 mM ATPgammaS |
| 染色 | タイプ: NEGATIVE 詳細: protein adsorbed on carbon coated grids pretreated with 0.01% poly lysine chains. Stained with 2% uranyl acetate for 1 minute. |
| 凍結 | 凍結剤: NONE / 装置: OTHER |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| アライメント法 | Legacy - 非点収差: Objective lens astigmatism was corrected at 150,000 x magnification |
| 日付 | 2012年3月7日 |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 実像数: 220 / 平均電子線量: 20 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 68000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2 mm / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 50000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Fitting of separate domains was performed manually and locally optimised using Chimera. Known domain interfaces were used to guide the fit. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: Chain - Chain ID: A |
|---|---|
| ソフトウェア | 名称:  Chimera Chimera |
| 詳細 | Fitting of separate domains was performed manually and locally optimised using Chimera. Known domain interfaces were used to guide the fit. |
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
 ムービー
ムービー コントローラー
コントローラー







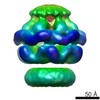
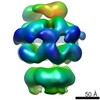










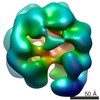

 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)























