+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human Nuclear exosome targeting (NEXT) complex homodimer bound to RNA (substrate 1) | |||||||||
 マップデータ マップデータ | human NEXT dimer bound to RNA - composite structure | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Helicase / ATPase / RNA / Exosome / RNA BINDING PROTEIN / RNA BINDING PROTEIN-RNA complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報co-transcriptional lncRNA 3' end processing, cleavage and polyadenylation pathway / snRNA catabolic process / TRAMP complex / snRNA binding / mRNA 3'-end processing / RNA catabolic process / maturation of 5.8S rRNA / regulation of alternative mRNA splicing, via spliceosome / pre-mRNA intronic binding / Major pathway of rRNA processing in the nucleolus and cytosol ...co-transcriptional lncRNA 3' end processing, cleavage and polyadenylation pathway / snRNA catabolic process / TRAMP complex / snRNA binding / mRNA 3'-end processing / RNA catabolic process / maturation of 5.8S rRNA / regulation of alternative mRNA splicing, via spliceosome / pre-mRNA intronic binding / Major pathway of rRNA processing in the nucleolus and cytosol / RNA processing / 14-3-3 protein binding / catalytic step 2 spliceosome / mRNA Splicing - Major Pathway / Regulation of endogenous retroelements by the Human Silencing Hub (HUSH) complex / meiotic cell cycle / mRNA splicing, via spliceosome / rRNA processing / single-stranded RNA binding / RNA helicase activity / nuclear speck / nuclear body / RNA helicase / DNA damage response / nucleolus / ATP hydrolysis activity / RNA binding / zinc ion binding / nucleoplasm / ATP binding / nucleus 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.06 Å | |||||||||
 データ登録者 データ登録者 | Puno MR / Lima CD | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2022 ジャーナル: Cell / 年: 2022タイトル: Structural basis for RNA surveillance by the human nuclear exosome targeting (NEXT) complex. 著者: M Rhyan Puno / Christopher D Lima /  要旨: RNA quality control relies on co-factors and adaptors to identify and prepare substrates for degradation by ribonucleases such as the 3' to 5' ribonucleolytic RNA exosome. Here, we determined ...RNA quality control relies on co-factors and adaptors to identify and prepare substrates for degradation by ribonucleases such as the 3' to 5' ribonucleolytic RNA exosome. Here, we determined cryogenic electron microscopy structures of human nuclear exosome targeting (NEXT) complexes bound to RNA that reveal mechanistic insights to substrate recognition and early steps that precede RNA handover to the exosome. The structures illuminate ZCCHC8 as a scaffold, mediating homodimerization while embracing the MTR4 helicase and flexibly anchoring RBM7 to the helicase core. All three subunits collaborate to bind the RNA, with RBM7 and ZCCHC8 surveying sequences upstream of the 3' end to facilitate RNA capture by MTR4. ZCCHC8 obscures MTR4 surfaces important for RNA binding and extrusion as well as MPP6-dependent recruitment and docking onto the RNA exosome core, interactions that contribute to RNA surveillance by coordinating RNA capture, translocation, and extrusion from the helicase to the exosome for decay. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24882.map.gz emd_24882.map.gz | 19.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24882-v30.xml emd-24882-v30.xml emd-24882.xml emd-24882.xml | 53.9 KB 53.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24882.png emd_24882.png | 60.6 KB | ||
| Filedesc metadata |  emd-24882.cif.gz emd-24882.cif.gz | 8.2 KB | ||
| その他 |  emd_24882_additional_1.map.gz emd_24882_additional_1.map.gz emd_24882_additional_10.map.gz emd_24882_additional_10.map.gz emd_24882_additional_11.map.gz emd_24882_additional_11.map.gz emd_24882_additional_12.map.gz emd_24882_additional_12.map.gz emd_24882_additional_13.map.gz emd_24882_additional_13.map.gz emd_24882_additional_14.map.gz emd_24882_additional_14.map.gz emd_24882_additional_15.map.gz emd_24882_additional_15.map.gz emd_24882_additional_16.map.gz emd_24882_additional_16.map.gz emd_24882_additional_17.map.gz emd_24882_additional_17.map.gz emd_24882_additional_18.map.gz emd_24882_additional_18.map.gz emd_24882_additional_2.map.gz emd_24882_additional_2.map.gz emd_24882_additional_3.map.gz emd_24882_additional_3.map.gz emd_24882_additional_4.map.gz emd_24882_additional_4.map.gz emd_24882_additional_5.map.gz emd_24882_additional_5.map.gz emd_24882_additional_6.map.gz emd_24882_additional_6.map.gz emd_24882_additional_7.map.gz emd_24882_additional_7.map.gz emd_24882_additional_8.map.gz emd_24882_additional_8.map.gz emd_24882_additional_9.map.gz emd_24882_additional_9.map.gz | 171.1 MB 172 MB 172 MB 171.3 MB 171.7 MB 171.5 MB 171.2 MB 171.4 MB 10.6 MB 171.8 MB 11.4 MB 11.8 MB 11.1 MB 171.7 MB 171.3 MB 171.5 MB 15.6 MB 11.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24882 http://ftp.pdbj.org/pub/emdb/structures/EMD-24882 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24882 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24882 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24882_validation.pdf.gz emd_24882_validation.pdf.gz | 340.6 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24882_full_validation.pdf.gz emd_24882_full_validation.pdf.gz | 340.2 KB | 表示 | |
| XML形式データ |  emd_24882_validation.xml.gz emd_24882_validation.xml.gz | 7.3 KB | 表示 | |
| CIF形式データ |  emd_24882_validation.cif.gz emd_24882_validation.cif.gz | 8.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24882 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24882 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24882 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24882 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24882.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24882.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | human NEXT dimer bound to RNA - composite structure | ||||||||||||||||||||||||||||||||||||
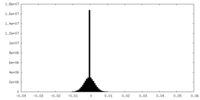
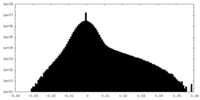
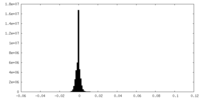
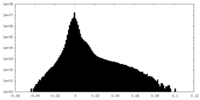
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.088 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
+追加マップ: focused refinement on protomer A MTR4 core, ZCCHC8...
+追加マップ: focused refinement on ZCCHC8-HD/KID MTR4-KOW half map 1
+追加マップ: focused refinement on ZCCHC8-HD/KID MTR4-KOW half map 2
+追加マップ: focused refinement on protomer A MTR4 half map 1
+追加マップ: Overall reconstruction half map 1
+追加マップ: focused refinement on protomer B MTR4 core, ZCCHC8...
+追加マップ: Overall reconstruction half map 2
+追加マップ: focused refinement on protomer A MTR4 core, ZCCHC8...
+追加マップ: focused refinement on protomer B MTR4 core, ZCCHC8...
+追加マップ: focused refinement on protomer B MTR4 core, ZCCHC8...
+追加マップ: focused refinement on protomer B MTR4 (component map...
+追加マップ: focused refinement on ZCCHC8-HD/KID MTR4-KOW (component map of...
+追加マップ: focused refinement on protomer A MTR4 core, ZCCHC8...
+追加マップ: focused refinement on protomer B MTR4 half map 1
+追加マップ: focused refinement on protomer A MTR4 half map 2
+追加マップ: focused refinement on protomer B MTR4 half map 2
+追加マップ: Overall reconstruction (component map of composite structure)
+追加マップ: focused refinement on protomer A MTR4 (component map...
- 試料の構成要素
試料の構成要素
-全体 : human Nuclear Exosome Targeting (NEXT)-RNA substrate 1 complex
| 全体 | 名称: human Nuclear Exosome Targeting (NEXT)-RNA substrate 1 complex |
|---|---|
| 要素 |
|
-超分子 #1: human Nuclear Exosome Targeting (NEXT)-RNA substrate 1 complex
| 超分子 | 名称: human Nuclear Exosome Targeting (NEXT)-RNA substrate 1 complex タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#4 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Exosome RNA helicase MTR4
| 分子 | 名称: Exosome RNA helicase MTR4 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号: RNA helicase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 118.224961 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGDMADAFGD ELFSVFEGDS TTAAGTKKDK EKDKGKWKGP PGSADKAGKR FDGKLQSEST NNGKNKRDVD FEGTDEPIFG KKPRIEESI TEDLSLADLM PRVKVQSVET VEGCTHEVAL PAEEDYLPLK PRVGKAAKEY PFILDAFQRE AIQCVDNNQS V LVSAHTSA ...文字列: SGDMADAFGD ELFSVFEGDS TTAAGTKKDK EKDKGKWKGP PGSADKAGKR FDGKLQSEST NNGKNKRDVD FEGTDEPIFG KKPRIEESI TEDLSLADLM PRVKVQSVET VEGCTHEVAL PAEEDYLPLK PRVGKAAKEY PFILDAFQRE AIQCVDNNQS V LVSAHTSA GKTVCAEYAI ALALREKQRV IFTSPIKALS NQKYREMYEE FQDVGLMTGD VTINPTASCL VMTTEILRSM LY RGSEVMR EVAWVIFDEI HYMRDSERGV VWEETIILLP DNVHYVFLSA TIPNARQFAE WICHLHKQPC HVIYTDYRPT PLQ HYIFPA GGDGLHLVVD ENGDFREDNF NTAMQVLRDA GDLAKGDQKG RKGGTKGPSN VFKIVKMIME RNFQPVIIFS FSKK DCEAY ALQMTKLDFN TDEEKKMVEE VFSNAIDCLS DEDKKLPQVE HVLPLLKRGI GIHHGGLLPI LKETIEILFS EGLIK ALFA TETFAMGINM PARTVLFTNA RKFDGKDFRW ISSGEYIQMS GRAGRRGMDD RGIVILMVDE KMSPTIGKQL LKGSAD PLN SAFHLTYNMV LNLLRVEEIN PEYMLEKSFY QFQHYRAIPG VVEKVKNSEE QYNKIVIPNE ESVVIYYKIR QQLAKLG KE IEEYIHKPKY CLPFLQPGRL VKVKNEGDDF GWGVVVNFSK KSNVKPNSGE LDPLYVVEVL LRCSKESLKN SATEAAKP A KPDEKGEMQV VPVLVHLLSA ISSVRLYIPK DLRPVDNRQS VLKSIQEVQK RFPDGIPLLD PIDDMGIQDQ GLKKVIQKV EAFEHRMYSH PLHNDPNLET VYTLCEKKAQ IAIDIKSAKR ELKKARTVLQ MDELKCRKRV LRRLGFATSS DVIEMKGRVA CEISSADEL LLTEMMFNGL FNDLSAEQAT ALLSCFVFQE NSSEMPKLTE QLAGPLRQMQ ECAKRIAKVS AEAKLEIDEE T YLSSFKPH LMDVVYTWAT GATFAHICKM TDVFEGSIIR CMRRLEELLR QMCQAAKAIG NTELENKFAE GITKIKRDIV FA ASLYL UniProtKB: Exosome RNA helicase MTR4 |
-分子 #2: Zinc finger CCHC domain-containing protein 8,Zinc finger CCHC dom...
| 分子 | 名称: Zinc finger CCHC domain-containing protein 8,Zinc finger CCHC domain-containing protein 8 タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 69.259805 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGDMAAEVYF GDLELFEPFD HPEESIPKPV HTRFKDDDGD EEDENGVGDA ELRERLRQCE ETIEQLRAEN QELKRKLNIL TRPSGILVN DTKLDGPILQ ILFMNNAISK QYHQEIEEFV SNLVKRFEEQ QKNDVEKTSF NLLPQPSSIV LEEDHKVEES C AIKNNKEA ...文字列: SGDMAAEVYF GDLELFEPFD HPEESIPKPV HTRFKDDDGD EEDENGVGDA ELRERLRQCE ETIEQLRAEN QELKRKLNIL TRPSGILVN DTKLDGPILQ ILFMNNAISK QYHQEIEEFV SNLVKRFEEQ QKNDVEKTSF NLLPQPSSIV LEEDHKVEES C AIKNNKEA FSVVGSVLYF TNFCLDKLGQ PLLNENPQLS EGWEIPKYHQ VFSHIVSLEG QEIQVKAKRP KPHCFNCGSE EH QMKDCPM PRNAARISEK RKEYMDACGE ANNQNFQQRY HAEEVEERFG RFKPGVISEE LQDALGVTDK SLPPFIYRMR QLG YPPGWL KEAELENSGL ALYDGKDGTD GETEVGEIQQ NKSVTYDLSK LVNYPGFNIS TPRGIPDEWR IFGSIPMQAC QQKD VFANY LTSNFQAPGV KSGGAVDEDA LTLEELEEQQ RRIWAALEQA ESVNSDSDVP VDTPLTGNSV ASSPCPNELD LPVPE GKTS EKQTLDEPEV PEIFTKKSEA GHASSPDSEV TSLCQKEKAE LAPVNTEGAL LDNGSVVPNC DISNGGSQKL FPADTS PST ATKIHSPIPD MSKFATGITP FEFENMAEST GMYLRIRSLL KNSPRNQQKN KKASE UniProtKB: Zinc finger CCHC domain-containing protein 8, Zinc finger CCHC domain-containing protein 8 |
-分子 #3: RNA-binding protein 7
| 分子 | 名称: RNA-binding protein 7 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 9.462986 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: SGDEADRTLF VGNLETKVTE ELLFELFHQA GPVIKVKIPK DKDGKPKQFA FVNFKHEVSV PYAMNLLNGI KLYGRPIKIQ FRS UniProtKB: RNA-binding protein 7 |
-分子 #4: RNA (46-MER)
| 分子 | 名称: RNA (46-MER) / タイプ: rna / ID: 4 詳細: The actual RNA sequence is: ACAUGAGGAUCACCCAUGUAAUCUCUUUCAAAAAA(2PU)ACAAAAAAAA. (2PU) is represented by "N" (any nucleotide. This residue is missing in the coordinates and the chemical is an ...詳細: The actual RNA sequence is: ACAUGAGGAUCACCCAUGUAAUCUCUUUCAAAAAA(2PU)ACAAAAAAAA. (2PU) is represented by "N" (any nucleotide. This residue is missing in the coordinates and the chemical is an internal 2' pyrene modified uridine コピー数: 2 |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 14.605818 KDa |
| 配列 | 文字列: ACAUGAGGAU CACCCAUGUA AUCUCUUUCA AAAAA(N)ACAA AAAAAA |
-分子 #5: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 5 / コピー数: 2 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 8 mg/mL |
|---|---|
| 緩衝液 | pH: 8 詳細: 20 mM Tris-Cl pH 8.0, 50 mM NaCl, 0.1 mM TCEP supplemented with 0.02% (v/v) IGEPAL CA-630 |
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV / 詳細: 30 s wait time, blot for 2.5 s before plunging. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 平均電子線量: 67.6 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: Ab initio model from cryosparc |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.06 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION 詳細: A total of 618,412 particles were used for the consensus reconstruction with an overall resolution of 4.06 Angstrom (FSC 0.143 cut off). Focused 3D classification and local refinement were ...詳細: A total of 618,412 particles were used for the consensus reconstruction with an overall resolution of 4.06 Angstrom (FSC 0.143 cut off). Focused 3D classification and local refinement were performed in several regions of the complex. A composite map was generated using focused reconstructions of ZCCHC8 HD/KID-MTR4 KOW (3.26 Angstrom, FSC = 0.143; 117,561 particles), protomer A MTR4 (3.42 Angstrom, FSC = 0.143; 225,213 particles), protomer B MTR4 (3.54 Angstrom, FSC = 0.143; 236,602 particles), protomer A MTR4 core-ZCCHC8 PSP-RBM7 RRM (4.06 Angstrom, FSC = 0.143; 44,800 particles), and protomer B MTR4 core-ZCCHC8 PSP-RBM7 RRM (4.4 Angstrom, FSC = 0.143 37,088 particles). 使用した粒子像数: 618412 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー














 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)