+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24620 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Negative stain electron microscopy single particle reconstruction of monoclonal antibody DH1025.1 Fab in complex with HIV-1 Env CH505 SOSIP trimer | |||||||||
 マップデータ マップデータ | Final sharpened map of negative stain electron microscopy single particle reconstruction of monoclonal antibody DH1025.1 Fab in complex with HIV-1 Env CH505 SOSIP trimer. | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報membrane fusion involved in viral entry into host cell / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell ...membrane fusion involved in viral entry into host cell / positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human immunodeficiency virus 2 (ヒト免疫不全ウイルス) / Human immunodeficiency virus 2 (ヒト免疫不全ウイルス) /    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
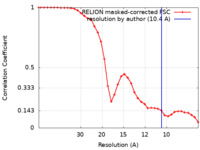
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 10.4 Å | |||||||||
 データ登録者 データ登録者 | Edwards RJ / Mansouri K | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Transl Med / 年: 2022 ジャーナル: Sci Transl Med / 年: 2022タイトル: Stabilized HIV-1 envelope immunization induces neutralizing antibodies to the CD4bs and protects macaques against mucosal infection. 著者: Kevin O Saunders / Robert J Edwards / Kedamawit Tilahun / Kartik Manne / Xiaozhi Lu / Derek W Cain / Kevin Wiehe / Wilton B Williams / Katayoun Mansouri / Giovanna E Hernandez / Laura ...著者: Kevin O Saunders / Robert J Edwards / Kedamawit Tilahun / Kartik Manne / Xiaozhi Lu / Derek W Cain / Kevin Wiehe / Wilton B Williams / Katayoun Mansouri / Giovanna E Hernandez / Laura Sutherland / Richard Scearce / Robert Parks / Maggie Barr / Todd DeMarco / Chloe M Eater / Amanda Eaton / Georgeanna Morton / Benjamin Mildenberg / Yunfei Wang / R Wes Rountree / Mark A Tomai / Christopher B Fox / M Anthony Moody / S Munir Alam / Sampa Santra / Mark G Lewis / Thomas N Denny / George M Shaw / David C Montefiori / Priyamvada Acharya / Barton F Haynes /  要旨: A successful HIV-1 vaccine will require induction of a polyclonal neutralizing antibody (nAb) response, yet vaccine-mediated induction of such a response in primates remains a challenge. We found ...A successful HIV-1 vaccine will require induction of a polyclonal neutralizing antibody (nAb) response, yet vaccine-mediated induction of such a response in primates remains a challenge. We found that a stabilized HIV-1 CH505 envelope (Env) trimer formulated with a Toll-like receptor 7/8 agonist induced potent HIV-1 polyclonal nAbs that correlated with protection from homologous simian-human immunodeficiency virus (SHIV) infection. The serum dilution that neutralized 50% of virus replication (ID titer) required to protect 90% of macaques was 1:364 against the challenge virus grown in primary rhesus CD4 T cells. Structural analyses of vaccine-induced nAbs demonstrated targeting of the Env CD4 binding site or the N156 glycan and the third variable loop base. Autologous nAb specificities similar to those elicited in macaques by vaccination were isolated from the human living with HIV from which the CH505 Env immunogen was derived. CH505 viral isolates were isolated that mutated the V1 to escape both the infection-induced and vaccine-induced antibodies. These results define the specificities of a vaccine-induced nAb response and the protective titers of HIV-1 vaccine-induced nAbs required to protect nonhuman primates from low-dose mucosal challenge by SHIVs bearing a primary transmitted/founder Env. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24620.map.gz emd_24620.map.gz | 3.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24620-v30.xml emd-24620-v30.xml emd-24620.xml emd-24620.xml | 31.7 KB 31.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24620_fsc.xml emd_24620_fsc.xml | 3.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24620.png emd_24620.png | 77.2 KB | ||
| その他 |  emd_24620_additional_1.map.gz emd_24620_additional_1.map.gz emd_24620_half_map_1.map.gz emd_24620_half_map_1.map.gz emd_24620_half_map_2.map.gz emd_24620_half_map_2.map.gz | 2.4 MB 2.4 MB 2.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24620 http://ftp.pdbj.org/pub/emdb/structures/EMD-24620 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24620 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24620 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24620_validation.pdf.gz emd_24620_validation.pdf.gz | 489.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24620_full_validation.pdf.gz emd_24620_full_validation.pdf.gz | 488.6 KB | 表示 | |
| XML形式データ |  emd_24620_validation.xml.gz emd_24620_validation.xml.gz | 9.1 KB | 表示 | |
| CIF形式データ |  emd_24620_validation.cif.gz emd_24620_validation.cif.gz | 12 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24620 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24620 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24620 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24620 | HTTPS FTP |
-関連構造データ
| 関連構造データ | C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24620.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24620.map.gz / 形式: CCP4 / 大きさ: 3.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Final sharpened map of negative stain electron microscopy single particle reconstruction of monoclonal antibody DH1025.1 Fab in complex with HIV-1 Env CH505 SOSIP trimer. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.02 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
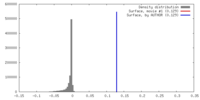
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: Raw, unsharpened map of negative stain electron microscopy...
| ファイル | emd_24620_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw, unsharpened map of negative stain electron microscopy single particle reconstruction of monoclonal antibody DH1025.1 Fab in complex with HIV-1 Env CH505 SOSIP trimer. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
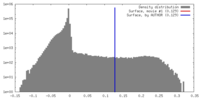
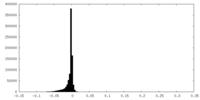
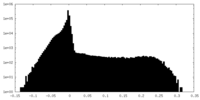
| 密度ヒストグラム |
-ハーフマップ: Half-map 1 of negative stain electron microscopy single...
| ファイル | emd_24620_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 of negative stain electron microscopy single particle reconstruction of monoclonal antibody DH1025.1 Fab in complex with HIV-1 Env CH505 SOSIP trimer. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
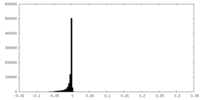
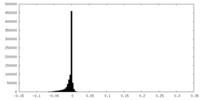
| 密度ヒストグラム |
-ハーフマップ: Half-map 2 of negative stain electron microscopy single...
| ファイル | emd_24620_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 of negative stain electron microscopy single particle reconstruction of monoclonal antibody DH1025.1 Fab in complex with HIV-1 Env CH505 SOSIP trimer. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
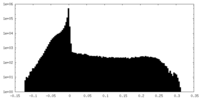
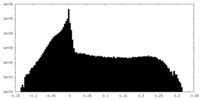
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of HIV Env CH505TFchim.6R.SOSIP.664.v4.1 with DH1025.1 an...
| 全体 | 名称: Complex of HIV Env CH505TFchim.6R.SOSIP.664.v4.1 with DH1025.1 antibody Fab |
|---|---|
| 要素 |
|
-超分子 #1: Complex of HIV Env CH505TFchim.6R.SOSIP.664.v4.1 with DH1025.1 an...
| 超分子 | 名称: Complex of HIV Env CH505TFchim.6R.SOSIP.664.v4.1 with DH1025.1 antibody Fab タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Human immunodeficiency virus 2 (ヒト免疫不全ウイルス) Human immunodeficiency virus 2 (ヒト免疫不全ウイルス) |
| 分子量 | 理論値: 23 KDa |
-超分子 #2: HIV Env CH505TFchim.6R.SOSIP.664.v4.1 trimer
| 超分子 | 名称: HIV Env CH505TFchim.6R.SOSIP.664.v4.1 trimer / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Human immunodeficiency virus 2 (ヒト免疫不全ウイルス) Human immunodeficiency virus 2 (ヒト免疫不全ウイルス) |
-超分子 #3: DH1025.1 Antibody Fab
| 超分子 | 名称: DH1025.1 Antibody Fab / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #4: DH1025.1 Antibody Fab Heavy Chain
| 超分子 | 名称: DH1025.1 Antibody Fab Heavy Chain / タイプ: complex / ID: 4 / 親要素: 3 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:  |
-超分子 #5: DH1025.1 Antibody Fab Light Chain
| 超分子 | 名称: DH1025.1 Antibody Fab Light Chain / タイプ: complex / ID: 5 / 親要素: 3 / 含まれる分子: #3 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: HIV Env CH505TFchim.6R.SOSIP.664.v4.1
| 分子 | 名称: HIV Env CH505TFchim.6R.SOSIP.664.v4.1 / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: AENLWVTVYY GVPVWKEAKT TLFCASDAKA YEKEVHNVWA THACVPTDPN PQEMVLKNVT ENFNMWKNDM VDQMHEDVIS LWDQSLKPCV KLTPLCVTLN CTNATASNSS IIEGMKNCSF NITTELRDKR EKKNALFYKL DIVQLDGNSS QYRLINCNTS VITQACPKVS ...文字列: AENLWVTVYY GVPVWKEAKT TLFCASDAKA YEKEVHNVWA THACVPTDPN PQEMVLKNVT ENFNMWKNDM VDQMHEDVIS LWDQSLKPCV KLTPLCVTLN CTNATASNSS IIEGMKNCSF NITTELRDKR EKKNALFYKL DIVQLDGNSS QYRLINCNTS VITQACPKVS FDPIPIHYCA PAGYAILKCN NKTFTGTGPC NNVSTVQCTH GIKPVVSTQL LLNGSLAEGE IIIRSENITN NVKTIIVHLN ESVKIECTRP NNKTRTSIRI GPGQAFYATG QVIGDIREAY CNINESKWNE TLQRVSKKLK EYFPHKNITF QPSSGGDLEI TTHSFNCGGE FFYCNTSSLF NRTYMANSTD MANSTETNST RTITIHCRIK QIINMWQEVG RAMYAPPIAG NITCISNITG LLLTRDGGKN NTETFRPGGG NMKDNWRSEL YKYKVVKIEP LGVAPTRCKR RVVGRRRRRR AVGIGAVFLG FLGAAGSTMG AASMTLTVQA RNLLSGIVQQ QSNLLRAPEA QQHLLKLTVW GIKQLQARVL AVERYLRDQQ LLGIWGCSGK LICCTNVPWN SSWSNRNLSE IWDNMTWLQW DKEISNYTQI IYGLLEESQN QQEKNEQDLL ALD |
-分子 #2: DH1025.1 Antibody Fab Heavy Chain
| 分子 | 名称: DH1025.1 Antibody Fab Heavy Chain / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: QVHLQESGPG LVKPSETLSL TCAVSGGSVS YYNWWSWIRQ PPGKGLEWIG YISGSSGNTY YSPSLKSRVT ISTDTSKNQF SLRLSSVTAA DTAVYYCASV LQYLDWVLPD RVGEVHWFDV WGPGVLVSVS S astkg ps vfplapss r stsestaal gclvkdyfpe ...文字列: QVHLQESGPG LVKPSETLSL TCAVSGGSVS YYNWWSWIRQ PPGKGLEWIG YISGSSGNTY YSPSLKSRVT ISTDTSKNQF SLRLSSVTAA DTAVYYCASV LQYLDWVLPD RVGEVHWFDV WGPGVLVSVS S astkg ps vfplapss r stsestaal gclvkdyfpe pvtvswnsg s ltsgvhtf pa vlqssgl ysl ssvvtv psss lgtqt yvcnv nhkp sntkvd krv eiktcgg |
-分子 #3: DH1025.1 Antibody Fab Light Chain
| 分子 | 名称: DH1025.1 Antibody Fab Light Chain / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: DIQMTQSPSS LSASVGDRVT ISCQASQGIS SRLAWYQQRP GKAPKLLIHT ASSLQSGVPS RFSGSGSGTE FTLTISSLQP EDFATYYCQQ HHSHPLTFGG GTKVEIK lf ppsseelq a nkatlvcli sdfypgvvkv awkadgsav n agvetttp sk qsnnkya ...文字列: DIQMTQSPSS LSASVGDRVT ISCQASQGIS SRLAWYQQRP GKAPKLLIHT ASSLQSGVPS RFSGSGSGTE FTLTISSLQP EDFATYYCQQ HHSHPLTFGG GTKVEIK lf ppsseelq a nkatlvcli sdfypgvvkv awkadgsav n agvetttp sk qsnnkya ass ylslts dqwk shksy scqvt hegs tvektv apa ecs |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
| ||||||||||||
| 染色 | タイプ: NEGATIVE / 材質: uranyl formate 詳細: For negative staining, 5 microliter of the diluted filter retentate was applied to a freshly glow-discharged, 300 mesh, carbon-coated EM grid and incubated 10-12 s, then rinsed with 70 ...詳細: For negative staining, 5 microliter of the diluted filter retentate was applied to a freshly glow-discharged, 300 mesh, carbon-coated EM grid and incubated 10-12 s, then rinsed with 70 microliter of 2% uranyl formate delivered in one smooth ejection from a pipet. The drop of uranyl formate remaining on the grid was allowed to incubate for 60 s, then the grid was blotted with filter paper and allowed to air dry. | ||||||||||||
| グリッド | モデル: Homemade / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 支持フィルム - Film thickness: 5.0 nm / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. | ||||||||||||
| 詳細 | Frozen aliquots of SOSIP and Fab were thawed in an Aluminum block at room temperature for 2-3 minutes, then 10 microgram of SOSIP were mixed with 36 microgram Fab (~1:15 molar ratio) and diluted to 100 microliter final volume with HEPES-buffered saline (HBS), containing 20 mM HEPES buffer and 150 mM NaCl, pH 7.4, augmented with 5% glycerol. Mixture was incubated at 22 degrees C overnight. The following day, a 0.08% glutaraldehyde fixative was prepared by diluting an 8% stock 1/100 with HBS. Then 400 microliter of fixative was added to the SOSIP-Fab mixture and incubated 5 min at room temperature to crosslink the complex, followed by addition of 40 microliter of a 1 M Tris solution, pH 7.4 to quench any unreacted glutaraldehyde. The reaction mixture was then transferred to a 0.5-ml Amicon Ultra centrifugal filter device with a 100-kDa cutoff filter (Millipore, #UFC510096) and spun 10 min at 14,000 g in a benchtop centrifuge held at 20 degrees C to remove excess Fab. The filter retentate, usually ~25 microliter, was collected and diluted 1:1 with HBS augmented with 5% glycerol. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI/PHILIPS EM420 |
|---|---|
| 撮影 | フィルム・検出器のモデル: OTHER / デジタル化 - サイズ - 横: 2048 pixel / デジタル化 - サイズ - 縦: 2048 pixel / 撮影したグリッド数: 1 / 実像数: 102 / 平均露光時間: 0.25 sec. / 平均電子線量: 32.0 e/Å2 詳細: Images were collected with a minimal dose approach using a manually controlled low dose unit to keep the beam blanked except during initial grid orientation and image collection. To create an ...詳細: Images were collected with a minimal dose approach using a manually controlled low dose unit to keep the beam blanked except during initial grid orientation and image collection. To create an initial atlas, the entire grid was imaged once at 49x magnification at very low beam intensity by decreasing the emission current, strongly focusing C1 (Spot Size 6) and defocusing C2 (Intensity knob fully clockwise). The stage was then quickly translated to center the desired gridsquare, with an estimated exposure of < 2 s at this very low illumination level, at which point the beam was blanked. A 3,300x magnification was then selected so that a single gridsquare approximately fills the scintillator screen. The beam was then unblanked and the stage manually tilted and the eucentric height quickly adjusted by noting the movement of the gridbars, and then the stage was translated to center on the lower-left corner of the gridsquare, with an estimated exposure of < 2 s at this very low illumination level, at which point the beam was blanked. A magnfication of 82,000x was then selected and C2 focused by a known number of clicks of the Intensity knob so that the beam would approximately fill the scintillator screen. The beam was then unblanked, the objective lens focused by eye and then adjusted to approximately 0.5 micron underfocused, and C2 adjusted so the beam just filled the scintillator screen. Because this focusing step involved a long exposure to the focused beam, no data was collected in this area. For data collection, the stage was translated blindly, with the beam blanked, by noting the micrometer markings on the left-hand stage control, to move to an un-irradiated area that was approximately two screen-widths vertically away from the initial position. The beam was then unblanked, an image captured with 0.25 s exposure to the focused beam without any adjustment of the objective lens focus, and the beam then re-blanked. The FFT of the collected image was examined and the position of the first Thon ring noted and the objective lens focus adjusted if needed for subsequent images. The stage was then moved blindly in the same direction to an un-irradiated area and a new image collected. In this way images were collected until the gridbar was encountered, at which point the beam was unblanked and the stage moved so that the gridbar occluded all of the beam except a small sliver of the gridsquare at the bottom or top of the screen, and the right hand stage control was used to translate the specimen to a new location at least two screen widths away horizontally. In this way the entire gridsquare could be imaged in a rastered fashion with each area only irradiated with the full strength beam during the 0.25 s image capture. |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.0 mm / 最大 デフォーカス(公称値): 1.5 µm / 最小 デフォーカス(公称値): 0.2 µm / 倍率(公称値): 82000 |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC / ホルダー冷却材: NITROGEN |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera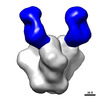















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)