登録情報 データベース : EMDB / ID : EMD-24482タイトル Complex III2 from Candida albicans, inhibitor free Candida albicans CIII2, inhibitor free 複合体 : Respiratory complex III2 from Candida albicansリガンド : x 4種 / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
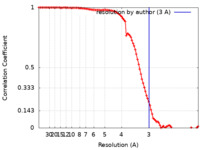
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Candida albicans SC5314 (酵母) / Candida albicans (strain SC5314 / ATCC MYA-2876) (酵母)手法 / / 解像度 : 3.0 Å Di Trani JM / Rubinstein JL 資金援助 Organization Grant number 国 Canadian Institutes of Health Research (CIHR) PJT162186 Canadian Institutes of Health Research (CIHR) FDN154288 Knut and Alice Wallenberg Foundation 2019.0043 Swedish Research Council 2018-04619 Canada Research Chairs
ジャーナル : Structure / 年 : 2022タイトル : Rieske head domain dynamics and indazole-derivative inhibition of Candida albicans complex III.著者 : Justin M Di Trani / Zhongle Liu / Luke Whitesell / Peter Brzezinski / Leah E Cowen / John L Rubinstein / 要旨 : Electron transfer between respiratory complexes drives transmembrane proton translocation, which powers ATP synthesis and membrane transport. The homodimeric respiratory complex III (CIII) oxidizes ... Electron transfer between respiratory complexes drives transmembrane proton translocation, which powers ATP synthesis and membrane transport. The homodimeric respiratory complex III (CIII) oxidizes ubiquinol to ubiquinone, transferring electrons to cytochrome c and translocating protons through a mechanism known as the Q cycle. The Q cycle involves ubiquinol oxidation and ubiquinone reduction at two different sites within each CIII monomer, as well as movement of the head domain of the Rieske subunit. We determined structures of Candida albicans CIII by cryoelectron microscopy (cryo-EM), revealing endogenous ubiquinone and visualizing the continuum of Rieske head domain conformations. Analysis of these conformations does not indicate cooperativity in the Rieske head domain position or ligand binding in the two CIIIs of the CIII dimer. Cryo-EM with the indazole derivative Inz-5, which inhibits fungal CIII and is fungicidal when administered with fungistatic azole drugs, showed that Inz-5 inhibition alters the equilibrium of Rieske head domain positions. 履歴 登録 2021年7月20日 - ヘッダ(付随情報) 公開 2021年9月15日 - マップ公開 2021年9月15日 - 更新 2025年5月28日 - 現状 2025年5月28日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Candida albicans SC5314 (酵母) /
Candida albicans SC5314 (酵母) /  Candida albicans (strain SC5314 / ATCC MYA-2876) (酵母)
Candida albicans (strain SC5314 / ATCC MYA-2876) (酵母) データ登録者
データ登録者 カナダ,
カナダ,  スウェーデン, 5件
スウェーデン, 5件  引用
引用 ジャーナル: Structure / 年: 2022
ジャーナル: Structure / 年: 2022

 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_24482.map.gz
emd_24482.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-24482-v30.xml
emd-24482-v30.xml emd-24482.xml
emd-24482.xml EMDBヘッダ
EMDBヘッダ emd_24482_fsc.xml
emd_24482_fsc.xml FSCデータファイル
FSCデータファイル emd_24482.png
emd_24482.png emd-24482.cif.gz
emd-24482.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-24482
http://ftp.pdbj.org/pub/emdb/structures/EMD-24482 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24482
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24482 emd_24482_validation.pdf.gz
emd_24482_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_24482_full_validation.pdf.gz
emd_24482_full_validation.pdf.gz emd_24482_validation.xml.gz
emd_24482_validation.xml.gz emd_24482_validation.cif.gz
emd_24482_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24482
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24482 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24482
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24482 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_24482.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_24482.map.gz / 形式: CCP4 / 大きさ: 91.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー


























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)