+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24450 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HUMAN RETINAL VARIANT IMPDH1(546) TREATED WITH GTP, ATP, IMP, NAD+; INTERFACE-CENTERED | ||||||||||||
 マップデータ マップデータ | HUMAN RETINAL VARIANT IMPDH1(546) TREATED WITH GTP, ATP, IMP, NAD+, INTERFACE-CENTERED | ||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード | METABOLISM / FILAMENT / ALLOSTERY / ADENINE / OXIDOREDUCTASE | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Purine ribonucleoside monophosphate biosynthesis / IMP dehydrogenase activity / IMP dehydrogenase / GMP biosynthetic process / Azathioprine ADME / GTP biosynthetic process / azurophil granule lumen / secretory granule lumen / Potential therapeutics for SARS / ficolin-1-rich granule lumen ...Purine ribonucleoside monophosphate biosynthesis / IMP dehydrogenase activity / IMP dehydrogenase / GMP biosynthetic process / Azathioprine ADME / GTP biosynthetic process / azurophil granule lumen / secretory granule lumen / Potential therapeutics for SARS / ficolin-1-rich granule lumen / nucleic acid binding / nucleotide binding / Neutrophil degranulation / DNA binding / RNA binding / extracellular region / metal ion binding / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | ||||||||||||
 データ登録者 データ登録者 | Burrell AL / Kollman JM | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2022 ジャーナル: Nat Struct Mol Biol / 年: 2022タイトル: IMPDH1 retinal variants control filament architecture to tune allosteric regulation. 著者: Anika L Burrell / Chuankai Nie / Meerit Said / Jacqueline C Simonet / David Fernández-Justel / Matthew C Johnson / Joel Quispe / Rubén M Buey / Jeffrey R Peterson / Justin M Kollman /   要旨: Inosine-5'-monophosphate dehydrogenase (IMPDH), a key regulatory enzyme in purine nucleotide biosynthesis, dynamically assembles filaments in response to changes in metabolic demand. Humans have two ...Inosine-5'-monophosphate dehydrogenase (IMPDH), a key regulatory enzyme in purine nucleotide biosynthesis, dynamically assembles filaments in response to changes in metabolic demand. Humans have two isoforms: IMPDH2 filaments reduce sensitivity to feedback inhibition, while IMPDH1 assembly remains uncharacterized. IMPDH1 plays a unique role in retinal metabolism, and point mutants cause blindness. Here, in a series of cryogenic-electron microscopy structures we show that human IMPDH1 assembles polymorphic filaments with different assembly interfaces in extended and compressed states. Retina-specific splice variants introduce structural elements that reduce sensitivity to GTP inhibition, including stabilization of the extended filament form. Finally, we show that IMPDH1 disease mutations fall into two classes: one disrupts GTP regulation and the other has no effect on GTP regulation or filament assembly. These findings provide a foundation for understanding the role of IMPDH1 in retinal function and disease and demonstrate the diverse mechanisms by which metabolic enzyme filaments are allosterically regulated. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24450.map.gz emd_24450.map.gz | 4.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24450-v30.xml emd-24450-v30.xml emd-24450.xml emd-24450.xml | 10.9 KB 10.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_24450.png emd_24450.png | 127.7 KB | ||
| Filedesc metadata |  emd-24450.cif.gz emd-24450.cif.gz | 5.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24450 http://ftp.pdbj.org/pub/emdb/structures/EMD-24450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24450 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24450 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7rgiMC  7rerC  7resC  7rfeC  7rffC  7rfgC  7rfhC  7rfiC  7rgdC  7rglC  7rgmC  7rgqC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24450.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24450.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HUMAN RETINAL VARIANT IMPDH1(546) TREATED WITH GTP, ATP, IMP, NAD+, INTERFACE-CENTERED | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.16 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Assembly interface of IMPDH1 filament bound to ATP, IMP, NAD+
| 全体 | 名称: Assembly interface of IMPDH1 filament bound to ATP, IMP, NAD+ |
|---|---|
| 要素 |
|
-超分子 #1: Assembly interface of IMPDH1 filament bound to ATP, IMP, NAD+
| 超分子 | 名称: Assembly interface of IMPDH1 filament bound to ATP, IMP, NAD+ タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 55405 MDa |
-分子 #1: Inosine-5'-monophosphate dehydrogenase 1
| 分子 | 名称: Inosine-5'-monophosphate dehydrogenase 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO / EC番号: IMP dehydrogenase |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 58.37077 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MADYLISGGT GYVPEDGLTA QQLFASADGL TYNDFLILPG FIDFIADEVD LTSALTRKIT LKTPLISSPM DTVTEADMAI AMALMGGIG FIHHNCTPEF QANEVRKVKK FEQGFITDPV VLSPSHTVGD VLEAKMRHGF SGIPITETGT MGSKLVGIVT S RDIDFLAE ...文字列: MADYLISGGT GYVPEDGLTA QQLFASADGL TYNDFLILPG FIDFIADEVD LTSALTRKIT LKTPLISSPM DTVTEADMAI AMALMGGIG FIHHNCTPEF QANEVRKVKK FEQGFITDPV VLSPSHTVGD VLEAKMRHGF SGIPITETGT MGSKLVGIVT S RDIDFLAE KDHTTLLSEV MTPRIELVVA PAGVTLKEAN EILQRSKKGK LPIVNDCDEL VAIIARTDLK KNRDYPLASK DS QKQLLCG AAVGTREDDK YRLDLLTQAG VDVIVLDSSQ GNSVYQIAMV HYIKQKYPHL QVIGGNVVTA AQAKNLIDAG VDG LRVGMG CGSICITQEV MACGRPQGTA VYKVAEYARR FGVPIIADGG IQTVGHVVKA LALGASTVMM GSLLAATTEA PGEY FFSDG VRLKKYRGMG SLDAMEKSSS SQKRYFSEGD KVKIAQGVSG SIQDKGSIQK FVPYLIAGIQ HGCQDIGARS LSVLR SMMY SGELKFEKRT MSAQIEGGVH GLHSYTFLPF TKSGCTEDSG GGRGGGGDAP QCPLLGTASL HN UniProtKB: Inosine-5'-monophosphate dehydrogenase 1 |
-分子 #2: NICOTINAMIDE-ADENINE-DINUCLEOTIDE
| 分子 | 名称: NICOTINAMIDE-ADENINE-DINUCLEOTIDE / タイプ: ligand / ID: 2 / コピー数: 8 / 式: NAD |
|---|---|
| 分子量 | 理論値: 663.425 Da |
| Chemical component information |  ChemComp-NAD: |
-分子 #3: INOSINIC ACID
| 分子 | 名称: INOSINIC ACID / タイプ: ligand / ID: 3 / コピー数: 8 / 式: IMP |
|---|---|
| 分子量 | 理論値: 348.206 Da |
| Chemical component information | 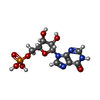 ChemComp-I: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: EMDB MAP |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.6 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 66449 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー






















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)





















