+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24444 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Mouse GITR (mGITR) with DTA-1 Fab fragment | |||||||||
 マップデータ マップデータ | mGITR with DTA-1 Fab | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Mouse glucocorticoid-induced tumor necrosis factor receptor / mGITR / TNF receptor / DTA-1 / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報TNFs bind their physiological receptors / tumor necrosis factor receptor activity / positive regulation of cell adhesion / bioluminescence / generation of precursor metabolites and energy / external side of plasma membrane / apoptotic process / negative regulation of apoptotic process / extracellular region 類似検索 - 分子機能 | |||||||||
| 生物種 |   Muromegalovirus G4 (ウイルス) Muromegalovirus G4 (ウイルス) | |||||||||
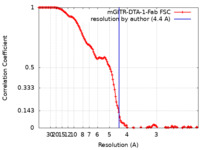
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 4.4 Å | |||||||||
 データ登録者 データ登録者 | Meyerson JR / He C | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Therapeutic antibody activation of the glucocorticoid-induced TNF receptor by a clustering mechanism. 著者: Changhao He / Rachana R Maniyar / Yahel Avraham / Roberta Zappasodi / Radda Rusinova / Walter Newman / Heidi Heath / Jedd D Wolchok / Rony Dahan / Taha Merghoub / Joel R Meyerson /   要旨: GITR is a TNF receptor, and its activation promotes immune responses and drives antitumor activity. The receptor is activated by the GITR ligand (GITRL), which is believed to cluster receptors into a ...GITR is a TNF receptor, and its activation promotes immune responses and drives antitumor activity. The receptor is activated by the GITR ligand (GITRL), which is believed to cluster receptors into a high-order array. Immunotherapeutic agonist antibodies also activate the receptor, but their mechanisms are not well characterized. We solved the structure of full-length mouse GITR bound to Fabs from the antibody DTA-1. The receptor is a dimer, and each subunit binds one Fab in an orientation suggesting that the antibody clusters receptors. Binding experiments with purified proteins show that DTA-1 IgG and GITRL both drive extensive clustering of GITR. Functional data reveal that DTA-1 and the anti-human GITR antibody TRX518 activate GITR in their IgG forms but not as Fabs. Thus, the divalent character of the IgG agonists confers an ability to mimic GITRL and cluster and activate GITR. These findings will inform the clinical development of this class of antibodies for immuno-oncology. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24444.map.gz emd_24444.map.gz | 203.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24444-v30.xml emd-24444-v30.xml emd-24444.xml emd-24444.xml | 12.1 KB 12.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24444_fsc.xml emd_24444_fsc.xml | 13.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24444.png emd_24444.png | 62.2 KB | ||
| Filedesc metadata |  emd-24444.cif.gz emd-24444.cif.gz | 5.9 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24444 http://ftp.pdbj.org/pub/emdb/structures/EMD-24444 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24444 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24444 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24444_validation.pdf.gz emd_24444_validation.pdf.gz | 486.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24444_full_validation.pdf.gz emd_24444_full_validation.pdf.gz | 486.4 KB | 表示 | |
| XML形式データ |  emd_24444_validation.xml.gz emd_24444_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  emd_24444_validation.cif.gz emd_24444_validation.cif.gz | 18.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24444 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24444 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24444 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24444 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24444.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24444.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | mGITR with DTA-1 Fab | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.096 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : mGITR with DTA-1 Fab
| 全体 | 名称: mGITR with DTA-1 Fab |
|---|---|
| 要素 |
|
-超分子 #1: mGITR with DTA-1 Fab
| 超分子 | 名称: mGITR with DTA-1 Fab / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Tumor necrosis factor receptor superfamily member 18,Enhanced gre...
| 分子 | 名称: Tumor necrosis factor receptor superfamily member 18,Enhanced green fluorescent protein タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Muromegalovirus G4 (ウイルス) Muromegalovirus G4 (ウイルス) |
| 分子量 | 理論値: 54.848238 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGAWAMLYGV SMLCVLDLGQ PSVVEEPGCG PGKVQNGSGN NTRCCSLYAP GKEDCPKERC ICVTPEYHCG DPQCKICKHY PCQPGQRVE SQGDIVFGFR CVACAMGTFS AGRDGHCRLW TNCSQFGFLT MFPGNKTHNA VCIPEPLPTE QYGHLTVIFL V MAACIFFL ...文字列: MGAWAMLYGV SMLCVLDLGQ PSVVEEPGCG PGKVQNGSGN NTRCCSLYAP GKEDCPKERC ICVTPEYHCG DPQCKICKHY PCQPGQRVE SQGDIVFGFR CVACAMGTFS AGRDGHCRLW TNCSQFGFLT MFPGNKTHNA VCIPEPLPTE QYGHLTVIFL V MAACIFFL TTVQLGLHIW QLRRQHMCPR ETQPFAEVQL SAEDACSFQF PEEERGEQTE EKCHLGGRWP AGLVPRGSAA AM VSKGEEL FTGVVPILVE LDGDVNGHKF SVSGEGEGDA TYGKLTLKFI CTTGKLPVPW PTLVTTLTYG VQCFSRYPDH MKQ HDFFKS AMPEGYVQER TIFFKDDGNY KTRAEVKFEG DTLVNRIELK GIDFKEDGNI LGHKLEYNYN SHNVYIMADK QKNG IKVNF KIRHNIEDGS VQLADHYQQN TPIGDGPVLL PDNHYLSTQS KLSKDPNEKR DHMVLLEFVT AAGITLGMDE LYKSG LRHH HHHHHH UniProtKB: Tumor necrosis factor receptor superfamily member 18, Enhanced green fluorescent protein |
-分子 #2: DTA-1 (heavy chain)
| 分子 | 名称: DTA-1 (heavy chain) / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 51.014621 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGWSCIILFL VATATGVHSE LQLVESGGGL VQPGRSLKLS CSASGFIFSN SYMAWVRQAP KKGLEWVATI NPSGSRTYYP DSVKGRFTI SRDTAKSSLY LQMNSLKSED TATYYCARHD LSFDYWGQGV MVTVSSASTK GPSVFPLAPS SKSTSGGTAA L GCLVKDYF ...文字列: MGWSCIILFL VATATGVHSE LQLVESGGGL VQPGRSLKLS CSASGFIFSN SYMAWVRQAP KKGLEWVATI NPSGSRTYYP DSVKGRFTI SRDTAKSSLY LQMNSLKSED TATYYCARHD LSFDYWGQGV MVTVSSASTK GPSVFPLAPS SKSTSGGTAA L GCLVKDYF PEPVTVSWNS GALTSGVHTF PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKRVEPKSC DK THTCPPC PAPELLGGPS VFLFPPKPKD TLMISRTPEV TCVVVDVSHE DPEVKFNWYV DGVEVHNAKT KPREEQYNST YRV VSVLTV LHQDWLNGKE YKCKVSNKAL PAPIEKTISK AKGQPREPQV YTLPPSREEM TKNQVSLTCL VKGFYPSDIA VEWE SNGQP ENNYKTTPPV LDSDGSFFLY SKLTVDKSRW QQGNVFSCSV MHEALHNHYT QKSLSLSPGK |
-分子 #3: DTA-1 (light chain)
| 分子 | 名称: DTA-1 (light chain) / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 26.320057 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MGWSCIILFL VATATGVHSQ FTLTQPKSVS GSLRSTITIP CDRSSGGIRD SYVSWYQQHL GRPPLNVIYA DDQRPSEVSD RFSGSIDSS SNSASLTITN LQMDDEADYF CQSYDSDFDV YIFGGGTKLT VLGQRTVAAP SVFIFPPSDE QLKSGTASVV C LLNNFYPR ...文字列: MGWSCIILFL VATATGVHSQ FTLTQPKSVS GSLRSTITIP CDRSSGGIRD SYVSWYQQHL GRPPLNVIYA DDQRPSEVSD RFSGSIDSS SNSASLTITN LQMDDEADYF CQSYDSDFDV YIFGGGTKLT VLGQRTVAAP SVFIFPPSDE QLKSGTASVV C LLNNFYPR EAKVQWKVDN ALQSGNSQES VTEQDSKDST YSLSSTLTLS KADYEKHKVY ACEVTHQGLS SPVTKSFNRG EC |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 55.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー














 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)