+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24252 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of MFSD2A | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | membrane protein / TRANSPORT PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報lysophosphatidylcholine flippase activity / regulation of phosphatidylethanolamine metabolic process / : / oleate transmembrane transporter activity / Synthesis of PC / regulation of phosphatidylcholine metabolic process / regulation of neuron projection arborization / retina morphogenesis in camera-type eye / photoreceptor cell morphogenesis / lysophospholipid translocation ...lysophosphatidylcholine flippase activity / regulation of phosphatidylethanolamine metabolic process / : / oleate transmembrane transporter activity / Synthesis of PC / regulation of phosphatidylcholine metabolic process / regulation of neuron projection arborization / retina morphogenesis in camera-type eye / photoreceptor cell morphogenesis / lysophospholipid translocation / fatty acid transmembrane transporter activity / lysophospholipid:sodium symporter activity / lysophospholipid transport / photoreceptor cell outer segment organization / lipid transport across blood-brain barrier / negative regulation of fatty acid beta-oxidation / very-low-density lipoprotein particle assembly / positive regulation of triglyceride biosynthetic process / regulation of dendrite development / retinal pigment epithelium development / transcytosis / establishment of blood-brain barrier / symporter activity / long-chain fatty acid transmembrane transporter activity / maintenance of blood-brain barrier / motor behavior / carbohydrate transport / regulation of multicellular organism growth / long-chain fatty acid transport / fatty acid transport / energy homeostasis / cellular response to starvation / hippocampus development / brain development / cognition / cytoplasmic ribonucleoprotein granule / positive regulation of cell growth / endoplasmic reticulum membrane / plasma membrane / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
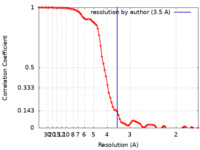
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Zhang J / Feng L | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2021 ジャーナル: Nature / 年: 2021タイトル: Structure and mechanism of blood-brain-barrier lipid transporter MFSD2A. 著者: Chase A P Wood / Jinru Zhang / Deniz Aydin / Yan Xu / Benjamin J Andreone / Urs H Langen / Ron O Dror / Chenghua Gu / Liang Feng /  要旨: MFSD2A is a sodium-dependent lysophosphatidylcholine symporter that is responsible for the uptake of docosahexaenoic acid into the brain, which is crucial for the development and performance of the ...MFSD2A is a sodium-dependent lysophosphatidylcholine symporter that is responsible for the uptake of docosahexaenoic acid into the brain, which is crucial for the development and performance of the brain. Mutations that affect MFSD2A cause microcephaly syndromes. The ability of MFSD2A to transport lipid is also a key mechanism that underlies its function as an inhibitor of transcytosis to regulate the blood-brain barrier. Thus, MFSD2A represents an attractive target for modulating the permeability of the blood-brain barrier for drug delivery. Here we report the cryo-electron microscopy structure of mouse MFSD2A. Our structure defines the architecture of this important transporter, reveals its unique extracellular domain and uncovers its substrate-binding cavity. The structure-together with our functional studies and molecular dynamics simulations-identifies a conserved sodium-binding site, reveals a potential lipid entry pathway and helps to rationalize MFSD2A mutations that underlie microcephaly syndromes. These results shed light on the critical lipid transport function of MFSD2A and provide a framework to aid in the design of specific modulators for therapeutic purposes. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24252.map.gz emd_24252.map.gz | 43.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24252-v30.xml emd-24252-v30.xml emd-24252.xml emd-24252.xml | 13.9 KB 13.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24252_fsc.xml emd_24252_fsc.xml | 10.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24252.png emd_24252.png | 106.5 KB | ||
| Filedesc metadata |  emd-24252.cif.gz emd-24252.cif.gz | 5.3 KB | ||
| その他 |  emd_24252_half_map_1.map.gz emd_24252_half_map_1.map.gz emd_24252_half_map_2.map.gz emd_24252_half_map_2.map.gz | 36.8 MB 36.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24252 http://ftp.pdbj.org/pub/emdb/structures/EMD-24252 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24252 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24252 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24252_validation.pdf.gz emd_24252_validation.pdf.gz | 848.9 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24252_full_validation.pdf.gz emd_24252_full_validation.pdf.gz | 848.4 KB | 表示 | |
| XML形式データ |  emd_24252_validation.xml.gz emd_24252_validation.xml.gz | 14.1 KB | 表示 | |
| CIF形式データ |  emd_24252_validation.cif.gz emd_24252_validation.cif.gz | 18.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24252 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24252 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24252 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24252 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24252.map.gz / 形式: CCP4 / 大きさ: 47.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24252.map.gz / 形式: CCP4 / 大きさ: 47.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: #2
| ファイル | emd_24252_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_24252_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : membrane protein complex
| 全体 | 名称: membrane protein complex |
|---|---|
| 要素 |
|
-超分子 #1: membrane protein complex
| 超分子 | 名称: membrane protein complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Sodium-dependent lysophosphatidylcholine symporter 1
| 分子 | 名称: Sodium-dependent lysophosphatidylcholine symporter 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 59.038602 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: MAKGEGAESG SAAGLLPTSI LQASERPVQV KKEPKKKQQL SICNKLCYAV GGAPYQLTGC ALGFFLHIYL LDVAKVEPLP ASIILFVGR AWDAFTDPLV GFCISKSSWT RLGRLMPWII FSTPLAIIAY FLIWFVPDFP SGTESSHGFL WYLLFYCLFE T LVTCFHVP ...文字列: MAKGEGAESG SAAGLLPTSI LQASERPVQV KKEPKKKQQL SICNKLCYAV GGAPYQLTGC ALGFFLHIYL LDVAKVEPLP ASIILFVGR AWDAFTDPLV GFCISKSSWT RLGRLMPWII FSTPLAIIAY FLIWFVPDFP SGTESSHGFL WYLLFYCLFE T LVTCFHVP YSALTMFIST EQSERDSATA YRMTVEVLGT VIGTAIQGQI VGQAKAPCLQ DQNGSVVVSE VANRTQSTAS LK DTQNAYL LAAGIIASIY VLCAFILILG VREQRELYES QQAESMPFFQ GLRLVMGHGP YVKLIAGFLF TSLAFMLVEG NFA LFCTYT LDFRNEFQNL LLAIMLSATF TIPIWQWFLT RFGKKTAVYI GISSAVPFLI LVALMERNLI VTYVVAVAAG VSVA AAFLL PWSMLPDVID DFHLKHPHSP GTEPIFFSFY VFFTKFASGV SLGVSTLSLD FANYQRQGCS QPEQVKFTLK MLVTM APII LILLGLLLFK LYPIDEEKRR QNKKALQALR EEASSSGCSD TDSTELASIL UniProtKB: Sodium-dependent lysophosphatidylcholine symporter 1 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R2/1 / 材質: GOLD / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 96 % / チャンバー内温度: 293 K / 装置: LEICA EM GP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 X (Sec.)
X (Sec.) Y (Row.)
Y (Row.) Z (Col.)
Z (Col.)