+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-2410 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Hsc70-induced Changes in Clathrin-Auxilin Cage Structure Suggest a Role for Clathrin Light Chains in Cage Disassembly | |||||||||
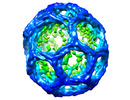
 マップデータ マップデータ | Clathrin-auxilin-hsc70 complex | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Endocytosis / coated vesicles / clathrin / hsc70 / auxilin | |||||||||
| 生物種 |   | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 34.0 Å | |||||||||
 データ登録者 データ登録者 | Young A / Stoilova-McPhie S / Rothnie A / Vallis Y / Harvey-Smith P / Ranson N / Kent H / Brodsky FM / Pearse BM / Roseman A / Smith CJ | |||||||||
 引用 引用 |  ジャーナル: Traffic / 年: 2013 ジャーナル: Traffic / 年: 2013タイトル: Hsc70-induced changes in clathrin-auxilin cage structure suggest a role for clathrin light chains in cage disassembly. 著者: Anna Young / Svetla Stoilova-McPhie / Alice Rothnie / Yvonne Vallis / Phillip Harvey-Smith / Neil Ranson / Helen Kent / Frances M Brodsky / Barbara M F Pearse / Alan Roseman / Corinne J Smith /  要旨: The molecular chaperone, Hsc70, together with its co-factor, auxilin, facilitates the ATP-dependent removal of clathrin during clathrin-mediated endocytosis in cells. We have used cryo-electron ...The molecular chaperone, Hsc70, together with its co-factor, auxilin, facilitates the ATP-dependent removal of clathrin during clathrin-mediated endocytosis in cells. We have used cryo-electron microscopy to determine the 3D structure of a complex of clathrin, auxilin(401-910) and Hsc70 at pH 6 in the presence of ATP, frozen within 20 seconds of adding Hsc70 in order to visualize events that follow the binding of Hsc70 to clathrin and auxilin before clathrin disassembly. In this map, we observe density beneath the vertex of the cage that we attribute to bound Hsc70. This density emerges asymmetrically from the clathrin vertex, suggesting preferential binding by Hsc70 for one of the three possible sites at the vertex. Statistical comparison with a map of whole auxilin and clathrin previously published by us reveals the location of statistically significant differences which implicate involvement of clathrin light chains in structural rearrangements which occur after Hsc70 is recruited. Clathrin disassembly assays using light scattering suggest that loss of clathrin light chains reduces the efficiency with which auxilin facilitates this reaction. These data support a regulatory role for clathrin light chains in clathrin disassembly in addition to their established role in regulating clathrin assembly. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_2410.map.gz emd_2410.map.gz | 58.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-2410-v30.xml emd-2410-v30.xml emd-2410.xml emd-2410.xml | 9 KB 9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_2410.png emd_2410.png | 470.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-2410 http://ftp.pdbj.org/pub/emdb/structures/EMD-2410 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2410 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-2410 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_2410_validation.pdf.gz emd_2410_validation.pdf.gz | 250.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_2410_full_validation.pdf.gz emd_2410_full_validation.pdf.gz | 249.3 KB | 表示 | |
| XML形式データ |  emd_2410_validation.xml.gz emd_2410_validation.xml.gz | 6.9 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2410 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2410 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2410 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-2410 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_2410.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_2410.map.gz / 形式: CCP4 / 大きさ: 62.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Clathrin-auxilin-hsc70 complex | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Clathrin, auxilin (residues 401-910) and Hsc70
| 全体 | 名称: Clathrin, auxilin (residues 401-910) and Hsc70 |
|---|---|
| 要素 |
|
-超分子 #1000: Clathrin, auxilin (residues 401-910) and Hsc70
| 超分子 | 名称: Clathrin, auxilin (residues 401-910) and Hsc70 / タイプ: sample / ID: 1000 / 集合状態: Not experimentally determined / Number unique components: 3 |
|---|
-分子 #1: Hsc70
| 分子 | 名称: Hsc70 / タイプ: protein_or_peptide / ID: 1 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #2: auxilin401-910
| 分子 | 名称: auxilin401-910 / タイプ: protein_or_peptide / ID: 2 / 組換発現: Yes |
|---|---|
| 由来(天然) | 生物種:  |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6 詳細: 20mM MES, 2mM magnesium acetate, 25mM KCl, 10mM (NH4)2SO4, 1.8mM Hepes, 18mM NaCl, 1mM DTT |
|---|---|
| グリッド | 詳細: Quantifoil holey carbon grids, glow discharged in amylamine atmosphere |
| 凍結 | 凍結剤: ETHANE / 装置: HOMEMADE PLUNGER Timed resolved state: Vitrified rapidly following addition of Hsc70-ATP |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | JEOL 2011 |
|---|---|
| 日付 | 2004年1月1日 |
| 撮影 | カテゴリ: FILM / フィルム・検出器のモデル: KODAK SO-163 FILM / デジタル化 - スキャナー: NIKON COOLSCAN |
| 電子線 | 加速電圧: 200 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 4.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 40000 |
| 試料ステージ | 試料ホルダーモデル: GATAN LIQUID NITROGEN |
- 画像解析
画像解析
| CTF補正 | 詳細: Whole micrographs |
|---|---|
| 最終 再構成 | 想定した対称性 - 点群: D6 (2回x6回 2面回転対称) アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 34.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: MRC, SPIDER, FREALIGN 詳細: The EM map has been sharpened to match a Fourier amplitude profile derived from a model of the hexagonal barrel (the biological unit) created from the crystal structure atomic model pdb ...詳細: The EM map has been sharpened to match a Fourier amplitude profile derived from a model of the hexagonal barrel (the biological unit) created from the crystal structure atomic model pdb id1XI5. Fourier filtered to 30 Angstroms. 使用した粒子像数: 1051 |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera