+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-24072 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Ab1245 Fab in complex with BG505 SOSIP.664 and 8ANC195 Fab | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | VIRAL PROTEIN-IMMUNE SYSTEM complex / non-neutralizing / ANTIVIRAL PROTEIN / ANTIVIRAL PROTEIN-VIRAL PROTEIN complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / host cell plasma membrane / virion membrane / structural molecule activity / identical protein binding / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /    Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) | |||||||||
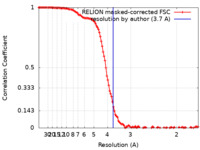
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Abernathy ME / Bjorkman PJ | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: NPJ Vaccines / 年: 2021 ジャーナル: NPJ Vaccines / 年: 2021タイトル: Antibody elicited by HIV-1 immunogen vaccination in macaques displaces Env fusion peptide and destroys a neutralizing epitope. 著者: Morgan E Abernathy / Harry B Gristick / Jost Vielmetter / Jennifer R Keeffe / Priyanthi N P Gnanapragasam / Yu E Lee / Amelia Escolano / Rajeev Gautam / Michael S Seaman / Malcolm A Martin / ...著者: Morgan E Abernathy / Harry B Gristick / Jost Vielmetter / Jennifer R Keeffe / Priyanthi N P Gnanapragasam / Yu E Lee / Amelia Escolano / Rajeev Gautam / Michael S Seaman / Malcolm A Martin / Michel C Nussenzweig / Pamela J Bjorkman /  要旨: HIV-1 vaccine design aims to develop an immunogen that elicits broadly neutralizing antibodies against a desired epitope, while eliminating responses to off-target regions of HIV-1 Env. We report ...HIV-1 vaccine design aims to develop an immunogen that elicits broadly neutralizing antibodies against a desired epitope, while eliminating responses to off-target regions of HIV-1 Env. We report characterization of Ab1245, an off-target antibody against the Env gp120-gp41 interface, from V3-glycan patch immunogen-primed and boosted macaques. A 3.7 Å cryo-EM structure of an Ab1245-Env complex reveals one Ab1245 Fab binding asymmetrically to Env trimer at the gp120-gp41 interface using its long CDRH3 to mimic regions of gp41. The mimicry includes positioning of a CDRH3 methionine into the gp41 tryptophan clasp, resulting in displacement of the fusion peptide and fusion peptide-proximal region. Despite fusion peptide displacement, Ab1245 is non-neutralizing even at high concentrations, raising the possibility that only two fusion peptides per trimer are required for viral-host membrane fusion. These structural analyses facilitate immunogen design to prevent elicitation of Ab1245-like antibodies that block neutralizing antibodies against the fusion peptide. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_24072.map.gz emd_24072.map.gz | 135.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-24072-v30.xml emd-24072-v30.xml emd-24072.xml emd-24072.xml | 29.1 KB 29.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_24072_fsc.xml emd_24072_fsc.xml | 12 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_24072.png emd_24072.png | 138.3 KB | ||
| マスクデータ |  emd_24072_msk_1.map emd_24072_msk_1.map | 144.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-24072.cif.gz emd-24072.cif.gz | 7.9 KB | ||
| その他 |  emd_24072_additional_1.map.gz emd_24072_additional_1.map.gz emd_24072_half_map_1.map.gz emd_24072_half_map_1.map.gz emd_24072_half_map_2.map.gz emd_24072_half_map_2.map.gz | 132 MB 35.9 MB 35.9 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-24072 http://ftp.pdbj.org/pub/emdb/structures/EMD-24072 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24072 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-24072 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_24072_validation.pdf.gz emd_24072_validation.pdf.gz | 828.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_24072_full_validation.pdf.gz emd_24072_full_validation.pdf.gz | 827.8 KB | 表示 | |
| XML形式データ |  emd_24072_validation.xml.gz emd_24072_validation.xml.gz | 19.4 KB | 表示 | |
| CIF形式データ |  emd_24072_validation.cif.gz emd_24072_validation.cif.gz | 25.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24072 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24072 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24072 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-24072 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_24072.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_24072.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8654 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_24072_msk_1.map emd_24072_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: blurred map
| ファイル | emd_24072_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | blurred map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_24072_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_24072_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Complex of NHP-isolated Ab1245 Fab and bNAb 8ANC195 G52K5 Fab bou...
+超分子 #1: Complex of NHP-isolated Ab1245 Fab and bNAb 8ANC195 G52K5 Fab bou...
+超分子 #2: 8ANC195 G52K5 Fab
+超分子 #3: Ab1245 Fab
+超分子 #4: BG505 SOSIP.664 trimer
+分子 #1: 8ANC195 G52K5 Fab heavy chain
+分子 #2: 8ANC195 G52K5 Fab light chain
+分子 #3: HIV-1 Envelope Glycoprotein BG505 SOSIP.664 gp41
+分子 #4: Envelope glycoprotein BG505 SOSIP.664 gp120
+分子 #5: Ab1245 Fab heavy chain
+分子 #6: Ab1245 Fab light chain
+分子 #14: 2-acetamido-2-deoxy-beta-D-glucopyranose
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 4.7 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 8 構成要素:
| |||||||||
| グリッド | モデル: Quantifoil R2/2 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV 詳細: 0 blot force, 3 second blot time, 3 uL sample added. | |||||||||
| 詳細 | Ab1245 was incubated with BG505 SOSIP at room temperature for 3 hours, after which 8ANC195 Fab was added and the complex was incubated overnight at RT before SEC purification and concentration to 4.7 mg/mL. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 2307 / 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 精密化 | プロトコル: RIGID BODY FIT | ||||||||
| 得られたモデル |  PDB-7mxe: |
 ムービー
ムービー コントローラー
コントローラー















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)