+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23836 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
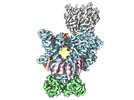
| タイトル | Human Hedgehog acyltransferase (HHAT) in complex with palmitoyl-CoA and two Fab antibody fragments | |||||||||
 マップデータ マップデータ | final map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報N-terminal peptidyl-L-cysteine N-palmitoylation / O-acyltransferase activity / HHAT G278V doesn't palmitoylate Hh-Np / palmitoyltransferase activity / smoothened signaling pathway / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / Hedgehog ligand biogenesis / Golgi membrane / endoplasmic reticulum membrane / GTP binding ...N-terminal peptidyl-L-cysteine N-palmitoylation / O-acyltransferase activity / HHAT G278V doesn't palmitoylate Hh-Np / palmitoyltransferase activity / smoothened signaling pathway / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / Hedgehog ligand biogenesis / Golgi membrane / endoplasmic reticulum membrane / GTP binding / Golgi apparatus / endoplasmic reticulum 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) / Homo sapiens (ヒト) /  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||
 データ登録者 データ登録者 | Long SB / Jiang Y | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Science / 年: 2021 ジャーナル: Science / 年: 2021タイトル: Substrate and product complexes reveal mechanisms of Hedgehog acylation by HHAT. 著者: Yiyang Jiang / Thomas L Benz / Stephen B Long /  要旨: Hedgehog proteins govern crucial developmental steps in animals and drive certain human cancers. Before they can function as signaling molecules, Hedgehog precursor proteins must undergo amino- ...Hedgehog proteins govern crucial developmental steps in animals and drive certain human cancers. Before they can function as signaling molecules, Hedgehog precursor proteins must undergo amino-terminal palmitoylation by Hedgehog acyltransferase (HHAT). We present cryo-electron microscopy structures of human HHAT in complex with its palmitoyl-coenzyme A substrate and of a product complex with a palmitoylated Hedgehog peptide at resolutions of 2.7 and 3.2 angstroms, respectively. The structures reveal how HHAT overcomes the challenges of bringing together substrates that have different physiochemical properties from opposite sides of the endoplasmic reticulum membrane within a membrane-embedded active site for catalysis. These principles are relevant to related enzymes that catalyze the acylation of Wnt and of the appetite-stimulating hormone ghrelin. The structural and mechanistic insights may advance the development of inhibitors for cancer. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23836.map.gz emd_23836.map.gz | 47.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23836-v30.xml emd-23836-v30.xml emd-23836.xml emd-23836.xml | 15.6 KB 15.6 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23836.png emd_23836.png | 157.5 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23836 http://ftp.pdbj.org/pub/emdb/structures/EMD-23836 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23836 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23836 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23836_validation.pdf.gz emd_23836_validation.pdf.gz | 350 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23836_full_validation.pdf.gz emd_23836_full_validation.pdf.gz | 349.6 KB | 表示 | |
| XML形式データ |  emd_23836_validation.xml.gz emd_23836_validation.xml.gz | 7.5 KB | 表示 | |
| CIF形式データ |  emd_23836_validation.cif.gz emd_23836_validation.cif.gz | 8.5 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23836 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23836 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23836 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23836 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23836.map.gz / 形式: CCP4 / 大きさ: 272.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23836.map.gz / 形式: CCP4 / 大きさ: 272.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | final map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.532 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
+全体 : HHAT in complex with palmitoyl-CoA and two Fab antibody fragments
+超分子 #1: HHAT in complex with palmitoyl-CoA and two Fab antibody fragments
+分子 #1: Protein-cysteine N-palmitoyltransferase HHAT
+分子 #2: 1C06 Fab heavy chain
+分子 #3: 1C06 Fab light chain
+分子 #4: 3H02 Fab heavy chain
+分子 #5: 3H02 Fab light chain
+分子 #6: PROTOPORPHYRIN IX CONTAINING FE
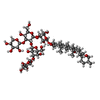
+分子 #7: Palmitoyl-CoA
+分子 #8: Digitonin
+分子 #9: water
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 26.5 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 2.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 174058 |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
 ムービー
ムービー コントローラー
コントローラー