+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23657 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
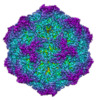
| タイトル | Canine parvovirus and Fab14 at partial occupancy | |||||||||
 マップデータ マップデータ | Canine parvovirus and Fab14 at partial occupancy | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | canine parvovirus / CPV / VIRUS | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont entry into host cell via permeabilization of host membrane / T=1 icosahedral viral capsid / clathrin-dependent endocytosis of virus by host cell / virion attachment to host cell / structural molecule activity 類似検索 - 分子機能 | |||||||||
| 生物種 |  Canine parvovirus type 2 (ウイルス) / Canine parvovirus type 2 (ウイルス) /  Canine parvovirus (ウイルス) Canine parvovirus (ウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.26 Å | |||||||||
 データ登録者 データ登録者 | Goteschius DJ / Hartmann SR | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2021 ジャーナル: Proc Natl Acad Sci U S A / 年: 2021タイトル: High-resolution asymmetric structure of a Fab-virus complex reveals overlap with the receptor binding site. 著者: Daniel J Goetschius / Samantha R Hartmann / Lindsey J Organtini / Heather Callaway / Kai Huang / Carol M Bator / Robert E Ashley / Alexander M Makhov / James F Conway / Colin R Parrish / Susan L Hafenstein /  要旨: Canine parvovirus is an important pathogen causing severe diseases in dogs, including acute hemorrhagic enteritis, myocarditis, and cerebellar disease. Overlap on the surface of parvovirus capsids ...Canine parvovirus is an important pathogen causing severe diseases in dogs, including acute hemorrhagic enteritis, myocarditis, and cerebellar disease. Overlap on the surface of parvovirus capsids between the antigenic epitope and the receptor binding site has contributed to cross-species transmission, giving rise to closely related variants. It has been shown that Mab 14 strongly binds and neutralizes canine but not feline parvovirus, suggesting this antigenic site also controls species-specific receptor binding. To visualize the conformational epitope at high resolution, we solved the cryogenic electron microscopy (cryo-EM) structure of the Fab-virus complex. We also created custom software, Icosahedral Subparticle Extraction and Correlated Classification, to solve a Fab-virus complex with only a few Fab bound per capsid and visualize local structures of the Fab-bound and -unbound antigenic sites extracted from the same complex map. Our results identified the antigenic epitope that had significant overlap with the receptor binding site, and the structures revealed that binding of Fab induced conformational changes to the virus. We were also able to assign the order and position of attached Fabs to allow assessment of complementarity between the Fabs bound to different positions. This approach therefore provides a method for using cryo-EM to investigate complementarity of antibody binding. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23657.map.gz emd_23657.map.gz | 323.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23657-v30.xml emd-23657-v30.xml emd-23657.xml emd-23657.xml | 13.7 KB 13.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23657.png emd_23657.png | 248.6 KB | ||
| Filedesc metadata |  emd-23657.cif.gz emd-23657.cif.gz | 5.2 KB | ||
| その他 |  emd_23657_half_map_1.map.gz emd_23657_half_map_1.map.gz emd_23657_half_map_2.map.gz emd_23657_half_map_2.map.gz | 297 MB 295.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23657 http://ftp.pdbj.org/pub/emdb/structures/EMD-23657 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23657 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23657 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23657_validation.pdf.gz emd_23657_validation.pdf.gz | 821 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23657_full_validation.pdf.gz emd_23657_full_validation.pdf.gz | 820.5 KB | 表示 | |
| XML形式データ |  emd_23657_validation.xml.gz emd_23657_validation.xml.gz | 17 KB | 表示 | |
| CIF形式データ |  emd_23657_validation.cif.gz emd_23657_validation.cif.gz | 20.4 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23657 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23657 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23657 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23657 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23657.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23657.map.gz / 形式: CCP4 / 大きさ: 371.3 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Canine parvovirus and Fab14 at partial occupancy | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
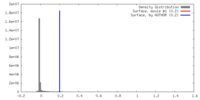
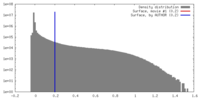
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: Half-map 1
| ファイル | emd_23657_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 1 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
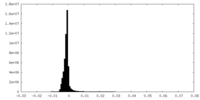
| 密度ヒストグラム |
-ハーフマップ: Half-map 2
| ファイル | emd_23657_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half-map 2 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
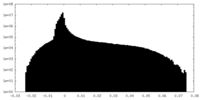
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Canine parvovirus
| 全体 | 名称:  Canine parvovirus (ウイルス) Canine parvovirus (ウイルス) |
|---|---|
| 要素 |
|
-超分子 #1: Canine parvovirus
| 超分子 | 名称: Canine parvovirus / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 10788 / 生物種: Canine parvovirus / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: No / ウイルス・中空状態: No |
|---|
-分子 #1: Capsid protein 2
| 分子 | 名称: Capsid protein 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Canine parvovirus type 2 (ウイルス) Canine parvovirus type 2 (ウイルス) |
| 分子量 | 理論値: 64.705559 KDa |
| 配列 | 文字列: MSDGAVQPDG GQPAVRNERA TGSGNGSGGG GGGGSGGVGI STGTFNNQTE FKFLENGWVE ITANSSRLVH LNMPESENYR RVVVNNMDK TAVNGNMALD DIHAQIVTPW SLVDANAWGV WFNPGDWQLI VNTMSELHLV SFEQEIFNVV LKTVSESATQ P PTKVYNND ...文字列: MSDGAVQPDG GQPAVRNERA TGSGNGSGGG GGGGSGGVGI STGTFNNQTE FKFLENGWVE ITANSSRLVH LNMPESENYR RVVVNNMDK TAVNGNMALD DIHAQIVTPW SLVDANAWGV WFNPGDWQLI VNTMSELHLV SFEQEIFNVV LKTVSESATQ P PTKVYNND LTASLMVALD SNNTMPFTPA AMRSETLGFY PWKPTIPTPW RYYFQWDRTL IPSHTGTSGT PTNIYHGTDP DD VQFYTIE NSVPVHLLRT GDEFATGTFF FDCKPCRLTH TWQTNRALGL PPFLNSLPQS EGATNFGDIG VQQDKRRGVT QMG NTNYIT EATIMRPAEV GYSAPYYSFE ASTQGPFKTP IAAGRGGAQT DENQAADGNP RYAFGRQHGQ KTTTTGETPE RFTY IAHQD TGRYPEGDWI QNINFNLPVT NDNVLLPTDP IGGKTGINYT NIFNTYGPLT ALNNVPPVYP NGQIWDKEFD TDLKP RLHV NAPFVCQNNC PGQLFVKVAP NLTNEYDPDA SANMSRIVTY SDFWWKGKLV FKAKLRASHT WNPIQQMSIN VDNQFN YVP SNIGGMKIVY EKSQLAPRKL Y UniProtKB: Capsid protein 2 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 2.26 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 162627 |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: OTHER |
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)