+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23611 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM reconstruction of Grafix-stabilized PaFS octameric prenyltransferase domain | |||||||||
 マップデータ マップデータ | Unsharpened, asymmetric octameric core prenyltransferase domain of Grafix-stabilized PaFS with particles subjected to partial signal subtraction | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報fusicocca-2,10(14)-diene synthase / alcohol biosynthetic process / mycotoxin biosynthetic process / geranylgeranyl diphosphate synthase / geranylgeranyl diphosphate synthase activity / isoprenoid biosynthetic process / lyase activity / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  Diaporthe amygdali (菌類) Diaporthe amygdali (菌類) | |||||||||
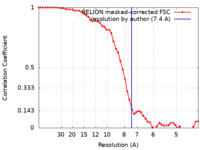
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 7.4 Å | |||||||||
 データ登録者 データ登録者 | Faylo JL / van Eeuwen T / Murakami K / Christianson DW | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Structural insight on assembly-line catalysis in terpene biosynthesis. 著者: Jacque L Faylo / Trevor van Eeuwen / Hee Jong Kim / Jose J Gorbea Colón / Benjamin A Garcia / Kenji Murakami / David W Christianson /  要旨: Fusicoccadiene synthase from Phomopsis amygdali (PaFS) is a unique bifunctional terpenoid synthase that catalyzes the first two steps in the biosynthesis of the diterpene glycoside Fusicoccin A, a ...Fusicoccadiene synthase from Phomopsis amygdali (PaFS) is a unique bifunctional terpenoid synthase that catalyzes the first two steps in the biosynthesis of the diterpene glycoside Fusicoccin A, a mediator of 14-3-3 protein interactions. The prenyltransferase domain of PaFS generates geranylgeranyl diphosphate, which the cyclase domain then utilizes to generate fusicoccadiene, the tricyclic hydrocarbon skeleton of Fusicoccin A. Here, we use cryo-electron microscopy to show that the structure of full-length PaFS consists of a central octameric core of prenyltransferase domains, with the eight cyclase domains radiating outward via flexible linker segments in variable splayed-out positions. Cryo-electron microscopy and chemical crosslinking experiments additionally show that compact conformations can be achieved in which cyclase domains are more closely associated with the prenyltransferase core. This structural analysis provides a framework for understanding substrate channeling, since most of the geranylgeranyl diphosphate generated by the prenyltransferase domains remains on the enzyme for cyclization to form fusicoccadiene. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23611.map.gz emd_23611.map.gz | 7.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23611-v30.xml emd-23611-v30.xml emd-23611.xml emd-23611.xml | 17.7 KB 17.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23611_fsc.xml emd_23611_fsc.xml | 5.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23611.png emd_23611.png | 115.9 KB | ||
| マスクデータ |  emd_23611_msk_1.map emd_23611_msk_1.map | 10.5 MB |  マスクマップ マスクマップ | |
| その他 |  emd_23611_half_map_1.map.gz emd_23611_half_map_1.map.gz emd_23611_half_map_2.map.gz emd_23611_half_map_2.map.gz | 7.3 MB 7.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23611 http://ftp.pdbj.org/pub/emdb/structures/EMD-23611 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23611 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23611 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23611_validation.pdf.gz emd_23611_validation.pdf.gz | 462.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23611_full_validation.pdf.gz emd_23611_full_validation.pdf.gz | 461.9 KB | 表示 | |
| XML形式データ |  emd_23611_validation.xml.gz emd_23611_validation.xml.gz | 11.2 KB | 表示 | |
| CIF形式データ |  emd_23611_validation.cif.gz emd_23611_validation.cif.gz | 14 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23611 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23611 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23611 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23611 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23611.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23611.map.gz / 形式: CCP4 / 大きさ: 10.5 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened, asymmetric octameric core prenyltransferase domain of Grafix-stabilized PaFS with particles subjected to partial signal subtraction | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.16 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
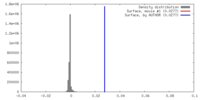
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23611_msk_1.map emd_23611_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
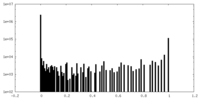
| 密度ヒストグラム |
-ハーフマップ: Unsharpened, unfiltered half-map of asymmetric octameric core prenyltransferase...
| ファイル | emd_23611_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened, unfiltered half-map of asymmetric octameric core prenyltransferase domain of Grafix-stabilized PaFS with particles subjected to partial signal subtraction | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
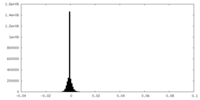
| 密度ヒストグラム |
-ハーフマップ: Unsharpened, unfiltered half-map of asymmetric octameric core prenyltransferase...
| ファイル | emd_23611_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Unsharpened, unfiltered half-map of asymmetric octameric core prenyltransferase domain of Grafix-stabilized PaFS with particles subjected to partial signal subtraction | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
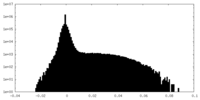
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Apo octameric core prenyltransferase domain of PaFS
| 全体 | 名称: Apo octameric core prenyltransferase domain of PaFS |
|---|---|
| 要素 |
|
-超分子 #1: Apo octameric core prenyltransferase domain of PaFS
| 超分子 | 名称: Apo octameric core prenyltransferase domain of PaFS / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all 詳細: Reconstruction of single domain of full-length complex was calculated by partial signal subtraction. Refinement imposed C2 symmetry. |
|---|---|
| 由来(天然) | 生物種:  Diaporthe amygdali (菌類) Diaporthe amygdali (菌類) |
| 組換発現 | 生物種:  |
| 分子量 | 実験値: 392 KDa |
-分子 #1: Fusicoccadiene synthase from Phomopsis amygdali
| 分子 | 名称: Fusicoccadiene synthase from Phomopsis amygdali / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO / EC番号: fusicocca-2,10(14)-diene synthase |
|---|---|
| 由来(天然) | 生物種:  Diaporthe amygdali (菌類) Diaporthe amygdali (菌類) |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MGSSHHHHHH SSGLVPRGSH MEFKYSEVVE PSTYYTEGLC EGIDVRKSKF TTLEDRGAIR AHEDWNKHIG PCREYRGTLG PRFSFISVAV PECIPERLEV ISYANEFAFL HDDVTDHVGH DTGEVENDEM MTVFLEAAHT GAIDTSNKVD IRRAGKKRIQ SQLFLEMLAI ...文字列: MGSSHHHHHH SSGLVPRGSH MEFKYSEVVE PSTYYTEGLC EGIDVRKSKF TTLEDRGAIR AHEDWNKHIG PCREYRGTLG PRFSFISVAV PECIPERLEV ISYANEFAFL HDDVTDHVGH DTGEVENDEM MTVFLEAAHT GAIDTSNKVD IRRAGKKRIQ SQLFLEMLAI DPECAKTTMK SWARFVEVGS SRQHETRFVE LAKYIPYRIM DVGEMFWFGL VTFGLGLHIP DHELELCREL MANAWIAVGL QNDIWSWPKE RDAATLHGKD HVVNAIWVLM QEHQTDVDGA MQICRKLIVE YVAKYLEVIE ATKNDESISL DLRKYLDAML YSISGNVVWS LECPRYNPDV SFNKTQLEWM RQGLPSLESC PVLARSPEID SDESAVSPTA DESDSTEDSL GSGSRQDSSL STGLSLSPVH SNEGKDLQRV DTDHIFFEKA VLEAPYDYIA SMPSKGVRDQ FIDALNDWLR VPDVKVGKIK DAVRVLHNSS LLLDDFQDNS PLRRGKPSTH NIFGSAQTVN TATYSIIKAI GQIMEFSAGE SVQEVMNSIM ILFQGQAMDL FWTYNGHVPS EEEYYRMIDQ KTGQLFSIAT SLLLNAADNE IPRTKIQSCL HRLTRLLGRC FQICDDYQNL VSADYTKQKG FCEDLDEGKW SLALIHMIHK QRSHMALLNV LSTGRKHGGM TLEQKQFVLD IIEEEKSLDY TRSVMMDLHV QLRAEIGRIE ILLDSPNPAM RLLLELLRV |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 2 mg/mL |
|---|---|
| 緩衝液 | pH: 7.7 |
| 凍結 | 凍結剤: ETHANE |
| 詳細 | PaFS was subjected to gradient fixation using glutaraldehyde |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 6544 / 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: AB INITIO MODEL |
|---|
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)