+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23311 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Synechocystis sp. UTEX2470 Cyanophycin synthetase 1 with ATP | |||||||||
 マップデータ マップデータ | Synechocystis sp. UTEX2470 CphA1 with ATP | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | cyanophycin / CphA1 / ATP grasp / ligase | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cyanophycin synthase (L-aspartate-adding) / cyanophycin synthase (L-arginine-adding) / cyanophycin synthetase activity (L-aspartate-adding) / cyanophycin synthetase activity (L-arginine-adding) / biosynthetic process / ATP binding / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |   | |||||||||
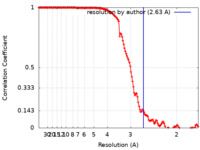
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 2.63 Å | |||||||||
 データ登録者 データ登録者 | Sharon I / Schmeing TM | |||||||||
 引用 引用 |  ジャーナル: Nat Chem Biol / 年: 2021 ジャーナル: Nat Chem Biol / 年: 2021タイトル: Structures and function of the amino acid polymerase cyanophycin synthetase. 著者: Itai Sharon / Asfarul S Haque / Marcel Grogg / Indrajit Lahiri / Dieter Seebach / Andres E Leschziner / Donald Hilvert / T Martin Schmeing /    要旨: Cyanophycin is a natural biopolymer produced by a wide range of bacteria, consisting of a chain of poly-L-Asp residues with L-Arg residues attached to the β-carboxylate sidechains by isopeptide ...Cyanophycin is a natural biopolymer produced by a wide range of bacteria, consisting of a chain of poly-L-Asp residues with L-Arg residues attached to the β-carboxylate sidechains by isopeptide bonds. Cyanophycin is synthesized from ATP, aspartic acid and arginine by a homooligomeric enzyme called cyanophycin synthetase (CphA1). CphA1 has domains that are homologous to glutathione synthetases and muramyl ligases, but no other structural information has been available. Here, we present cryo-electron microscopy and X-ray crystallography structures of cyanophycin synthetases from three different bacteria, including cocomplex structures of CphA1 with ATP and cyanophycin polymer analogs at 2.6 Å resolution. These structures reveal two distinct tetrameric architectures, show the configuration of active sites and polymer-binding regions, indicate dynamic conformational changes and afford insight into catalytic mechanism. Accompanying biochemical interrogation of substrate binding sites, catalytic centers and oligomerization interfaces combine with the structures to provide a holistic understanding of cyanophycin biosynthesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23311.map.gz emd_23311.map.gz | 122.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23311-v30.xml emd-23311-v30.xml emd-23311.xml emd-23311.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23311_fsc.xml emd_23311_fsc.xml | 13.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23311.png emd_23311.png | 138.7 KB | ||
| マスクデータ |  emd_23311_msk_1.map emd_23311_msk_1.map | 244.1 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-23311.cif.gz emd-23311.cif.gz | 5.8 KB | ||
| その他 |  emd_23311_half_map_1.map.gz emd_23311_half_map_1.map.gz emd_23311_half_map_2.map.gz emd_23311_half_map_2.map.gz | 226.4 MB 226.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23311 http://ftp.pdbj.org/pub/emdb/structures/EMD-23311 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23311 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23311 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23311_validation.pdf.gz emd_23311_validation.pdf.gz | 945.3 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23311_full_validation.pdf.gz emd_23311_full_validation.pdf.gz | 944.8 KB | 表示 | |
| XML形式データ |  emd_23311_validation.xml.gz emd_23311_validation.xml.gz | 22.3 KB | 表示 | |
| CIF形式データ |  emd_23311_validation.cif.gz emd_23311_validation.cif.gz | 28.7 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23311 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23311 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23311 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23311 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23311.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23311.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
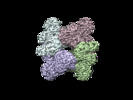
| 注釈 | Synechocystis sp. UTEX2470 CphA1 with ATP | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.855 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-マスク #1
| ファイル |  emd_23311_msk_1.map emd_23311_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Synechocystis sp. UTEX2470 CphA1 with ATP half map A
| ファイル | emd_23311_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Synechocystis sp. UTEX2470 CphA1 with ATP half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Synechocystis sp. UTEX2470 CphA1 with ATP half map B
| ファイル | emd_23311_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Synechocystis sp. UTEX2470 CphA1 with ATP half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cyanophycin synthetase 1 with Atp
| 全体 | 名称: Cyanophycin synthetase 1 with Atp |
|---|---|
| 要素 |
|
-超分子 #1: Cyanophycin synthetase 1 with Atp
| 超分子 | 名称: Cyanophycin synthetase 1 with Atp / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Cyanophycin synthase
| 分子 | 名称: Cyanophycin synthase / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO / EC番号: cyanophycin synthase (L-aspartate-adding) |
|---|---|
| 由来(天然) | 生物種:  株: PCC 6714 |
| 分子量 | 理論値: 95.758836 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: MKILKTLTLR GPNYWSIRRK KLIVMRLDLE DLAERPSNSI PGFYEGLIKV LPSLVEHFCS PGYQGGFLER VKEGTYMGHI VEHVALELQ ELVGMTAGFG RTRETSTPGV YNVVYEYVDE QAGRYAGRAA VRLCRSLVDT GDYPRLELEK DLEDLRDLGA N SALGPSTE ...文字列: MKILKTLTLR GPNYWSIRRK KLIVMRLDLE DLAERPSNSI PGFYEGLIKV LPSLVEHFCS PGYQGGFLER VKEGTYMGHI VEHVALELQ ELVGMTAGFG RTRETSTPGV YNVVYEYVDE QAGRYAGRAA VRLCRSLVDT GDYPRLELEK DLEDLRDLGA N SALGPSTE TIVTEAEARK IPWMLLSARA MVQLGYGVYQ QRIQATLSSH SGILGVELAC DKEGTKTILQ DAGIPVPRGT TI QYFDDLE EAINDVGGYP VVIKPLDGNH GRGITINVRH WQEAIAAYDL AAEESKSRAI IVERYYEGSD HRVLVVNGKL VAV AERIPA HVTGDGSSTI SELIEKTNQD PNRGDGHDNI LTKIVVNKTA IDVMERQGYN LDSVLPKDEV VYLRATANLS TGGI AIDRT DDIHPENIWL MERVAKVIGL DIAGIDVVTS DISKPLRETN GVIVEVNAAP GFRMHVAPSQ GLPRNVAAPV LDMLF PPGT PSRIPILAVT GTNGKTTTTR LLAHIYRQTG KTVGYTSTDA IYINEYCVEK GDNTGPQSAG VILRDPTVEV AVLETA RGG ILRAGLAFDS CDVGVVLNVA ADHLGLGDID TIEQMAKVKS VIAEVVDPSG YAVLNADDPL VAAMADKVKA KVAYFSM NP DNPIIQAHVR RNGIAAVYES GYLSILEGSW TLRVEQAKLI PMTMGGMAPF MIANALAACL AAFVNGLDVE VIRQGVRT F TTSAEQTPGR MNLFNLGQHH ALVDYAHNPA GYRAVGDFVK NWQGQRFGVV GGPGDRRDSD LIELGQIAAQ VFDRIIVKE DDDKRGRSEG ETADLIVKGI LQENPGASYE VILDETIALN KALDQVEEKG LVVVFPESVT RAIDLIKVRN PIGENLYFQ UniProtKB: Cyanophycin synthetase |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 8 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information |  ChemComp-ATP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 12 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: C-flat-1.2/1.3 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 20 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)