+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23170 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Dihedral ring D2_Wm-01_trunc | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 生物種 | synthetic construct (人工物) | |||||||||
| 手法 | 単粒子再構成法 / ネガティブ染色法 / 解像度: 19.0 Å | |||||||||
 データ登録者 データ登録者 | Park YJ / Veesler D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Design of multi-scale protein complexes by hierarchical building block fusion. 著者: Yang Hsia / Rubul Mout / William Sheffler / Natasha I Edman / Ivan Vulovic / Young-Jun Park / Rachel L Redler / Matthew J Bick / Asim K Bera / Alexis Courbet / Alex Kang / T J Brunette / Una ...著者: Yang Hsia / Rubul Mout / William Sheffler / Natasha I Edman / Ivan Vulovic / Young-Jun Park / Rachel L Redler / Matthew J Bick / Asim K Bera / Alexis Courbet / Alex Kang / T J Brunette / Una Nattermann / Evelyn Tsai / Ayesha Saleem / Cameron M Chow / Damian Ekiert / Gira Bhabha / David Veesler / David Baker /  要旨: A systematic and robust approach to generating complex protein nanomaterials would have broad utility. We develop a hierarchical approach to designing multi-component protein assemblies from two ...A systematic and robust approach to generating complex protein nanomaterials would have broad utility. We develop a hierarchical approach to designing multi-component protein assemblies from two classes of modular building blocks: designed helical repeat proteins (DHRs) and helical bundle oligomers (HBs). We first rigidly fuse DHRs to HBs to generate a large library of oligomeric building blocks. We then generate assemblies with cyclic, dihedral, and point group symmetries from these building blocks using architecture guided rigid helical fusion with new software named WORMS. X-ray crystallography and cryo-electron microscopy characterization show that the hierarchical design approach can accurately generate a wide range of assemblies, including a 43 nm diameter icosahedral nanocage. The computational methods and building block sets described here provide a very general route to de novo designed protein nanomaterials. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23170.map.gz emd_23170.map.gz | 1.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23170-v30.xml emd-23170-v30.xml emd-23170.xml emd-23170.xml | 17.5 KB 17.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_23170.png emd_23170.png | 69.5 KB | ||
| その他 |  emd_23170_additional_1.map.gz emd_23170_additional_1.map.gz emd_23170_half_map_1.map.gz emd_23170_half_map_1.map.gz emd_23170_half_map_2.map.gz emd_23170_half_map_2.map.gz | 1018.9 KB 1.8 MB 1.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23170 http://ftp.pdbj.org/pub/emdb/structures/EMD-23170 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23170 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23170 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23170_validation.pdf.gz emd_23170_validation.pdf.gz | 361 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23170_full_validation.pdf.gz emd_23170_full_validation.pdf.gz | 360.5 KB | 表示 | |
| XML形式データ |  emd_23170_validation.xml.gz emd_23170_validation.xml.gz | 6.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23170 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23170 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23170 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23170 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23170.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23170.map.gz / 形式: CCP4 / 大きさ: 2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 6.4 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: #1
| ファイル | emd_23170_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
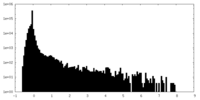
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_23170_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
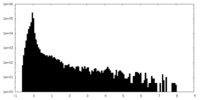
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_23170_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
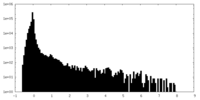
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : D2_Wm-01_trunc
| 全体 | 名称: D2_Wm-01_trunc |
|---|---|
| 要素 |
|
-超分子 #1: D2_Wm-01_trunc
| 超分子 | 名称: D2_Wm-01_trunc / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 組換発現 | 生物種:  |
-分子 #1: D2_Wm-01_truncA
| 分子 | 名称: D2_Wm-01_truncA / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: MGTREESLKE QLRSLREQAE LAARLLRLQR EGSSDEDVKE LVAEIIKLIM EQLLLIAEQL LGRS EAAEL ALRAIRLALE LCRQSTDLEE CLRLLKTAIK ALENALRHPD STTAKARLMA ITARLLAQQ LRTQHPDSQA ARDAEKLADQ AERAVRLATR LYEEHPNAEI ...文字列: MGTREESLKE QLRSLREQAE LAARLLRLQR EGSSDEDVKE LVAEIIKLIM EQLLLIAEQL LGRS EAAEL ALRAIRLALE LCRQSTDLEE CLRLLKTAIK ALENALRHPD STTAKARLMA ITARLLAQQ LRTQHPDSQA ARDAEKLADQ AERAVRLATR LYEEHPNAEI SEMCSQAAYA AALMASIAAI LAQR HPDSQ IARDLIRLAS ELAEMVKRMC ERGGSWGLEH HHHHH |
-分子 #2: D2_Wm-01_truncB
| 分子 | 名称: D2_Wm-01_truncB / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 配列 | 文字列: MGTREELAKE LLRSLREQAE SLARQLRLQR EGSSDEDVKE LAAEQIKLIM EQLLLIAELT LGRS EAAEL ALDAIRQALE ACRTMDNQEA CTRLLKLAIQ MLELATRAPD AEAAKLALEA AKKAIELAN RHPGSQAAED ATKLAQQAME AVRLALKLYE EHPNADIADL ...文字列: MGTREELAKE LLRSLREQAE SLARQLRLQR EGSSDEDVKE LAAEQIKLIM EQLLLIAELT LGRS EAAEL ALDAIRQALE ACRTMDNQEA CTRLLKLAIQ MLELATRAPD AEAAKLALEA AKKAIELAN RHPGSQAAED ATKLAQQAME AVRLALKLYE EHPNADIADL CRRAAAEAAE AASKAAELAQ RHPD SQAAR DAIKLASQAA EAVKLACELA QEHPNADKAK LCILLASAAA LLASIAAMLA QRHPDSQEA RDMIRIASEL AELVKEICER |
-実験情報
-構造解析
| 手法 | ネガティブ染色法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 染色 | タイプ: NEGATIVE / 材質: UF |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI 12 |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 50.0 e/Å2 |
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER / 詳細: cryoSPARC ab initio |
|---|---|
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 19.0 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 65970 |
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
 ムービー
ムービー コントローラー
コントローラー



 UCSF Chimera
UCSF Chimera




















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)