+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-23135 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of RTX-bound full-length TRPV1 at pH 5.5 | ||||||||||||||||||
 マップデータ マップデータ | RTX-bound full-length TRPV1 at pH 5.5 | ||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報temperature-gated ion channel activity / response to capsazepine / negative regulation of establishment of blood-brain barrier / sensory perception of mechanical stimulus / peptide secretion / urinary bladder smooth muscle contraction / detection of chemical stimulus involved in sensory perception of pain / smooth muscle contraction involved in micturition / TRP channels / cellular response to temperature stimulus ...temperature-gated ion channel activity / response to capsazepine / negative regulation of establishment of blood-brain barrier / sensory perception of mechanical stimulus / peptide secretion / urinary bladder smooth muscle contraction / detection of chemical stimulus involved in sensory perception of pain / smooth muscle contraction involved in micturition / TRP channels / cellular response to temperature stimulus / cellular response to acidic pH / fever generation / detection of temperature stimulus involved in thermoception / thermoception / negative regulation of systemic arterial blood pressure / glutamate secretion / chloride channel regulator activity / response to pH / dendritic spine membrane / monoatomic cation transmembrane transporter activity / excitatory extracellular ligand-gated monoatomic ion channel activity / negative regulation of heart rate / cellular response to ATP / temperature homeostasis / response to pain / cellular response to alkaloid / calcium ion import across plasma membrane / behavioral response to pain / diet induced thermogenesis / cellular response to cytokine stimulus / detection of temperature stimulus involved in sensory perception of pain / intracellularly gated calcium channel activity / negative regulation of mitochondrial membrane potential / ligand-gated monoatomic ion channel activity / extracellular ligand-gated monoatomic ion channel activity / monoatomic cation channel activity / GABA-ergic synapse / sensory perception of pain / phosphatidylinositol binding / cellular response to nerve growth factor stimulus / phosphoprotein binding / calcium ion transmembrane transport / microglial cell activation / calcium channel activity / lipid metabolic process / cellular response to growth factor stimulus / response to peptide hormone / calcium ion transport / positive regulation of nitric oxide biosynthetic process / transmembrane signaling receptor activity / cellular response to tumor necrosis factor / cellular response to heat / positive regulation of cytosolic calcium ion concentration / response to heat / monoatomic ion transmembrane transport / postsynaptic membrane / protein homotetramerization / calmodulin binding / neuron projection / positive regulation of apoptotic process / external side of plasma membrane / neuronal cell body / dendrite / negative regulation of transcription by RNA polymerase II / ATP binding / identical protein binding / membrane / metal ion binding / plasma membrane 類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |  | ||||||||||||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.64 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Zhang K / Julius D / Cheng Y | ||||||||||||||||||
| 資金援助 |  フランス, フランス,  米国, 5件 米国, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2021 ジャーナル: Cell / 年: 2021タイトル: Structural snapshots of TRPV1 reveal mechanism of polymodal functionality. 著者: Kaihua Zhang / David Julius / Yifan Cheng /  要旨: Many transient receptor potential (TRP) channels respond to diverse stimuli and conditionally conduct small and large cations. Such functional plasticity is presumably enabled by a uniquely dynamic ...Many transient receptor potential (TRP) channels respond to diverse stimuli and conditionally conduct small and large cations. Such functional plasticity is presumably enabled by a uniquely dynamic ion selectivity filter that is regulated by physiological agents. What is currently missing is a "photo series" of intermediate structural states that directly address this hypothesis and reveal specific mechanisms behind such dynamic channel regulation. Here, we exploit cryoelectron microscopy (cryo-EM) to visualize conformational transitions of the capsaicin receptor, TRPV1, as a model to understand how dynamic transitions of the selectivity filter in response to algogenic agents, including protons, vanilloid agonists, and peptide toxins, permit permeation by small and large organic cations. These structures also reveal mechanisms governing ligand binding substates, as well as allosteric coupling between key sites that are proximal to the selectivity filter and cytoplasmic gate. These insights suggest a general framework for understanding how TRP channels function as polymodal signal integrators. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_23135.map.gz emd_23135.map.gz | 40.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-23135-v30.xml emd-23135-v30.xml emd-23135.xml emd-23135.xml | 15.5 KB 15.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_23135_fsc.xml emd_23135_fsc.xml | 7.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_23135.png emd_23135.png | 245.9 KB | ||
| その他 |  emd_23135_half_map_1.map.gz emd_23135_half_map_1.map.gz emd_23135_half_map_2.map.gz emd_23135_half_map_2.map.gz | 29.7 MB 29.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-23135 http://ftp.pdbj.org/pub/emdb/structures/EMD-23135 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23135 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-23135 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_23135_validation.pdf.gz emd_23135_validation.pdf.gz | 485.2 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_23135_full_validation.pdf.gz emd_23135_full_validation.pdf.gz | 484.8 KB | 表示 | |
| XML形式データ |  emd_23135_validation.xml.gz emd_23135_validation.xml.gz | 14.2 KB | 表示 | |
| CIF形式データ |  emd_23135_validation.cif.gz emd_23135_validation.cif.gz | 18.6 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23135 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23135 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23135 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-23135 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7l2oMC  7l2hC  7l2iC  7l2jC  7l2kC  7l2lC  7l2mC  7l2nC  7l2pC  7l2rC  7l2sC  7l2tC  7l2uC  7l2vC  7l2wC  7l2xC  7mz5C  7mz6C  7mz7C  7mz9C  7mzaC  7mzbC  7mzcC  7mzdC  7mzeC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_23135.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_23135.map.gz / 形式: CCP4 / 大きさ: 42.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RTX-bound full-length TRPV1 at pH 5.5 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-ハーフマップ: RTX-bound full-length TRPV1 at pH 5.5
| ファイル | emd_23135_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RTX-bound full-length TRPV1 at pH 5.5 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: RTX-bound full-length TRPV1 at pH 5.5
| ファイル | emd_23135_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | RTX-bound full-length TRPV1 at pH 5.5 | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : full-length TRPV1 in nanodisc
| 全体 | 名称: full-length TRPV1 in nanodisc |
|---|---|
| 要素 |
|
-超分子 #1: full-length TRPV1 in nanodisc
| 超分子 | 名称: full-length TRPV1 in nanodisc / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Transient receptor potential cation channel subfamily V member 1
| 分子 | 名称: Transient receptor potential cation channel subfamily V member 1 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 95.328156 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GAMGSEQRAS LDSEESESPP QENSCLDPPD RDPNCKPPPV KPHIFTTRSR TRLFGKGDSE EASPLDCPYE EGGLASCPII TVSSVLTIQ RPGDGPASVR PSSQDSVSAG EKPPRLYDRR SIFDAVAQSN CQELESLLPF LQRSKKRLTD SEFKDPETGK T CLLKAMLN ...文字列: GAMGSEQRAS LDSEESESPP QENSCLDPPD RDPNCKPPPV KPHIFTTRSR TRLFGKGDSE EASPLDCPYE EGGLASCPII TVSSVLTIQ RPGDGPASVR PSSQDSVSAG EKPPRLYDRR SIFDAVAQSN CQELESLLPF LQRSKKRLTD SEFKDPETGK T CLLKAMLN LHNGQNDTIA LLLDVARKTD SLKQFVNASY TDSYYKGQTA LHIAIERRNM TLVTLLVENG ADVQAAANGD FF KKTKGRP GFYFGELPLS LAACTNQLAI VKFLLQNSWQ PADISARDSV GNTVLHALVE VADNTVDNTK FVTSMYNEIL ILG AKLHPT LKLEEITNRK GLTPLALAAS SGKIGVLAYI LQREIHEPEC RHLSRKFTEW AYGPVHSSLY DLSCIDTCEK NSVL EVIAY SSSETPNRHD MLLVEPLNRL LQDKWDRFVK RIFYFNFFVY CLYMIIFTAA AYYRPVEGLP PYKLKNTVGD YFRVT GEIL SVSGGVYFFF RGIQYFLQRR PSLKSLFVDS YSEILFFVQS LFMLVSVVLY FSQRKEYVAS MVFSLAMGWT NMLYYT RGF QQMGIYAVMI EKMILRDLCR FMFVYLVFLF GFSTAVVTLI EDGKNNSLPM ESTPHKCRGS ACKPGNSYNS LYSTCLE LF KFTIGMGDLE FTENYDFKAV FIILLLAYVI LTYILLLNML IALMGETVNK IAQESKNIWK LQRAITILDT EKSFLKCM R KAFRSGKLLQ VGFTPDGKDD YRWCFRVDEV NWTTWNTNVG IINEDPGNCE GVKRTLSFSL RSGRVSGRNW KNFALVPLL RDASTRDRHA TQQEEVQLKH YTGSLKPEDA EVFKDSMVPG EK |
-分子 #2: resiniferatoxin
| 分子 | 名称: resiniferatoxin / タイプ: ligand / ID: 2 / コピー数: 4 / 式: 6EU |
|---|---|
| 分子量 | 理論値: 628.708 Da |
| Chemical component information | 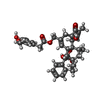 ChemComp-6EU: |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 5.5 |
|---|---|
| 糖包埋 | 材質: nanodisc |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TALOS ARCTICA |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 64.0 e/Å2 |
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD |
| 実験機器 |  モデル: Talos Arctica / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

































 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)