| 登録情報 | データベース: EMDB / ID: EMD-22919
|
|---|
| タイトル | Full-length human mitochondrial Hsp90 (TRAP1) SpyCatcher/SpyTag-SdhB heterodimer in the presence of AMP-PNP |
|---|
 マップデータ マップデータ | |
|---|
 試料 試料 | - 複合体: Full-length human mitochondrial Hsp90 (TRAP1) SpyCatcher/SpyTag-SdhB heterodimer in the presence of AMP-PNP
- タンパク質・ペプチド: Heat shock protein 75 kDa, mitochondrial, SpyCatcher
- タンパク質・ペプチド: Heat shock protein 75 kDa, mitochondrial, SpyTag, Succinate dehydrogenase [ubiquinone] iron-sulfur subunit, mitochondrial (chimera)
- リガンド: PHOSPHOAMINOPHOSPHONIC ACID-ADENYLATE ESTER
- リガンド: MAGNESIUM ION
- リガンド: POTASSIUM ION
|
|---|
 キーワード キーワード | Hsp90 / CHAPERONE / TRAP1 / SdhB / mitochondria / SpyTag / SpyCatcher |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
translational attenuation / negative regulation of cellular respiration / 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; キノンあるいはその類似化合物を用いる / succinate metabolic process / respiratory chain complex II (succinate dehydrogenase) / mitochondrial electron transport, succinate to ubiquinone / Citric acid cycle (TCA cycle) / succinate dehydrogenase (quinone) activity / succinate dehydrogenase / Maturation of TCA enzymes and regulation of TCA cycle ...translational attenuation / negative regulation of cellular respiration / 酸化還元酵素; CH-OHの結合に対し酸化酵素として働く; キノンあるいはその類似化合物を用いる / succinate metabolic process / respiratory chain complex II (succinate dehydrogenase) / mitochondrial electron transport, succinate to ubiquinone / Citric acid cycle (TCA cycle) / succinate dehydrogenase (quinone) activity / succinate dehydrogenase / Maturation of TCA enzymes and regulation of TCA cycle / Respiratory electron transport / tumor necrosis factor receptor binding / negative regulation of intrinsic apoptotic signaling pathway in response to hydrogen peroxide / 3 iron, 4 sulfur cluster binding / ubiquinone binding / proton motive force-driven mitochondrial ATP synthesis / negative regulation of reactive oxygen species biosynthetic process / : / tricarboxylic acid cycle / aerobic respiration / respiratory electron transport chain / ATP-dependent protein folding chaperone / mitochondrial membrane / mitochondrial intermembrane space / 2 iron, 2 sulfur cluster binding / unfolded protein binding / protein folding / 4 iron, 4 sulfur cluster binding / electron transfer activity / mitochondrial inner membrane / mitochondrial matrix / protein kinase binding / ATP hydrolysis activity / mitochondrion / RNA binding / nucleoplasm / ATP binding / metal ion binding / membrane / plasma membrane類似検索 - 分子機能 : / 4Fe-4S dicluster domain / Succinate dehydrogenase/fumarate reductase iron-sulphur protein / Succinate dehydogenase/fumarate reductase N-terminal / 2Fe-2S iron-sulfur cluster binding domain / Alpha-helical ferredoxin / 2Fe-2S ferredoxin, iron-sulphur binding site / 2Fe-2S ferredoxin-type iron-sulfur binding region signature. / HSP90, C-terminal domain / Heat shock protein Hsp90, N-terminal ...: / 4Fe-4S dicluster domain / Succinate dehydrogenase/fumarate reductase iron-sulphur protein / Succinate dehydogenase/fumarate reductase N-terminal / 2Fe-2S iron-sulfur cluster binding domain / Alpha-helical ferredoxin / 2Fe-2S ferredoxin, iron-sulphur binding site / 2Fe-2S ferredoxin-type iron-sulfur binding region signature. / HSP90, C-terminal domain / Heat shock protein Hsp90, N-terminal / Heat shock protein Hsp90 family / Hsp90 protein / Histidine kinase-, DNA gyrase B-, and HSP90-like ATPase / Beta-grasp domain superfamily / 2Fe-2S ferredoxin-type iron-sulfur binding domain profile. / 2Fe-2S ferredoxin-type iron-sulfur binding domain / 2Fe-2S ferredoxin-like superfamily / 4Fe-4S ferredoxin, iron-sulphur binding, conserved site / 4Fe-4S ferredoxin-type iron-sulfur binding region signature. / 4Fe-4S ferredoxin-type iron-sulfur binding domain profile. / 4Fe-4S ferredoxin-type, iron-sulphur binding domain / Histidine kinase-like ATPases / Histidine kinase/HSP90-like ATPase superfamily / Ribosomal protein S5 domain 2-type fold類似検索 - ドメイン・相同性 Succinate dehydrogenase [ubiquinone] iron-sulfur subunit, mitochondrial / Heat shock protein 75 kDa, mitochondrial類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) |
|---|
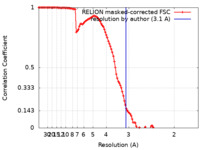
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.1 Å |
|---|
 データ登録者 データ登録者 | Liu YX / Agard DA |
|---|
| 資金援助 |  米国, 6件 米国, 6件 | Organization | Grant number | 国 |
|---|
| Howard Hughes Medical Institute (HHMI) | |  米国 米国 | | American Heart Association | 18POST33990362 |  米国 米国 | | National Institutes of Health/National Institute of General Medical Sciences (NIH/NIGMS) | R35GM118099 |  米国 米国 | | National Institutes of Health/National Cancer Institute (NIH/NCI) | U54CA209891 |  米国 米国 | | National Institutes of Health/National Institute of Mental Health (NIH/NIMH) | U01MH115747 |  米国 米国 | | National Institutes of Health/National Institute Of Allergy and Infectious Diseases (NIH/NIAID) | U19AI135990 |  米国 米国 |
|
|---|
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Published
タイトル: Cryo-EM analysis of human mitochondrial Hsp90 in multiple tetrameric states
著者: Liu YX / Agard DA / Elnatan D / Sun M / Larson AG |
|---|
| 履歴 | | 登録 | 2020年11月1日 | - |
|---|
| ヘッダ(付随情報) 公開 | 2021年11月3日 | - |
|---|
| マップ公開 | 2021年11月3日 | - |
|---|
| 更新 | 2025年6月4日 | - |
|---|
| 現状 | 2025年6月4日 | 処理サイト: RCSB / 状態: 公開 |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト) / synthetic construct (人工物)
Homo sapiens (ヒト) / synthetic construct (人工物) データ登録者
データ登録者 米国, 6件
米国, 6件  引用
引用 ジャーナル: To Be Published
ジャーナル: To Be Published 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_22919.map.gz
emd_22919.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-22919-v30.xml
emd-22919-v30.xml emd-22919.xml
emd-22919.xml EMDBヘッダ
EMDBヘッダ emd_22919_fsc.xml
emd_22919_fsc.xml FSCデータファイル
FSCデータファイル emd_22919.png
emd_22919.png emd-22919.cif.gz
emd-22919.cif.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-22919
http://ftp.pdbj.org/pub/emdb/structures/EMD-22919 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22919
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22919 emd_22919_validation.pdf.gz
emd_22919_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_22919_full_validation.pdf.gz
emd_22919_full_validation.pdf.gz emd_22919_validation.xml.gz
emd_22919_validation.xml.gz emd_22919_validation.cif.gz
emd_22919_validation.cif.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22919
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22919 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22919
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22919 リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_22919.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_22919.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) 試料の構成要素
試料の構成要素 Homo sapiens (ヒト)
Homo sapiens (ヒト)
 Homo sapiens (ヒト)
Homo sapiens (ヒト)

 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー



























 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)