+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22828 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | HCMV prefusion gB in complex with fusion inhibitor WAY-174865 | |||||||||
 マップデータ マップデータ | HCMV gB in prefusion conformation | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | fusogen / prefusion / HCMV / gB / antibody / VIRAL PROTEIN / fusion inhibitor / VIRAL PROTEIN-INHIBITOR complex | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報host cell Golgi membrane / host cell endosome membrane / viral envelope / symbiont entry into host cell / virion attachment to host cell / host cell plasma membrane / virion membrane / membrane 類似検索 - 分子機能 | |||||||||
| 生物種 |  Human cytomegalovirus (strain Towne) (ヘルペスウイルス) Human cytomegalovirus (strain Towne) (ヘルペスウイルス) | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Liu Y / Heim PK | |||||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2021 ジャーナル: Sci Adv / 年: 2021タイトル: Prefusion structure of human cytomegalovirus glycoprotein B and structural basis for membrane fusion. 著者: Yuhang Liu / Kyle P Heim / Ye Che / Xiaoyuan Chi / Xiayang Qiu / Seungil Han / Philip R Dormitzer / Xinzhen Yang /  要旨: Human cytomegalovirus (HCMV) causes congenital disease with long-term morbidity. HCMV glycoprotein B (gB) transitions irreversibly from a metastable prefusion to a stable postfusion conformation to ...Human cytomegalovirus (HCMV) causes congenital disease with long-term morbidity. HCMV glycoprotein B (gB) transitions irreversibly from a metastable prefusion to a stable postfusion conformation to fuse the viral envelope with a host cell membrane during entry. We stabilized prefusion gB on the virion with a fusion inhibitor and a chemical cross-linker, extracted and purified it, and then determined its structure to 3.6-Å resolution by electron cryomicroscopy. Our results revealed the structural rearrangements that mediate membrane fusion and details of the interactions among the fusion loops, the membrane-proximal region, transmembrane domain, and bound fusion inhibitor that stabilized gB in the prefusion state. The structure rationalizes known gB antigenic sites. By analogy to successful vaccine antigen engineering approaches for other viral pathogens, the high-resolution prefusion gB structure provides a basis to develop stabilized prefusion gB HCMV vaccine antigens. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22828.map.gz emd_22828.map.gz | 5.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22828-v30.xml emd-22828-v30.xml emd-22828.xml emd-22828.xml | 16.1 KB 16.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22828_fsc.xml emd_22828_fsc.xml | 9.9 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22828.png emd_22828.png | 41.7 KB | ||
| Filedesc metadata |  emd-22828.cif.gz emd-22828.cif.gz | 6.6 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22828 http://ftp.pdbj.org/pub/emdb/structures/EMD-22828 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22828 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22828 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22828.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22828.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | HCMV gB in prefusion conformation | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.376 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
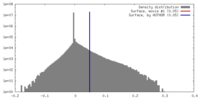
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : HCMV prefusion gB stabilized by a fusion inhibitor
| 全体 | 名称: HCMV prefusion gB stabilized by a fusion inhibitor |
|---|---|
| 要素 |
|
-超分子 #1: HCMV prefusion gB stabilized by a fusion inhibitor
| 超分子 | 名称: HCMV prefusion gB stabilized by a fusion inhibitor / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 詳細: SM5-1 FAb is recombinant expressed with 6xHis and Strep sequence at C-terminal as purification tags. The FAb was used to purify the gB protein from lab cultured HCMV virus (Towne strain)In ...詳細: SM5-1 FAb is recombinant expressed with 6xHis and Strep sequence at C-terminal as purification tags. The FAb was used to purify the gB protein from lab cultured HCMV virus (Towne strain)In the density map, the FAb is poorly resolved and not modeled |
|---|---|
| 由来(天然) | 生物種:  Human cytomegalovirus (strain Towne) (ヘルペスウイルス) Human cytomegalovirus (strain Towne) (ヘルペスウイルス)株: Towne |
| 分子量 | 理論値: 300 KDa |
-分子 #1: Envelope glycoprotein B
| 分子 | 名称: Envelope glycoprotein B / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Human cytomegalovirus (strain Towne) (ヘルペスウイルス) Human cytomegalovirus (strain Towne) (ヘルペスウイルス)株: Towne |
| 分子量 | 理論値: 102.067688 KDa |
| 配列 | 文字列: MESRIWCLVV CVNLCIVCLG AAVSSSSTRG TSATHSHHSS HTTSAAHSRS GSVSQRVTSS QTVSHGVNET IYNTTLKYGD VVGVNTTKY PYRVCSMAQG TDLIRFERNI VCTSMKPINE DLDEGIMVVY KRNIVAHTFK VRVYQKVLTF RRSYAYIHTT Y LLGSNTEY ...文字列: MESRIWCLVV CVNLCIVCLG AAVSSSSTRG TSATHSHHSS HTTSAAHSRS GSVSQRVTSS QTVSHGVNET IYNTTLKYGD VVGVNTTKY PYRVCSMAQG TDLIRFERNI VCTSMKPINE DLDEGIMVVY KRNIVAHTFK VRVYQKVLTF RRSYAYIHTT Y LLGSNTEY VAPPMWEIHH INSHSQCYSS YSRVIAGTVF VAYHRDSYEN KTMQLMPDDY SNTHSTRYVT VKDQWHSRGS TW LYRETCN LNCMVTITTA RSKYPYHFFA TSTGDVVDIS PFYNGTNRNA SYFGENADKF FIFPNYTIVS DFGRPNSALE THR LVAFLE RADSVISWDI QDEKNVTCQL TFWEASERTI RSEAEDSYHF SSAKMTATFL SKKQEVNMSD SALDCVRDEA INKL QQIFN TSYNQTYEKY GNVSVFETTG GLVVFWQGIK QKSLVELERL ANRSSLNLTH NRTKRSTDGN NATHLSNMES VHNLV YAQL QFTYDTLRGY INRALAQIAE AWCVDQRRTL EVFKELSKIN PSAILSAIYN KPIAARFMGD VLGLASCVTI NQTSVK VLR DMNVKESPGR CYSRPVVIFN FANSSYVQYG QLGEDNEILL GNHRTEECQL PSLKIFIAGN SAYEYVDYLF KRMIDLS SI STVDSMIALD IDPLENTDFR VLELYSQKEL RSSNVFDLEE IMREFNSYKQ RVKYVEDKVV DPLPPYLKGL DDLMSGLG A AGKAVGVAIG AVGGAVASVV EGVATFLKNP FGAFTIILVA IAVVIIIYLI YTRQRRLCMQ PLQNLFPYLV SADGTTVTS GNTKDTSLQA PPSYEESVYN SGRKGPGPPS SDASTAAPPY TNEQAYQMLL ALVRLDAEQR AQQNGTDSLD GQTGTQDKGQ KPNLLDRLR HRKNGYRHLK DSDEEENV UniProtKB: Envelope glycoprotein B |
-分子 #2: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 2 / コピー数: 30 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information |  ChemComp-NAG: |
-分子 #3: N-{4-[({(1S)-1-[3,5-bis(trifluoromethyl)phenyl]ethyl}carbamothioy...
| 分子 | 名称: N-{4-[({(1S)-1-[3,5-bis(trifluoromethyl)phenyl]ethyl}carbamothioyl)amino]phenyl}-1,3-thiazole-4-carboxamide タイプ: ligand / ID: 3 / コピー数: 3 / 式: WCY |
|---|---|
| 分子量 | 理論値: 518.498 Da |
| Chemical component information |  ChemComp-WCY: |
-分子 #4: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 4 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.8 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 / 詳細: PBS, 0.1 % DDM, 1 mM EDTA, 2 mg/L WAY-174865 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: GRAPHENE OXIDE / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK II / 詳細: 4 second, -2 force. |
| 詳細 | this sample was monodispersed |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-28 / 撮影したグリッド数: 3 / 実像数: 7768 / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 70.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm 最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 18000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)