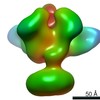
登録情報 データベース : EMDB / ID : EMD-22449タイトル Plant mitochondrial supercomplex III2+IV from Vigna radiata Plant mitochondrial supercomplex III2 IV from Vigna radiata. 複合体 : Mitochondrial Respiratory Complex III2機能・相同性 分子機能 ドメイン・相同性 構成要素
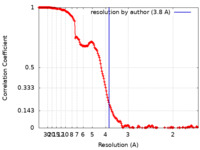
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Vigna radiata (リョクトウ)手法 / / 解像度 : 3.8 Å Maldonado M / Letts JA ジャーナル : Elife / 年 : 2021タイトル : Atomic structures of respiratory complex III, complex IV, and supercomplex III-IV from vascular plants.著者 : Maria Maldonado / Fei Guo / James A Letts / 要旨 : Mitochondrial complex III (CIII) and complex IV (CIV), which can associate into a higher-order supercomplex (SC III+IV), play key roles in respiration. However, structures of these plant complexes ... Mitochondrial complex III (CIII) and complex IV (CIV), which can associate into a higher-order supercomplex (SC III+IV), play key roles in respiration. However, structures of these plant complexes remain unknown. We present atomic models of CIII, CIV, and SC III+IV from determined by single-particle cryoEM. The structures reveal plant-specific differences in the MPP domain of CIII and define the subunit composition of CIV. Conformational heterogeneity analysis of CIII revealed long-range, coordinated movements across the complex, as well as the motion of CIII's iron-sulfur head domain. The CIV structure suggests that, in plants, proton translocation does not occur via the H channel. The supercomplex interface differs significantly from that in yeast and bacteria in its interacting subunits, angle of approach and limited interactions in the mitochondrial matrix. These structures challenge long-standing assumptions about the plant complexes and generate new mechanistic hypotheses. 履歴 登録 2020年8月12日 - ヘッダ(付随情報) 公開 2021年1月20日 - マップ公開 2021年1月20日 - 更新 2021年2月3日 - 現状 2021年2月3日 処理サイト : RCSB / 状態 : 公開
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 マップデータ
マップデータ 試料
試料 機能・相同性情報
機能・相同性情報 Vigna radiata (リョクトウ)
Vigna radiata (リョクトウ) データ登録者
データ登録者 引用
引用 ジャーナル: Elife / 年: 2021
ジャーナル: Elife / 年: 2021
 構造の表示
構造の表示 ムービービューア
ムービービューア SurfView
SurfView Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク emd_22449.map.gz
emd_22449.map.gz EMDBマップデータ形式
EMDBマップデータ形式 emd-22449-v30.xml
emd-22449-v30.xml emd-22449.xml
emd-22449.xml EMDBヘッダ
EMDBヘッダ emd_22449_fsc.xml
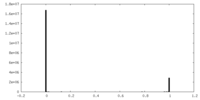
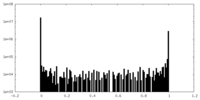
emd_22449_fsc.xml FSCデータファイル
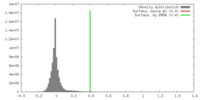
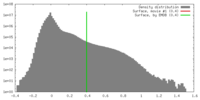
FSCデータファイル emd_22449.png
emd_22449.png emd_22449_msk_1.map
emd_22449_msk_1.map マスクマップ
マスクマップ emd_22449_half_map_1.map.gz
emd_22449_half_map_1.map.gz emd_22449_half_map_2.map.gz
emd_22449_half_map_2.map.gz http://ftp.pdbj.org/pub/emdb/structures/EMD-22449
http://ftp.pdbj.org/pub/emdb/structures/EMD-22449 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22449
ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22449 emd_22449_validation.pdf.gz
emd_22449_validation.pdf.gz EMDB検証レポート
EMDB検証レポート emd_22449_full_validation.pdf.gz
emd_22449_full_validation.pdf.gz emd_22449_validation.xml.gz
emd_22449_validation.xml.gz https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22449
https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22449 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22449
ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22449


 EMPIAR-10586 (タイトル: Cryo electron micrographs of digitonin-solubilized, amphipol-stabilized, sucrose-gradient-purified V. radiata mitochondrial membranes - mixed fraction containing CI*, CIII2 and SC III2+IV
EMPIAR-10586 (タイトル: Cryo electron micrographs of digitonin-solubilized, amphipol-stabilized, sucrose-gradient-purified V. radiata mitochondrial membranes - mixed fraction containing CI*, CIII2 and SC III2+IV リンク
リンク EMDB (EBI/PDBe) /
EMDB (EBI/PDBe) /  EMDataResource
EMDataResource マップ
マップ ダウンロード / ファイル: emd_22449.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES)
ダウンロード / ファイル: emd_22449.map.gz / 形式: CCP4 / 大きさ: 512 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) emd_22449_msk_1.map
emd_22449_msk_1.map 試料の構成要素
試料の構成要素 Vigna radiata (リョクトウ) / 組織: hypocotyl / Organelle: mitochondria
Vigna radiata (リョクトウ) / 組織: hypocotyl / Organelle: mitochondria 解析
解析 試料調製
試料調製 電子顕微鏡法
電子顕微鏡法 FIELD EMISSION GUN
FIELD EMISSION GUN
 ムービー
ムービー コントローラー
コントローラー






















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)