+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22219 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of CARD8-CARD filament | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Filament / inflammasome / signaling / UPA / FIIND / CARD / NLRP1 / IMMUNE SYSTEM | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報CARD8 inflammasome complex assembly / CARD8 inflammasome complex / NACHT domain binding / Formation of apoptosome / CARD domain binding / NLRP3 inflammasome complex / self proteolysis / negative regulation of NLRP3 inflammasome complex assembly / cysteine-type endopeptidase activator activity / negative regulation of lipopolysaccharide-mediated signaling pathway ...CARD8 inflammasome complex assembly / CARD8 inflammasome complex / NACHT domain binding / Formation of apoptosome / CARD domain binding / NLRP3 inflammasome complex / self proteolysis / negative regulation of NLRP3 inflammasome complex assembly / cysteine-type endopeptidase activator activity / negative regulation of lipopolysaccharide-mediated signaling pathway / Regulation of the apoptosome activity / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素 / negative regulation of interleukin-1 beta production / pattern recognition receptor activity / negative regulation of tumor necrosis factor-mediated signaling pathway / negative regulation of canonical NF-kappaB signal transduction / antiviral innate immune response / intrinsic apoptotic signaling pathway / activation of innate immune response / positive regulation of interleukin-1 beta production / molecular condensate scaffold activity / peptidase activity / regulation of apoptotic process / defense response to virus / inflammatory response / protein homodimerization activity / protein-containing complex / nucleoplasm / nucleus / cytoplasm / cytosol 類似検索 - 分子機能 | |||||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
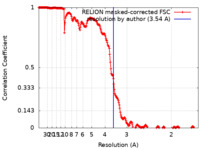
| 手法 | らせん対称体再構成法 / クライオ電子顕微鏡法 / 解像度: 3.54 Å | |||||||||
 データ登録者 データ登録者 | Hollingsworth LR / David L | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2021 ジャーナル: Nat Commun / 年: 2021タイトル: Mechanism of filament formation in UPA-promoted CARD8 and NLRP1 inflammasomes. 著者: L Robert Hollingsworth / Liron David / Yang Li / Andrew R Griswold / Jianbin Ruan / Humayun Sharif / Pietro Fontana / Elizabeth L Orth-He / Tian-Min Fu / Daniel A Bachovchin / Hao Wu /  要旨: NLRP1 and CARD8 are related cytosolic sensors that upon activation form supramolecular signalling complexes known as canonical inflammasomes, resulting in caspase-1 activation, cytokine maturation ...NLRP1 and CARD8 are related cytosolic sensors that upon activation form supramolecular signalling complexes known as canonical inflammasomes, resulting in caspase-1 activation, cytokine maturation and/or pyroptotic cell death. NLRP1 and CARD8 use their C-terminal (CT) fragments containing a caspase recruitment domain (CARD) and the UPA (conserved in UNC5, PIDD, and ankyrins) subdomain for self-oligomerization, which in turn form the platform to recruit the inflammasome adaptor ASC (apoptosis-associated speck-like protein containing a CARD) or caspase-1, respectively. Here, we report cryo-EM structures of NLRP1-CT and CARD8-CT assemblies, in which the respective CARDs form central helical filaments that are promoted by oligomerized, but flexibly linked, UPAs surrounding the filaments. Through biochemical and cellular approaches, we demonstrate that the UPA itself reduces the threshold needed for NLRP1-CT and CARD8-CT filament formation and signalling. Structural analyses provide insights on the mode of ASC recruitment by NLRP1-CT and the contrasting direct recruitment of caspase-1 by CARD8-CT. We also discover that subunits in the central NLRP1 filament dimerize with additional exterior CARDs, which roughly doubles its thickness and is unique among all known CARD filaments. Finally, we engineer and determine the structure of an ASC-caspase-1 octamer, which suggests that ASC uses opposing surfaces for NLRP1, versus caspase-1, recruitment. Together these structures capture the architecture and specificity of the active NLRP1 and CARD8 inflammasomes in addition to key heteromeric CARD-CARD interactions governing inflammasome signalling. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22219.map.gz emd_22219.map.gz | 925.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22219-v30.xml emd-22219-v30.xml emd-22219.xml emd-22219.xml | 13.1 KB 13.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_22219_fsc.xml emd_22219_fsc.xml | 22.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_22219.png emd_22219.png | 139.8 KB | ||
| Filedesc metadata |  emd-22219.cif.gz emd-22219.cif.gz | 5.7 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22219 http://ftp.pdbj.org/pub/emdb/structures/EMD-22219 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22219 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22219 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22219_validation.pdf.gz emd_22219_validation.pdf.gz | 691.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22219_full_validation.pdf.gz emd_22219_full_validation.pdf.gz | 690.7 KB | 表示 | |
| XML形式データ |  emd_22219_validation.xml.gz emd_22219_validation.xml.gz | 18.6 KB | 表示 | |
| CIF形式データ |  emd_22219_validation.cif.gz emd_22219_validation.cif.gz | 26.1 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22219 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22219 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22219 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22219 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  6xkjMC  6xkkC  7keuC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | |
| 電子顕微鏡画像生データ |  EMPIAR-10567 (タイトル: CARD8-CT filament / Data size: 1.2 TB EMPIAR-10567 (タイトル: CARD8-CT filament / Data size: 1.2 TBData #1: Unaligned multi-frame micrographs for the CARD8 CT filament [micrographs - multiframe]) |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22219.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22219.map.gz / 形式: CCP4 / 大きさ: 1000 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.8315 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
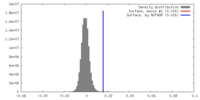
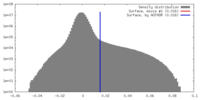
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : CARD8-CARD filament
| 全体 | 名称: CARD8-CARD filament |
|---|---|
| 要素 |
|
-超分子 #1: CARD8-CARD filament
| 超分子 | 名称: CARD8-CARD filament / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Caspase recruitment domain-containing protein 8
| 分子 | 名称: Caspase recruitment domain-containing protein 8 / タイプ: protein_or_peptide / ID: 1 / コピー数: 16 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 10.103373 KDa |
| 組換発現 | 生物種:  |
| 配列 | 文字列: AAFVKENHRQ LQARMGDLKG VLDDLQDNEV LTENEKELVE QEKTRQSKNE ALLSMVEKKG DLALDVLFRS ISERDPYLVS YLRQQNL UniProtKB: Caspase recruitment domain-containing protein 8 |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | らせん対称体再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 400 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. / 前処理 - 雰囲気: AIR | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK III |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 1208 / 平均露光時間: 1.25 sec. / 平均電子線量: 40.0 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / Cs: 2.7 mm / 最大 デフォーカス(公称値): -2.2 µm / 最小 デフォーカス(公称値): -0.8 µm / 倍率(公称値): 96000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー

















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)