+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-22042 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
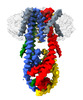
| タイトル | MCU-EMRE complex of a metazoan mitochondrial calcium uniporter | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ion channel / calcium channel / mitochondrial calcium uniporter / MCU / EMRE / mitochondria / MEMBRANE PROTEIN | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報uniporter activity / uniplex complex / mitochondrial calcium ion homeostasis / calcium import into the mitochondrion / calcium channel activity / metal ion binding 類似検索 - 分子機能 | |||||||||
| 生物種 |  | |||||||||
| 手法 | 単粒子再構成法 / クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Long SB / Wang C | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: J Mol Biol / 年: 2020 ジャーナル: J Mol Biol / 年: 2020タイトル: Structure and Reconstitution of an MCU-EMRE Mitochondrial Ca Uniporter Complex. 著者: Chongyuan Wang / Rozbeh Baradaran / Stephen Barstow Long /  要旨: The proteins MCU and EMRE form the minimal functional unit of the mitochondrial calcium uniporter complex in metazoans, a highly selective and tightly controlled Ca channel of the inner mitochondrial ...The proteins MCU and EMRE form the minimal functional unit of the mitochondrial calcium uniporter complex in metazoans, a highly selective and tightly controlled Ca channel of the inner mitochondrial membrane that regulates cellular metabolism. Here we present functional reconstitution of an MCU-EMRE complex from the red flour beetle, Tribolium castaneum, and a cryo-EM structure of the complex at 3.5 Å resolution. Using a novel assay, we demonstrate robust Ca uptake into proteoliposomes containing the purified complex. Uptake is dependent on EMRE and also on the mitochondrial lipid cardiolipin. The structure reveals a tetrameric channel with a single ion pore. EMRE is located at the periphery of the transmembrane domain and associates primarily with the first transmembrane helix of MCU. Coiled-coil and juxtamembrane domains within the matrix portion of the complex adopt markedly different conformations than in a structure of a human MCU-EMRE complex, suggesting that the structures represent different conformations of these functionally similar metazoan channels. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_22042.map.gz emd_22042.map.gz | 1.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-22042-v30.xml emd-22042-v30.xml emd-22042.xml emd-22042.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_22042.png emd_22042.png | 113 KB | ||
| Filedesc metadata |  emd-22042.cif.gz emd-22042.cif.gz | 6.1 KB | ||
| その他 |  emd_22042_additional.map.gz emd_22042_additional.map.gz | 95.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-22042 http://ftp.pdbj.org/pub/emdb/structures/EMD-22042 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22042 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-22042 | HTTPS FTP |
-検証レポート
| 文書・要旨 |  emd_22042_validation.pdf.gz emd_22042_validation.pdf.gz | 325.1 KB | 表示 |  EMDB検証レポート EMDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  emd_22042_full_validation.pdf.gz emd_22042_full_validation.pdf.gz | 324.6 KB | 表示 | |
| XML形式データ |  emd_22042_validation.xml.gz emd_22042_validation.xml.gz | 6.4 KB | 表示 | |
| CIF形式データ |  emd_22042_validation.cif.gz emd_22042_validation.cif.gz | 7.3 KB | 表示 | |
| アーカイブディレクトリ |  https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22042 https://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22042 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22042 ftp://ftp.pdbj.org/pub/emdb/validation_reports/EMD-22042 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_22042.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_22042.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.088 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
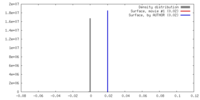
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
-追加マップ: unsharpened map
| ファイル | emd_22042_additional.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
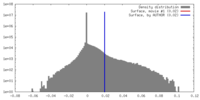
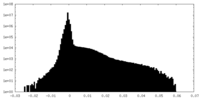
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Mitochondrial Calcium Uniporter (MCU) in complex with EMRE. Embed...
| 全体 | 名称: Mitochondrial Calcium Uniporter (MCU) in complex with EMRE. Embedded in lipid nanodiscs containing cardiolipin. |
|---|---|
| 要素 |
|
-超分子 #1: Mitochondrial Calcium Uniporter (MCU) in complex with EMRE. Embed...
| 超分子 | 名称: Mitochondrial Calcium Uniporter (MCU) in complex with EMRE. Embedded in lipid nanodiscs containing cardiolipin. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:  |
-分子 #1: Calcium uniporter protein,Protein EMRE homolog, mitochondrial-lik...
| 分子 | 名称: Calcium uniporter protein,Protein EMRE homolog, mitochondrial-like Protein fusion タイプ: protein_or_peptide / ID: 1 / コピー数: 8 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  |
| 分子量 | 理論値: 27.530723 KDa |
| 組換発現 | 生物種:  Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: GPTAAALERL TTEEVQGLSD VKTLVNQLYE ALNVREHQLQ KEVELTTQLE TLQQELLPLE EKKLELEQVA NRRSNWMAWA GLGLMSVQF GILARLTWWE YSWDIMEPVT YFVTYGTAMA AYAYFVLTRE EYILNDVRDR QQLLLLHKKA KKTGFDVNQY N VLKDQIAK ...文字列: GPTAAALERL TTEEVQGLSD VKTLVNQLYE ALNVREHQLQ KEVELTTQLE TLQQELLPLE EKKLELEQVA NRRSNWMAWA GLGLMSVQF GILARLTWWE YSWDIMEPVT YFVTYGTAMA AYAYFVLTRE EYILNDVRDR QQLLLLHKKA KKTGFDVNQY N VLKDQIAK LELDLKRLRD PLKLRLPPKA AASGSGSGEN LYFQGSGGLL PEPHRTSFGI IRLILTVVPG LLIGAAISKN IA NFL UniProtKB: Calcium uniporter protein, mitochondrial, Essential MCU regulator, mitochondrial |
-分子 #2: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 2 / コピー数: 1 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-実験情報
-構造解析
| 手法 | クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY ARRAY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 10 sec. / 前処理 - 雰囲気: OTHER | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK IV / 詳細: 2 second blot, blot force of 0. | ||||||||||||
| 詳細 | Monodisperse sample |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - 画像ごとのフレーム数: 1-40 / 撮影したグリッド数: 3 / 実像数: 8143 / 平均露光時間: 0.25 sec. / 平均電子線量: 1.9 e/Å2 |
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD / 最大 デフォーカス(公称値): -3.0 µm / 最小 デフォーカス(公称値): -1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
+ 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: AB INITIO MODEL / 温度因子: 127 / 当てはまり具合の基準: Correlation coefficient |
|---|---|
| 得られたモデル |  PDB-6x4s: |
 ムービー
ムービー コントローラー
コントローラー











 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)